Research progress of UbiA membrane-bound aromatic prenyltransferases in plants
-
摘要:
UbiA膜结合型芳香族异戊烯基转移酶(Prenyltransferases,PT)可催化异戊烯基单元转移到芳香族母核上形成C-C(或C-O)键,在植物中参与合成重要的代谢产物,如泛醌、质体醌、叶绿素、生育酚等。植物中多种具有异戊烯基的芳香族次生代谢物也是该类酶作用的产物。异戊烯基的引入增加了天然产物结构多样性和生物活性。本文介绍了植物中UbiA家族的基本类型,归纳了57个已鉴定功能的与次生代谢物(类黄酮、香豆素、二苯乙烯等)合成相关的UbiA PTs底物选择性、催化特点及其与初生代谢相关PTs的系统发育关系,并对异戊烯基转移酶基因的挖掘策略,以及利用微生物代谢工程定向合成活性异戊烯基化合物的应用前景进行了展望。
Abstract:UbiA membrane-bound aromatic prenyltransferases (UbiA PTs) catalyze the transfer of prenyl moieties to aromatic acceptor molecules to form C-C or C-O bonds, and participate in the biosynthesis of important plant chemicals, including ubiquinone, plastoquinone, chlorophyll, and tocopherol. A variety of aromatic secondary metabolites with prenyl groups in plants are also products of this class of enzyme. The introduction of prenyl groups increases the structural diversity and biological activity of natural products. In this paper, we introduce the basic types of UbiA families in plants, summarize the substrate selectivity and catalytic characteristics of 57 UbiA PTs related to biosynthesis of secondary metabolites (flavonoids, coumarins, stilbenes), and discuss their phylogenetic relationship with primary metabolism-related PTs. We also discuss the exploration strategies of prenyltransferase genes and the application prospects of targeted synthesis of active prenylated compounds by microbial metabolic engineering.
-
Keywords:
- Prenyltransferases /
- Primary metabolism /
- Secondary metabolism /
- Biosynthesis
-
青藏高原是生物多样性热点地区。高寒草甸是青藏高原典型的草地类型,生态脆弱并且对环境变化敏感[1, 2]。气候和人类干扰引起了高寒草甸“斑块状”退化以及植物多样性降低,而不同植物功能群所受影响往往并不一致[3, 4]。研究表明,高寒草甸退化整体表现为由禾状草优势向豆科和杂类草优势转变[5],禾状草中莎草科和禾本科之间土壤资源竞争激烈,而豆科和杂类草生态位重叠较多[6]。
相比植物物种,以植物功能群为单位研究植物多样性对生态系统过程和功能的影响具有一定的优势。不同植物功能群基于生理、形态、生活史或对环境的响应进行区分,差异较为明显[7]。例如,禾状草植株高大、具有发达的根系和分蘖能力,因此具有较强的光照、土壤养分资源的竞争能力和定殖能力[8];而大多数杂类草比禾状草根浅[9]。豆科植物与根瘤菌共生,其氮含量显著高于禾状草和杂类草,具有较高的氮元素吸收利用能力[10]。与直接采样调查[11, 12]或人工建群[13, 14]的研究方法相比,植物功能群移除实验更易人为控制,植物群落组成更接近自然环境。因此,植物功能群移除实验有助于了解高寒草甸退化过程中植物特定功能群损失对土壤生态系统功能的影响。
土壤真菌作为分解者在土壤养分循环和物质流动过程中有重要作用。植物-土壤-真菌群落相互作用与反馈是维持高寒草甸生态系统功能与服务的重要纽带[15]。地上植被可以通过凋落物或根系分泌物形式向土壤输入有机质,改变土壤养分条件,从而影响土壤真菌群落的组成结构与功能[16, 17]。因此,探究植物功能群移除对土壤真菌群落的影响对掌握高寒草甸植物群落对土壤生态系统生态功能的贡献具有重要意义。
过去草地生态系统中植物功能群对土壤真菌群落的影响多在温带地区开展研究[18-21],青藏高原高寒草甸植物对气候变化敏感而研究较少。有研究表明植物特定功能群对土壤微生物[22, 23]或真菌群落[24]有影响,却未进一步分析其对真菌群落结构、各组成类群和多样性的具体影响。部分研究局限于植物功能群对特定的真菌类群的影响,如菌根真菌[25, 26]。青藏高原土壤真菌群落对植物特定功能群损失的响应缺乏明确和系统性了解。以往的移除实验多采用移除单一功能群的方法,以观测此植物功能群在生态系统中的贡献,本研究尝试保留单一植物功能群与不进行移除处理的对照、移除所有植物功能群的处理相比较,以明确植物特定功能群对土壤真菌群落结构和多样性的影响。
因此,本研究将青藏高原高寒草甸的植被划分为禾状草、豆科和杂类草3个不同的植物功能群,采用高通量测序技术对不同植物功能群移除处理后的土壤真菌群落结构和组成进行测定,探究植物特定功能群损失对土壤真菌群落和调控土壤真菌群落特征变化的重要环境因子的影响,以期明确高寒草甸植物特定功能群物种损失对土壤真菌群落的影响,为土壤生态系统功能的维持机制提供科学依据。
1. 材料与方法
1.1 研究区概况
研究地点位于青藏高原东部甘肃省甘南草原生态系统国家野外科学观测研究站(33°58′N,101°53′E),海拔3 550 m。该地区年平均气温1.8 ℃,年均降水量约593 mm[27]。每年日照约为2 580 h,一年中霜期超过270 d。土壤类型为亚高山草甸土,属湿润寒冷的高原气候[28]。高寒草甸样地中线叶嵩草(Carex capillifolia (Decne.) S. R. Zhang)占据优势。将样地植物划分为以下3个功能群:禾状草(包含禾本科和莎草科)、豆科和杂类草。禾状草以垂穗披碱草(Elymus nutans Griseb)和异针茅(Stipa aliena Keng)为主;杂类草以小花草玉梅(Anemone rivularis Buch.-Ham.)、钝苞雪莲(Saussurea nigrescens Maxim.)、重冠紫菀(Aster diplostephioides (DC.) CB Clarke)为主;豆科植物以披针叶黄华(Thermopsis lanceolata R. Br.)、青海苜蓿(Medicago archiducis-nicolai Sirj.)为主[27]。
实验样地建立于2018年,采用拉丁方设计。实验共设5个不同的植物功能群移除处理:对照,不移除任何植物功能群的处理(CK)、移除豆科和杂类草而保留禾状草(Graminoids)、移除禾状草和杂类草而保留豆科(Legumes)、移除禾状草和豆科而保留杂类草(Forbs)以及移除所有植物功能群(None)。每个处理5个重复,共设置25个1.5 m × 1.5 m样方(图1)。样方之间间隔1 m作为缓冲带。由于样地所在位置有一定的坡度,因此将同一高度上5个处理作为一个区组,视作一个重复。2018年8月开始进行移除处理后,其后每年生长季都对样地进行多次移除处理,以达到较好的移除效果。每次移除处理都按照不同功能群类别将目标植物齐地剪除并移出。
1.2 样品采集
2021年9月采集土壤样品,每个样方划分为9个小样方,选取其中一个小样方的4个角和中心点钻取0~10 cm土层,混合为1个样品。土壤样品带回实验室后过 2 mm筛收集土壤中植物根系并去除石子,分为3部分。取其中10 g鲜土烘干用于土壤含水率的测定;40 g鲜土用于铵态氮、硝态氮、可溶性有机碳的测定和土壤真菌测序分析;剩余土壤在室温下风干,用于土壤总碳、总氮和有机碳的测定。
1.3 土壤理化性质和根碳、氮含量的测定
采用烘干法(105 ℃,24 h)测定土壤含水率(Soil water content,SWC)。采用水杨酸-次氯酸盐分光光度法测定铵态氮(Ammonium nitrogen,
NH+4 -N),硫酸肼还原法测定硝态氮(Nitrate nitrogen,NO−3 -N)。可溶性有机碳(Dissolved organic carbon,DOC)采用0.5 mol/L 的K2SO4浸提后,180 r/min离心1 h,上清液用0.45 μm的微孔滤膜过滤,得到的滤液用TOC分析仪测定。植物根系先80 ℃烘干,后45 ℃烘干至恒重,用于根系生物量的测定以及根总碳、根总氮的测定。土壤总碳(Total carbon,TC)、总氮(Total nitrogen,TN)、土壤有机碳(Soil organic carbon,SOC)、根总碳(Root total carbon,Root TC)、根总氮(Root total nitrogen,Root TN)含量均用全自动有机元素分析仪(Vario macro cube,Elementar)测定。1.4 土壤真菌总DNA提取、PCR扩增、测序、OTUs和功能类群的划分
使用CTAB/SDS方法从土壤样品中提取真菌总基因组 DNA。在1%琼脂糖凝胶上检测 DNA 浓度和纯度。所有PCR反应均使用Phusion® High-Fidelity PCR Master Mix(New England Biolabs)预混液进行,对目标片段进行PCR扩增。采用真菌通用引物ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')/ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')对真菌ITS1区进行扩增。PCR产物用2% 琼脂糖凝胶电泳检测,胶回收试剂盒(Qiagen,Germany)回收。使用 TruSeq® DNA PCR-Free Sample Preparation Kit(Illumina,USA)生成测序文库,基于 Illumina NovaSeq 平台进行测序。土壤真菌测序由北京诺禾致源科技股份有限公司完成。对测序序列Reads 拼接过滤后得到的有效序列用Usearch软件在97%相似度水平下聚类生成OTUs(Operational taxonomic units)。以UNITE为参考数据库对特征序列进行分类学注释后进行后续分析。利用 FUNGuild 数据库对土壤真菌群落进行功能注释,根据宿主种类和营养方式大体上分为13个功能类群。
1.5 数据处理及分析
采用R v4.1.3软件进行数据分析及可视化。真菌群落Alpha多样性指数等基于OTUs结果计算。不同处理间真菌群落结构的差异采用基于Bray-Curtis 距离的非度量多维尺度分析(NMDS)和相似性分析(ANOSIM)进行分析。对不同植物功能群移除处理下根系性质、土壤理化性质、土壤真菌群落结构组成、功能类群组成的差异采用线性混合模型进行分析。线性混合模型中以试验处理为固定因素,样方所在的区组为随机因素。土壤真菌群落与环境因子(根系性质和土壤理化性质)的相关性通过Spearman相关系数和Mantel test进行分析。Mantel test分析中真菌群落结构基于OTUs水平衡量,真菌群落功能基于FUNGuild注释结果的相对丰度衡量。土壤真菌多样性指数与环境因子的相关性采用Spearman相关分析。图表中数据为平均值 ± 标准误。
2. 结果与分析
2.1 植物根系性质和土壤理化性质
研究结果显示,样地植物根总碳的平均含量分布在271.2~332.0 g/kg。根总氮的平均含量分布在8.1~9.5 g/kg。根系碳氮比的平均值分布在30.1~34.6。平均根系生物量分布在0.5~0.9 kg/m2。不同植物功能群移除处理的根系性质(根总碳、根总氮、根系碳氮比、根系生物量)差异不显著(表1)。
表 1 不同植物功能群移除下根系性质Table 1. Root properties under removal of different plant functional groups指标Index CK Forbs Graminoids Legumes None 根总碳 / g/kg 332.0 ± 6.0a 275.6 ± 7.8a 279.1 ± 31.1a 271.2 ± 29.4a 300.3 ± 27.0a 根总氮 / g/kg 8.91 ± 0.85a 8.63 ± 0.74a 8.14 ± 0.25a 9.04 ± 0.65a 9.43 ± 0.49a 根系碳氮比 34.04 ± 5.44a 32.85 ± 2.76a 34.59 ± 4.35a 30.13 ± 2.81a 31.80 ± 2.13a 根系生物量 / kg/m2 0.68 ± 0.23a 0.85 ± 0.20a 0.77 ± 0.12a 0.72 ± 0.16a 0.54 ± 0.15a 注:同行不同小写字母表示在0.05水平上差异显著。CK:不移除;Forbs:移除禾状草和豆科而保留杂类草;Graminoids:移除豆科和杂类草而保留禾状草;Legumes:移除禾状草和杂类草而保留豆科;None:移除所有植物功能群。下同。 Notes: Different lowercase letters in the same row indicate significant differences at the 0.05 level. CK: No removals; Forbs: Removal of graminoids and legumes; Graminoids: Removal of legumes and forbs; Legumes: Removal of graminoids and forbs; None: Removal of all. Same below. 不同植物功能群移除处理下,SWC、TC、TN、C/N、SOC、DOC和
NH+4 -N、NO−3 -N含量均无显著差异(表2)。表 2 不同植物功能群移除下土壤理化性质Table 2. Soil properties under removal of different plant functional groups指标Index CK Forbs Graminoids Legumes None 土壤含水率 / % 54.72 ± 2.18a 55.10 ± 5.30a 54.91 ± 3.92a 50.80 ± 5.10a 51.86 ± 4.87a 总碳 / g/kg 65.05 ± 3.50a 67.44 ± 5.89a 60.38 ± 4.65a 64.97 ± 6.68a 61.19 ± 5.66a 总氮 / g/kg 5.85 ± 0.25a 6.08 ± 0.45a 5.53 ± 0.36a 5.82 ± 0.48a 5.56 ± 0.43a 碳氮比 11.09 ± 0.14a 11.06 ± 0.16a 10.89 ± 0.19a 11.09 ± 0.24a 10.96 ± 0.18a 土壤有机碳 / g/kg 67.72 ± 3.34a 67.15 ± 5.43a 60.60 ± 4.97a 63.01 ± 5.60a 62.43 ± 5.92a 可溶性有机碳 / mg/kg 309.4 ± 19.8a 309.1 ± 25.4a 263.9 ± 15.0a 286.2 ± 14.5a 269.3 ± 11.3a 铵态氮 / mg/kg 8.79 ± 1.16a 9.33 ± 0.45a 9.80 ± 1.88a 8.45 ± 1.58a 9.55 ± 1.23a 硝态氮 / mg/kg 6.10 ± 0.94a 5.22 ± 0.68a 4.76 ± 0.32a 4.14 ± 0.60a 6.62 ± 0.99a 2.2 土壤真菌群落结构组成、多样性和功能类群
从样地土壤样品中共获得814个OTUs(图2),隶属于12个门、27个纲、62个目、99个科和 177个属。其中,735个OTUs 为5种处理的样地所共有,对照处理中特有40个 OTUs(图2:A)。在门级水平上,除未分类的真菌类群,所有处理中都以子囊菌门、被孢霉门、担子菌门为优势门(相对丰度 > 10%),其相对丰度分别为51.0%~57.0%、19.5%~23.2%、16.4%~24.8%(图2:B)。基于Bray-Curtis距离的NMDS分析结果表明(图2:C),在门级水平上,不同植物功能群移除处理的土壤真菌群落结构相似。模型应力函数(Stress)值小于0.05,说明结果较为可靠。采用ANOSIM分析进一步验证,结果一致(P = 0.997)。
![]() 图 2 不同植物功能群移除下土壤真菌群落结构组成A:不同处理土壤真菌OTUs的Venn图;B:不同样品的门水平土壤真菌群落结构组成;C:不同处理下门水平的土壤真菌群落NMDS图。CK:不移除;Forbs:移除禾状草和豆科而保留杂类草;Graminoids:移除豆科和杂类草而保留禾状草;Legumes:移除禾状草和杂类草而保留豆科;None:移除所有植物功能群。Figure 2. Soil fungal community composition under removal of different plant functional groupsA: Venn diagram of soil fungal OTUs in different treatments; B: Phylum-level soil fungal community composition in different samples; C: NMDS diagram of phylum-level soil fungal community in different treatments. CK: No removals; Forbs: Removal of graminoids and legumes; Graminoids: Removal of legumes and forbs; Legumes: Removal of graminoids and forbs; None: Removal of all.
图 2 不同植物功能群移除下土壤真菌群落结构组成A:不同处理土壤真菌OTUs的Venn图;B:不同样品的门水平土壤真菌群落结构组成;C:不同处理下门水平的土壤真菌群落NMDS图。CK:不移除;Forbs:移除禾状草和豆科而保留杂类草;Graminoids:移除豆科和杂类草而保留禾状草;Legumes:移除禾状草和杂类草而保留豆科;None:移除所有植物功能群。Figure 2. Soil fungal community composition under removal of different plant functional groupsA: Venn diagram of soil fungal OTUs in different treatments; B: Phylum-level soil fungal community composition in different samples; C: NMDS diagram of phylum-level soil fungal community in different treatments. CK: No removals; Forbs: Removal of graminoids and legumes; Graminoids: Removal of legumes and forbs; Legumes: Removal of graminoids and forbs; None: Removal of all.土壤真菌群落以子囊菌门、被孢霉门、担子菌门为优势门。球囊菌门的相对丰度在保留杂类草、保留豆科的处理中显著高于对照。捕虫霉门的相对丰度在保留豆科处理中显著高于其他处理(表3)。
表 3 不同植物功能群移除下门水平土壤真菌相对丰度 / %Table 3. Relative abundance of soil fungi at the phylum level under removal of different plant functional groups / %门Phylum CK Forbs Graminoids Legumes None 子囊菌门 55.49 ± 5.04a 51.01 ± 4.37a 55.78 ± 6.89a 55.97 ± 3.79a 56.97 ± 4.11a 被孢霉门 22.98 ± 2.07a 21.58 ± 1.88a 19.53 ± 2.27a 20.97 ± 1.83a 23.17 ± 3.61a 担子菌门 18.67 ± 5.55a 24.84 ± 4.32a 20.27 ± 6.52a 18.51 ± 4.59a 16.38 ± 4.66a 壶菌门 0.39 ± 0.06a 0.24 ± 0.06a 1.54 ± 0.52a 1.35 ± 0.69a 0.78 ± 0.26a 根肿黑粉菌门 0.53 ± 0.28a 0.65 ± 0.23a 0.83 ± 0.40a 1.27 ± 0.37a 0.43 ± 0.07a 罗兹菌门 0.16 ± 0.06a 0.18 ± 0.01a 0.28 ± 0.14a 0.30 ± 0.04a 0.27 ± 0.06a 球囊菌门 0.05 ± 0.02b 0.19 ± 0.04a 0.17 ± 0.02ab 0.18 ± 0.05a 0.11 ± 0.02ab 油壶菌门 0.15 ± 0.05a 0.08 ± 0.02a 0.08 ± 0.02a 0.06 ± 0.01a 0.12 ± 0.02a 毛霉门 0.06 ± 0.01a 0.03 ± 0.01a 0.03 ± 0.01a 0.10 ± 0.04a 0.02 ± 0.00a 捕虫霉门 0.01 ± 0.00b 0.01 ± 0.00b 0.01 ± 0.00b 0.03 ± 0.01a 0.01 ± 0.00b 土壤真菌多样性分析结果表明,植物功能群移除对土壤真菌OTUs、香农指数、均匀度指数、辛普森指数、Chao1指数、Ace指数无显著影响(表4)。
表 4 不同植物功能群移除下土壤真菌多样性Table 4. Soil fungal diversity under removal of different plant functional groups指数Index CK Forbs Graminoids Legumes None OTUs 585.2 ± 47.2a 633 ± 39.7a 664.8 ± 7.2a 660 ± 11.7a 671.6 ± 6.1a H 4.19 ± 0.20a 4.13 ± 0.11a 4.10 ± 0.12a 4.18 ± 0.10a 4.26 ± 0.07a Pielou 0.66 ± 0.03a 0.64 ± 0.01a 0.63 ± 0.02a 0.64 ± 0.01a 0.66 ± 0.01a D 0.96 ± 0.01a 0.95 ± 0.01a 0.94 ± 0.01a 0.95 ± 0.01a 0.95 ± 0.01a Chao1 617.57 ± 45.20a 672.76 ± 34.79a 699.08 ± 9.69a 698.73 ± 8.42a 699.74 ± 3.97a Ace 613.88 ± 44.54a 661.72 ± 37.37a 697.51 ± 8.34a 693.77 ± 8.52a 696.92 ± 4.74a 注:OTUs,操作分类单位;Ace,样本中物种组成的丰富度和均匀度;Chao1,描述样品中所含OTUs数目的指数;D,辛普森指数;Pielou,均匀度指数; H,香农指数。 Notes: OTUs, operational taxonomic units; Ace index, species composition richness and evenness in a sample; Chao1 index, number of OTUs in a sample; D, Simpson index; Pielou, Pielou’s evenness index; H, Shannon-Wiener index. 由表5可知,所有处理中,土壤腐生营养型真菌相对丰度在土壤真菌群落中占比最高,约为36%~44%。丛枝菌根真菌相对丰度在保留禾状草、保留豆科处理中显著高于对照,保留杂类草、保留禾状草、保留豆科较对照处理分别增加210%、220%、230%。地衣寄生菌/腐生菌的相对丰度在保留杂类草处理中显著高于移除所有植物功能群处理,移除所有植物功能群处理较保留杂类草处理降低82%。
表 5 不同植物功能群移除下土壤真菌功能类群相对丰度 / %Table 5. Relative abundance of soil fungal functional groups under removal of different plant functional groups / %功能类群 Functional group CK Forbs Graminoids Legumes None 动物寄生真菌/病原菌 6.75 ± 3.09a 7.97 ± 3.28a 3.19 ± 0.46a 3.48 ± 0.60a 5.73 ± 2.23a 丛枝菌根真菌 0.10 ± 0.03b 0.31 ± 0.07ab 0.32 ± 0.03a 0.33 ± 0.09a 0.22 ± 0.04ab 粪腐生真菌 7.25 ± 1.12a 3.11 ± 1.38a 4.19 ± 1.38a 5.32 ± 4.02a 4.26 ± 0.61a 外生菌根真菌 6.75 ± 5.17a 0.10 ± 0.03a 0.12 ± 0.05a 0.13 ± 0.03a 0.23 ± 0.13a 内生真菌 0.36 ± 0.06a 0.49 ± 0.21a 0.42 ± 0.07a 0.39 ± 0.08a 0.78 ± 0.11a 真菌寄生菌 1.61 ± 0.62a 0.44 ± 0.05a 0.99 ± 0.25a 0.92 ± 0.39a 1.15 ± 0.45a 叶腐生菌 5.15 ± 0.67a 3.44 ± 0.42a 9.50 ± 3.62a 5.72 ± 1.83a 7.00 ± 1.15a 地衣寄生菌/腐生菌 1.15 ± 0.40ab 1.53 ± 0.49a 0.47 ± 0.10ab 0.81 ± 0.24ab 0.28 ± 0.06b 兰花菌根真菌 0.08 ± 0.04a 0.09 ± 0.05a 0.22 ± 0.07a 0.17 ± 0.09a 0.10 ± 0.06a 植物病原菌/腐生菌 8.15 ± 2.10a 5.24 ± 0.48a 5.87 ± 0.74a 9.37 ± 1.65a 8.76 ± 1.80a 土壤腐生菌 35.64 ± 2.13a 36.85 ± 4.02a 44.49 ± 5.56a 41.82 ± 5.81a 37.26 ± 4.39a 木质腐生菌 2.34 ± 0.23a 2.30 ± 0.27a 3.33 ± 0.80a 3.53 ± 0.69a 3.81 ± 0.69a 未定义腐生菌 24.67 ± 4.47a 38.14 ± 5.48a 26.90 ± 8.90a 28.01 ± 5.38a 30.42 ± 6.67a 2.3 环境因子对土壤真菌群落影响
Mantel test检验结果表明(图3),环境因子与土壤真菌群落结构和功能变化的相关性不显著。
![]() 图 3 环境因子对土壤真菌群落结构和功能的影响(Mantel test)SWC:土壤含水率;TC:总碳;TN:总氮;C:N:碳氮比;SOC:土壤有机碳;DOC:可溶性有机碳;NH+4-N:铵态氮;NO−3-N:硝态氮;Root TC:根总碳;Root TN:根总氮;Root C:N:根系碳氮比;Root biomass:根系生物量。下同。Figure 3. Effects of environmental factors on soil fungal community composition and functionSWC: Soil water content; TC: Total carbon; TN: Total nitrogen; C:N: Carbon-to-nitrogen ratio; SOC: Soil organic carbon; DOC: Dissolved organic carbon; NH+4-N: Ammonium nitrogen; NO−3-N: Nitrate nitrogen. Root TC: Root total carbon; Root TN: Root total nitrogen; Root C:N: Root carbon-to-nitrogen ratio. Same below.
图 3 环境因子对土壤真菌群落结构和功能的影响(Mantel test)SWC:土壤含水率;TC:总碳;TN:总氮;C:N:碳氮比;SOC:土壤有机碳;DOC:可溶性有机碳;NH+4-N:铵态氮;NO−3-N:硝态氮;Root TC:根总碳;Root TN:根总氮;Root C:N:根系碳氮比;Root biomass:根系生物量。下同。Figure 3. Effects of environmental factors on soil fungal community composition and functionSWC: Soil water content; TC: Total carbon; TN: Total nitrogen; C:N: Carbon-to-nitrogen ratio; SOC: Soil organic carbon; DOC: Dissolved organic carbon; NH+4-N: Ammonium nitrogen; NO−3-N: Nitrate nitrogen. Root TC: Root total carbon; Root TN: Root total nitrogen; Root C:N: Root carbon-to-nitrogen ratio. Same below.土壤真菌多样性指数与环境因子的相关分析结果表明(图4),土壤真菌群落OTUs、Chao1、Ace指数均与根系生物量呈极显著负相关(P < 0.01)。此3个指数均与根系碳氮比、土壤可溶性有机碳含量呈显著负相关(P < 0.05)。
![]() 图 4 土壤真菌多样性与环境因子相关性分析OTUs:操作分类单位;Shannon:香农指数;Pielou:均匀度指数; D:辛普森指数;Chao1:描述样品中所含OTUs数目的指数;Ace:样本中物种组成的丰富度和均匀度。Figure 4. Correlation of soil fungal diversity and environmental factorsOTUs: Operational taxonomic units; Shannon: Shannon-Wiener index; Pielou: Pielou’s evenness index; D: Simpson index; Chao1: An index describing the number of OTUs contained in a sample; Ace: An index describing the richness and evenness of species composition in a sample.
图 4 土壤真菌多样性与环境因子相关性分析OTUs:操作分类单位;Shannon:香农指数;Pielou:均匀度指数; D:辛普森指数;Chao1:描述样品中所含OTUs数目的指数;Ace:样本中物种组成的丰富度和均匀度。Figure 4. Correlation of soil fungal diversity and environmental factorsOTUs: Operational taxonomic units; Shannon: Shannon-Wiener index; Pielou: Pielou’s evenness index; D: Simpson index; Chao1: An index describing the number of OTUs contained in a sample; Ace: An index describing the richness and evenness of species composition in a sample.3. 讨论
3.1 植物功能群移除对植物根系和土壤性质的影响
通过连续3年对不同植物功能群移除,发现不同处理间的根系性质差异不显著。在高寒草甸的研究表明,豆科比禾状草、杂类草功能群的氮含量高、C:N低;禾状草具有低氮含量和高C:N的特征[29]。本研究中根系碳氮变化不显著,可能是因为草地植物根系完全分解周期以年为单位[30],高寒草甸低温环境抑制根凋落物的分解导致样地中残存较多其他功能群的根。也可能因为碳氮是植物维持代谢所需的基础元素,其内稳性较高,变异小[31]。姜林等[3]研究表明单一植物功能群移除处理对根系生物量、根系碳氮比的影响不明显。本研究进一步发现,根系生物量和根系碳氮比在同时移除多个植物功能群处理下变化仍不明显。但两项研究均表明,相比其他处理,根系生物量在移除所有植物功能群处理中最低。这说明根系生物量对高寒草甸不同植物功能群损失的响应在短期内较为稳定,但连续高强度的植物地上部分损失会造成根系生物量的降低。
本研究中土壤理化性质在各处理中差异不显著,可能主要有以下两点原因。一是,植物功能群移除对SOC的影响可能存在一个阈值,当影响低于这一阈值时SOC变化不明显。二是,植物功能群移除可能存在滞后效应。Li等[32]在青藏高原4年的移除实验结果表明,多数土壤理化性质变化不显著,其中SOC和TN含量因低温环境抑制了微生物和酶的活性而无明显变化。加拿大北部草原为期5年的研究发现,不同植物功能群移除下SWC、SOC、TN、
NH+4 -N和其他土壤理化性质变化不显著[33]。Chen等[22]在高寒草甸9年的研究表明,移除禾状草可降低TN、NH+4 -N和NO−3 -N的含量,移除豆科可降低DOC的含量,移除杂类草可降低SOC、TN和NO−3 -N的含量。3.2 植物功能群移除对土壤真菌群落结构与功能的影响
本研究中土壤真菌群落以子囊菌门、被孢霉门、担子菌门为优势门,与前人研究结果一致[15]。这可能是由于高寒草甸土壤中有机质和木质素含量较高[34, 35],子囊菌门中包含许多腐生真菌,能促进有机质的矿化[36],担子菌门包含的真菌能促进木质素分解[37]。
球囊菌门的相对丰度在未做植物移除的对照处理低于其他处理。可能是由于移除处理中植物功能群种类的降低使得植物对地上资源竞争降低,植物营养物质更多分配给地下部分而非地上部分,导致含有多种共生型真菌的球囊菌门相对丰度升高[38]。球囊菌门中含有约300种丛枝菌根真菌,是植物根系重要的共生菌[39],其相对丰度受根系质量、生物量调节。相比其他3种进行了植物移除的处理,球囊菌门真菌的相对丰度在移除所有植物功能群的处理中较低,可能与根系生物量的降低有关。前人的研究表明,球囊菌门受植物功能群移除影响显著,与本研究一致[14]。这可能说明青藏高原土壤真菌群落中球囊菌门真菌对植物群落变化敏感。
与移除所有植物功能群处理相比,捕虫霉门的相对丰度在保留豆科处理中显著增加,在保留杂类草和保留禾状草两种处理中无显著变化。说明豆科植物可能有益于捕虫霉门真菌的增加,禾状草与杂类草对其影响不显著;而对照处理中捕虫霉门的相对丰度相较移除所有植物功能群的处理无显著增加,说明禾状草或杂类草可能抑制豆科对捕虫霉门的正面影响。捕虫霉门中多数为微型无脊椎动物(如线虫等)的寄生菌[40]。植物是否通过影响食物来源如植食性线虫等对捕虫霉门真菌相对丰度产生影响还有待探究。
本研究中,土壤腐生营养型真菌相对丰度在各处理中占比均最高,与Yang等[41]的研究结果一致。保留杂类草、保留禾状草和保留豆科处理中丛枝菌根真菌相对丰度较对照处理高,可能是因为植物功能群移除后,植物根系通过增强与丛枝菌根真菌的共生来吸收土壤养分和水分进行植物的补偿生长[42]。地衣寄生菌/腐生菌的相对丰度在保留杂类草处理中显著高于移除所有植物功能群处理,说明杂类草提高了地衣寄生菌/腐生菌的相对丰度。
3.3 植物功能群移除对土壤真菌群落影响机制
本研究中土壤真菌丰富度指数(Chao1、Ace指数)与根系生物量、根系碳氮比呈负相关。这可能是因为根系生物量的增加,会增加土壤资源输入(如根系分泌物),促进共生营养型真菌定殖,扩大了共生营养型真菌在群落中的优势,从而改变了土壤真菌的多样性,降低了其他土壤真菌的丰富度[14]。根系碳氮比影响根系分泌物的增加、根系凋落物的分解速率。根系凋落物碳氮比低,有利于分解,可能为真菌提供了较高的食物资源多样性,有利于缓解土壤真菌群落内的竞争关系,进而提高土壤真菌的丰富度[43]。本研究中土壤真菌丰富度指数与土壤可溶性有机碳呈负相关。Yang等[41]的研究同样表明,青藏高原可溶性有机碳对土壤真菌多样性、丰富度有重要影响。相比简单的化合物,真菌更倾向于利用结构复杂的DOC作为碳源[44],DOC含量的变化可能影响不同真菌类群间的协同竞争关系,改变土壤真菌多样性。影响土壤真菌多样性的研究结果往往并不一致。姜鑫等[14]在高寒草地的移除实验表明,整体而言,植物功能群和土壤理化性质对土壤微生物多样性影响不显著,植物移除对土壤真菌香农指数无显著影响。本研究进一步验证了植物移除对土壤真菌群落香农指数、均匀度指数、辛普森指数均无显著影响。研究发现,根系碳氮含量与土壤真菌多样性不相关[45]。相反,Francioli等[21]的研究表明植物功能群对腐生真菌群落结构有显著影响,这种影响与根系碳氮比的差异密切相关。赵兴鸽等[36]研究发现土壤真菌丰富度与TN正相关,与SOC和土壤C:N负相关。Kang等[46]的研究表明高寒草甸土壤真菌多样性不仅与TN、SOC、C:N相关,还与TC 、SWC显著相关。其他高寒草甸研究表明,土壤真菌群落结构变化与SWC、SOC、TN、
NH+4 -N正相关,与植物根系生物量负相关[15]。本研究中土壤真菌群落结构和功能与环境因子相关性不显著,可能是因为植物移除处理3年后植物根系和土壤理化性质变化不显著。土壤真菌多样性和群落结构在不同植物功能群移除处理间无显著差异,可能是由于土壤真菌多样性与群落结构主要受空间、气候、土壤理化性质影响,受植物多样性影响较小[41]。赵文等[15]的研究结果显示土壤真菌群落结构受植物地上部分的影响小于植物-土壤相互作用。本研究中不同植物功能群移除处理下土壤真菌群落结构发生变化,但真菌多样性、根系性质和土壤理化性质变化不显著。为减少对土壤的扰动,植物移除实验一般剪除植物地上部分,而将地下根系保留在土壤中,进一步探索植物功能群移除对土壤真菌群落的影响还需要从更长的时间尺度上进行研究,并进一步分析移除植物的根系存留在土壤中的影响。
致谢:感谢陈鹏老师在真菌高通量测定以及万文结老师在文章写作过程中给予的帮助,感谢南京农业大学资源与环境科学学院郭辉教授与胡水金教授提供实验平台。感谢兰州大学和甘南草原生态系统国家野外科学观测研究站为采样提供的条件。
1 1)如需查阅附表内容请登录《植物科学学报》网站(http://www.plantscience.cn)查看本期文章。2 1)如需查阅附表内容请登录《植物科学学报》网站(http://www.plantscience.cn)查看本期文章。 -
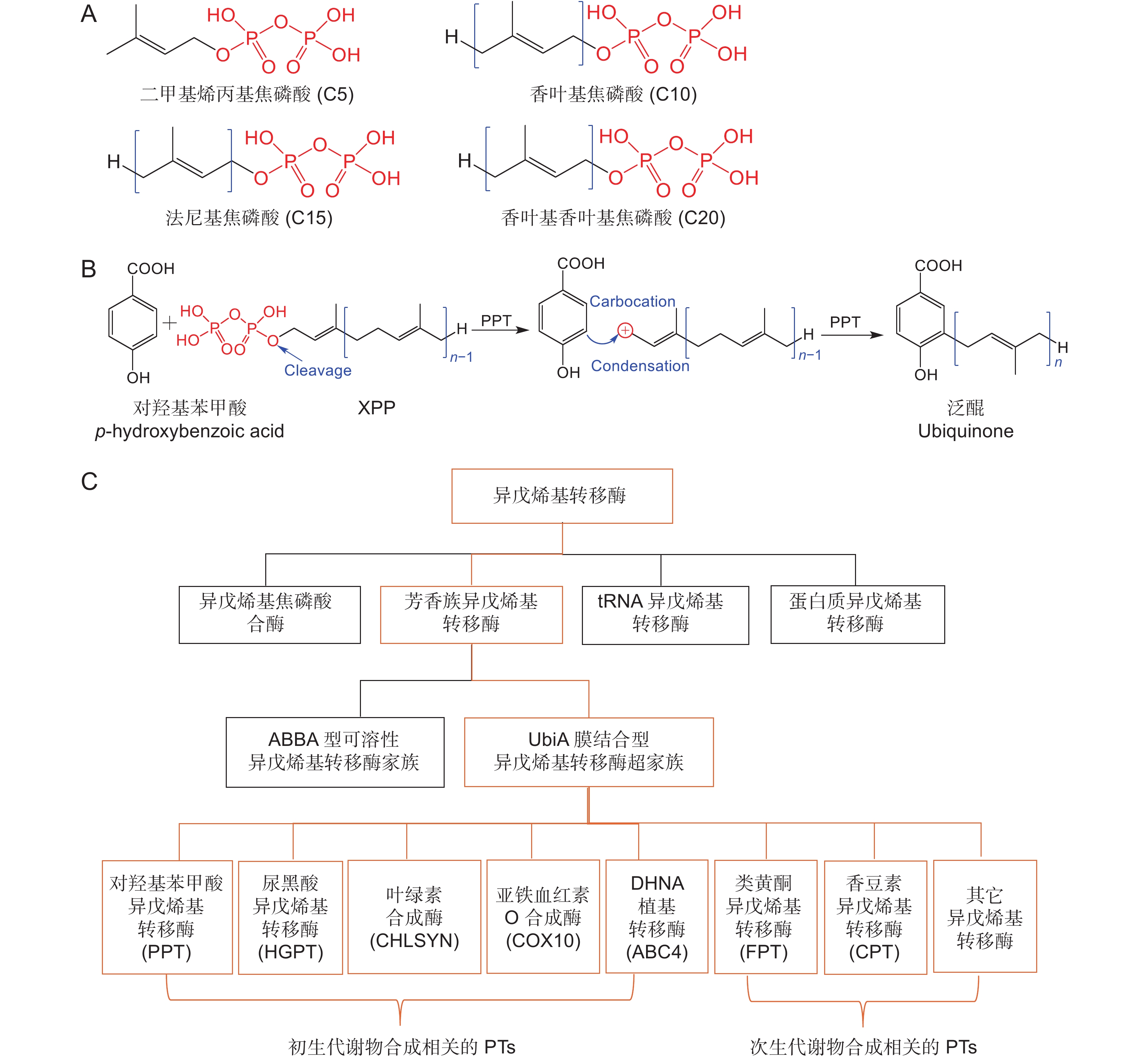
图 1 UbiA异戊烯基转移酶的分类、一般催化机制和供体的结构式
A:异戊二烯焦磷酸结构式;B:UbiA异戊烯基转移酶的一般催化机制;C:异戊烯基转移酶的分类。
Figure 1. Classification and general catalytic mechanism of UbiA prenyltransferases and structural formula of isoprenyl diphosphates
A: Structural formula of isoprenyl diphosphates; B: General catalytic mechanism of UbiA superfamily prenyltransferases; C: Classification of prenyltransferases.
表 1 植物类黄酮异戊烯基转移酶的催化特性
Table 1 Catalytic properties of flavonoid prenyltransferases in plants
物种名称
Species name蛋白名称
Protein name底物类型
Substrate type底物名称
Substrate name异戊烯基供体
Prenyl donor异戊烯基取代位点
Prenyl substitution site二价阳离子
Divalent cation参考文献
References苦参
Sophora flavescens Alt.SfN8DT-1 二氢黄酮 Liquiritigenin>Naringenin>
HesperetinDMAPP A环C-8位 Mg2 + [7] SfiLDT 查尔酮 Isoliquiritigenin DMAPP 未知 Mg2 + [33] SfG6DT 异黄酮 Genistein>Biochanina DMAPP、GPP1、FPP1 A环C-6位 Mg2 + >Ni2 + >
Mn2 + >Ca2 +[33] SfFPT 二氢查尔酮 Phloretin DMAPP、GPP2 A环C-3'位 Mg2 + >Ba2 + >Ca2 + >Fe2 + >Co2 + >Cu2 + >Zn2 + >Mn2 + [28] 二氢黄酮 Eriodictyol>Naringenin>
Pinocembrin>Liquiritigenin>
Hesperetin>Isosakuranetin>
Steppogenin>Tsugafolin>
SakuranetinA环C-8位 黄酮 Chrysin A环C-8位 二氢黄酮醇 Taxifolin A环C-8位 大豆
Glycine max (L.) Merr.GmG4DT 紫檀烷 Glycinol>Maackiain DMAPP A环C-4位 Mg2 + >Mn2 + >Co2 + [30] GmG2DT 紫檀烷 Glycinol DMAPP A环C-2位 Mg2 + >Mn2 + [29] GmPT01 紫檀烷 Glycinol DMAPP A环C-2位 Mg2 + [31] GmIDT1 异黄酮 Daidzein>Genistein DMAPP B环 Mg2 + >Mn2 + [29] GmIDT2 异黄酮 Daidzein≈Genistein DMAPP A环 Mg2 + >Mn2 + [29] GmIDT3 异黄酮 Daidzein、Genistein DMAPP 未知 Mg2 + [31] 白羽扇豆
Lupinus albus L.LaPT1 异黄酮 2-Hydroxygenistein>
GenisteinDMAPP B环C-3'位 Mg2 + >Mn2 + >Ni2 + >Co2 + >Zn2 + >Ca2 + [34] LaPT2 黄酮醇 Kaempferol>Kaempferide>Quercetin>Galangin>
Fesitin>MorinDMAPP A环C-8位 Mg2 + [12] 二氢黄酮 Naringenin DMAPP 未知 甘草
Glycyrrhiza uralensis Fisch.GuA6DT 黄酮 Apigenin>Chrysin>
Diosmtin>Luteolin>
Norartocarpetin>
ChrysoeroilDMAPP、GPP1 A环C-6位 Mg2 + >Mn2 + >Zn2 + >Fe2 + >Co2 + >Ca2 + >Ba2 + [11] GuILDT 查尔酮 2',4'-Dihydroxychalcone>
Isoliquiritigenin>
2,4,2',4'-Tetrahydro-xychalcone>
NaringeninchalconeDMAPP A环C-3'位 Mg2 + >Co2 + >Ni2 + >Fe2 + >Ba2 + >Mn2 + >Ca2 + [35] 百脉根
Lotus japonicus L.LjG6DT 异黄酮 Genistein DMAPP A环C-6位 Mg2 + >Co2 + >Mn2 + >Ca2 + >Zn2 + >Fe2 + [36] 补骨脂
Psoralea corylifolia (L.) Medik.PcM4DT 紫檀烷 Maackiain>3-Hydroxy-9-methoxypterocarpan DMAPP A环C-4位 Mg2 + >Mn2 + >Co2 + >Fe2 + >Ba2 + >Sr2 + >Ca2 + >Sn2 + >Ni2 + >Zn2 + [37] 桑
Morus alba L.MaIDT 查尔酮 Isoliquiritigenin>
2',4'-Dihydroxychalcone>
2,4,2',4'-Tetrahydroxychalcone>
ButeinDMAPP、GPP1 A环C-3'位 Mg2 + >Ba2 + >Ca2 + >Mn2 + >Fe2 + >Ni2 + [32] 异黄酮 Genistein>2'-Hydroxygenistein A环C-6位 黄酮 Apigenin A环C-6位 柘树
Cudrania tricuspidata (Carr.) Bur.CtIDT 查尔酮 Isoliquiritigenin>
2,4,2',4'-Tetrahydroxychalcone>
2',4'-Dihydroxychalcone>
ButeinDMAPP、GPP1 A环C-3'位 Mg2 + >Mn2 + >Ca2 + >Fe2 + >Ba2 + [32] 异黄酮 2'-Hydroxygenistein>
GenisteinA环C-6位 大麻
Cannabis sativa L.CsPT3 黄酮 Chrysoeriol>Apigenin DMAPP、GPP A环C-6位 Mg2 + [38] CsPT8 黄酮 Apigenin DMAPP 未知 Mg2 + [38] 啤酒花
Humulus lupulus L.HlPT-1 查尔酮 Naringenin chalcone DMAPP A环C-3'位 Mg2 + [39] 柔毛淫羊藿
Epimedium pubescens Maxim.EpPT8 黄酮醇 Kaempferol> Quercetin DMAPP A环C-8位 Mg2 + [40] 黄酮 Apigenin 箭叶淫羊藿
Epimedium sagittatum (Sieb. et Zucc.) Maxim.EsPT2 黄酮醇 Kaempferol>Kaempferide DMAPP A环C-8位 Mg2 + [41] 二氢黄酮 Naringenin 注:“>”用于表示对底物的催化活性顺序;1 研究只证明提供了该供体与最适底物发生异戊烯基化反应;2 GPP作为供体时,SfFPT仅催化pinocembrin,isosakuranetin和naringenin发生异戊烯基化反应。 Notes: “>” indicates order of catalytic activity to the substrate; 1 Prenylation of the donor with an optimal substrate is demonstrated; 2 SfFPT only catalyzed prenylation of pinocembrin, isosakuranetin, and naringenin when GPP was used as the prenyl donor. 表 2 植物中香豆素异戊烯基转移酶的催化特性
Table 2 Catalytic properties of coumarin prenyltransferases in plants
物种名称
Species
name蛋白名称
Protein name底物名称
Substrate name异戊烯基
供体
Prenyl donor异戊烯基取代位点
Prenyl substitution
site二价阳离子
Divalent cation参考文献
References欧芹Petroselinum crispum (Mill.) Hill PcPT Umbelliferone DMAPP C-6位>C-8位 Mg2 + [44] 欧防风Pastinaca sativa L. PsPT1 Umbelliferone DMAPP C-6位>C-8位 Mg2 + [42] PsPT2 Umbelliferone DMAPP C-8位>C-6位 Mg2 + [42] 柠檬Citrus limon (L.) Burm. F. ClPT1 Umbelliferone>Esculetin>5,7-hydroxycoumarin
>5-Methoxy-7-hydroxycoumarinGPP C-8位 Mg2 + [45] 无花果Ficus carica L. FcPT1 Umbelliferone DMAPP C-6位 Mg2 + [46] 5-Methoxy-7-hydroxycoumarin DMAPP 未知 Mg2 + 葡萄柚Citrus paradisi Macf. CpPT1 5,7-Dihydroxycoumarin, 8-Hydroxybergapten 5-Hydroxy-7-methoxycoumarin, Bergaptol, GPP 5-OH或8-OH Mg2 + >Ni2 + >Co2 + >Mn2 + >Zn2 + >Ca2 + [47] CpPT3 Umbelliferone GPP C-8位 Mg2 + [47] 小苦橙Citrus micrantha Wester CmiPT1a / b Bergaptol和Xanthotoxol GPP 5-OH或8-OH Mg2 + [47] 明日叶Angelica keiskei (Miquel) Koidz. AkPT1 Bergaptol和Xanthotoxol DMAPP 5-OH或8-OH Mg2 + >Mn2 + >Ca2 + [47] 大豆Glycine max (L.) Merr. GmC4DT Coumestrol DMAPP C-4位 Mg2 + >Mn2 + [29] 九里香Murraya exotica L. MePT1 Umbelliferone GPP C-8位、C-6位和7-OH Mg2 + [48] -
[1] Winkelblech J,Fan AL,Li SM. Prenyltransferases as key enzymes in primary and secondary metabolism[J]. Appl Microbiol Biotechnol,2015,99 (18):7379−7397. doi: 10.1007/s00253-015-6811-y
[2] Yang YH,Ke N,Liu SX,Li WK. Structural and biochemical analysis of intramembrane prenyltransferases in the UbiA superfamiIy[J]. Methods Enzymol,2017,584:309−347.
[3] Li WK. Bringing bioactive compounds into membranes:the UbiA superfamily of intramembrane aromatic prenyltransferases[J]. Trends Biochem Sci,2016,41 (4):356−370. doi: 10.1016/j.tibs.2016.01.007
[4] Bonitz T,Alva V,Saleh O,Lupas AN,Heide L. Evolutionary relationships of microbial aromatic prenyltransferases[J]. PLoS One,2011,6 (11):e27336. doi: 10.1371/journal.pone.0027336
[5] Young IG,Leppik RA,Hamilton JA,Gibson F. Biochemical and genetic studies on ubiquinone biosynthesis in Escherichia coli K-12:4-hydroxybenzoate octaprenyltransferase[J]. J Bacteriol,1972,110 (1):18−25. doi: 10.1128/jb.110.1.18-25.1972
[6] Wang J,Chu SS,Zhu Y,Cheng H,Yu DY. Positive selection drives neofunctionalization of the UbiA prenyltransferase gene family[J]. Plant Mol Biol,2015,87 (4-5):383−394. doi: 10.1007/s11103-015-0285-2
[7] Sasaki K,Mito K,Ohara K,Yamamoto H,Yazaki K. Cloning and characterization of naringenin 8-prenyltransferase,a flavonoid-specific prenyltransferase of Sophora flavescens[J]. Plant Physiol,2008,146 (3):1075−1084. doi: 10.1104/pp.107.110544
[8] Bo ST, Chang SK, Zhu H, Jiang YM, Yang B. Naturally occurring prenylated stilbenoids: food sources, biosynthesis, applications and health benefits[J]. Crit Rev Food Sci Nutr, 2022. Doi: 10.1080/10408398.2022.2056131.
[9] De Bruijn WJC,Levisson M,Beekwilder J,van Berkel WJH,Vincken JP. Plant aromatic prenyltransferases:tools for microbial cell factories[J]. Trends Biotechnol,2020,38 (8):917−934. doi: 10.1016/j.tibtech.2020.02.006
[10] Marin M,Manez S. Recent trends in the pharmacological activity of isoprenyl phenolics[J]. Curr Med Chem,2013,20 (2):272−279. doi: 10.2174/092986713804806676
[11] Li JH,Chen RD,Wang RS,Liu X,Xie D,et al. GuA6DT,a regiospecific prenyltransferase from Glycyrrhiza uralensis,catalyzes the 6-prenylation of flavones[J]. ChemBioChem,2014,15 (11):1673−1681. doi: 10.1002/cbic.201402160
[12] Liu JY,Xia YY,Jiang WB,Shen GA,Pang YZ. LaPT2 gene encodes a flavonoid prenyltransferase in white lupin[J]. Front Plant Sci,2021,12:673337. doi: 10.3389/fpls.2021.673337
[13] Okada K,Ohara K,Yazaki K,Nozaki K,Uchida N,et al. The AtPPT1 gene encoding 4-hydroxybenzoate polyprenyl diphosphate transferase in ubiquinone biosynthesis is required for embryo development in Arabidopsis thaliana[J]. Plant Mol Biol,2004,55 (4):567−577. doi: 10.1007/s11103-004-1298-4
[14] Ohara K,Yamamoto K,Hamamoto M,Sasaki K,Yazaki K. Functional characterization of OsPPT1,which encodes p-hydroxybenzoate polyprenyltransferase involved in ubiquinone biosynthesis in Oryza sativa[J]. Plant Cell Physiol,2006,47 (5):581−590. doi: 10.1093/pcp/pcj025
[15] Yazaki K,Kunihisa M,Fujisaki T,Sato F. Geranyl diphosphate:4-hydroxybenzoate geranyltransferase from Lithospermum erythrorhizon:cloning and characterization of a key enzyme in Shikonin biosynthesis[J]. J Biol Chem,2002,277 (8):6240−6246. doi: 10.1074/jbc.M106387200
[16] Ohara K,Muroya A,Fukushima N,Yazaki K. Functional characterization of LePGT1,a membrane-bound prenyltransferase involved in the geranylation of p-hydroxybenzoic acid[J]. Biochem J,2009,421 (2):231−241. doi: 10.1042/BJ20081968
[17] Wang S,Wang RS,Liu T,Zhan ZL,Kang LP,et al. Production of 3-geranyl-4-hydroxybenzoate acid in yeast,an important intermediate of Shikonin biosynthesis pathway[J]. FEMS Yeast Res,2017,17 (7):fox065.
[18] Venkatesh TV,Karunanandaa B,Free DL,Rottnek JM,Baszis SR,Valentin HE. Identification and characterization of an Arabidopsis homogentisate phytyltransferase paralog[J]. Planta,2006,223 (6):1134−1144. doi: 10.1007/s00425-005-0180-1
[19] Sadre R,Gruber J,Frentzen M. Characterization of homogentisate prenyltransferases involved in plastoquinone-9 and tocochromanol biosynthesis[J]. FEBS Lett,2006,580 (22):5357−5362. doi: 10.1016/j.febslet.2006.09.002
[20] Tian L,DellaPenna D,Dixon RA. The pds2 mutation is a lesion in the Arabidopsis homogentisate solanesyltransferase gene involved in plastoquinone biosynthesis[J]. Planta,2007,226 (4):1067−1073. doi: 10.1007/s00425-007-0564-5
[21] 姚兴兰,王磊,张兰. 植物维生素E生物强化研究进展[J]. 生物技术进展,2020,10(5):479−486. doi: 10.19586/j.2095-2341.2020.0046 Yao XL,Wang L,Zhang L. Progress of vitamin E biofortification in plants[J]. Current Biotechnology,2020,10 (5):479−486. doi: 10.19586/j.2095-2341.2020.0046
[22] Eckhardt U,Grimm B,Hörtensteiner S. Recent advances in chlorophyll biosynthesis and breakdown in higher plants[J]. Plant Mol Biol,2004,56 (1):1−14. doi: 10.1007/s11103-004-2331-3
[23] Hederstedt L. Heme A biosynthesis[J]. Biochim Biophys Acta,2012,1817 (6):920−927. doi: 10.1016/j.bbabio.2012.03.025
[24] Basset GJ,Latimer S,Fatihi A,Soubeyrand E,Block A. Phylloquinone (vitamin K1):occurrence,biosynthesis and functions[J]. Mini-Rev Med Chem,2017,17 (12):1028−1038.
[25] Ming LG,Lv X,Ma XN,Ge BF,Zhen P,et al. The prenyl group contributes to activities of phytoestrogen 8-prenynaringenin in enhancing bone formation and inhibiting bone resorption in vitro[J]. Endocrinology,2013,154 (3):1202−1214. doi: 10.1210/en.2012-2086
[26] Shi SC,Li JC,Zhao XM,Liu QB,Song SJ. A comprehensive review:biological activity,modification and synthetic methodologies of prenylated flavonoids[J]. Phytochemistry,2021,191:112895. doi: 10.1016/j.phytochem.2021.112895
[27] Yang XM,Jiang YM,Yang JL,He JR,Sun J,et al. Prenylated flavonoids,promising nutraceuticals with impressive biological activities[J]. Trends Food Sci Technol,2015,44 (1):93−104. doi: 10.1016/j.jpgs.2015.03.007
[28] Chen RD,Liu X,Zou JH,Yin YZ,Ou B,et al. Regio- and stereospecific prenylation of flavonoids by Sophora flavescens prenyltransferase[J]. Adv Synth Catal,2013,355 (9):1817−1828. doi: 10.1002/adsc.201300196
[29] Yoneyama K,Akashi T,Aoki T. Molecular characterization of soybean pterocarpan 2-dimethylallyltransferase in glyceollin biosynthesis:local gene and whole-genome duplications of prenyltransferase genes led to the structural diversity of soybean prenylated isoflavonoids[J]. Plant Cell Physiol,2016,57 (12):2497−2509. doi: 10.1093/pcp/pcw178
[30] Akashi T,Sasaki K,Aoki T,Ayabe S,Yazaki K. Molecular cloning and characterization of a cDNA for pterocarpan 4-dimethylallyltransferase catalyzing the key prenylation step in the biosynthesis of glyceollin,a soybean phytoalexin[J]. Plant Physiol,2009,149 (2):683−693. doi: 10.1104/pp.108.123679
[31] Sukumaran A,McDowell T,Chen L,Renaud J,Dhaubhadel S. Isoflavonoid-specific prenyltransferase gene family in soybean:GmPT01,a pterocarpan 2-dimethylallyltransferase involved in glyceollin biosynthesis[J]. Plant J,2018,96 (5):966−981. doi: 10.1111/tpj.14083
[32] Wang RS,Chen RD,Li JH,Liu X,Xie KB,et al. Molecular characterization and phylogenetic analysis of two novel regio-specific flavonoid prenyltransferases from Morus alba and Cudrania tricuspidata[J]. J Biol Chem,2014,289 (52):35815−35825. doi: 10.1074/jbc.M114.608265
[33] Sasaki K,Tsurumaru Y,Yamamoto H,Yazaki K. Molecular characterization of a membrane-bound prenyltransferase specific for isoflavone from Sophora flavescens[J]. J Biol Chem,2011,286 (27):24125−24134. doi: 10.1074/jbc.M111.244426
[34] Shen GA,Huhman D,Lei ZT,Snyder J,Sumner LW,Dixon RA. Characterization of an isoflavonoid-specific prenyltransferase from Lupinus albus[J]. Plant Physiol,2012,159 (1):70−80. doi: 10.1104/pp.112.195271
[35] Li JH,Chen RD,Wang RS,Liu X,Xie KB,et al. Biocatalytic access to diverse prenylflavonoids by combining a regiospecific C-prenyltransferase and a stereospecific chalcone isomerase[J]. Acta Pharm Sin B,2018,8 (4):678−686. doi: 10.1016/j.apsb.2018.01.009
[36] Liu JY,Jiang WB,Xia YY,Wang XM,Shen GA,Pang YZ. Genistein-specific G6DT gene for the inducible production of wighteone in Lotus japonicus[J]. Plant Cell Physiol,2018,59 (1):128−141. doi: 10.1093/pcp/pcx167
[37] He JB,Dong ZY,Hu ZM,Kuang Y,Fan JR,et al. Regio-specific prenylation of pterocarpans by a membrane-bound prenyltransferase from Psoralea corylifolia[J]. Org Biomol Chem,2018,16 (36):6760−6766. doi: 10.1039/C8OB01724G
[38] Rea KA,Casaretto JA,Al-Abdul-Wahid MS,Sukumaran A,Geddes-Mcalister J,et al. Biosynthesis of cannflavins A and B from Cannabis sativa L.[J]. Phytochemistry,2019,164:162−171. doi: 10.1016/j.phytochem.2019.05.009
[39] Tsurumaru Y,Sasaki K,Miyawaki T,Uto Y,Momma T,et al. HlPT-1,a membrane-bound prenyltransferase responsible for the biosynthesis of bitter acids in hops[J]. Biochem Biophys Res Commun,2012,417 (1):393−398. doi: 10.1016/j.bbrc.2011.11.125
[40] Shen G,Luo Y,Yao Y,Meng G,Zhang Y,et al. The discovery of a key prenyltransferase gene assisted by a chromosome-level Epimedium pubescens genome[J]. Front Plant Sci,2022,13:1034943.
[41] Wang PP,Li CJ,Li XD,Huang WJ,Wang Y,et al. Complete biosynthesis of the potential medicine icaritin by engineered Saccharomyces cerevisiae and Escherichia coli[J]. Sci Bull,2021,66 (18):1906−1916. doi: 10.1016/j.scib.2021.03.002
[42] Munakata R,Olry A,Karamat F,Courdavault V,Sugiyama A,et al. Molecular evolution of parsnip (Pastinaca sativa) membrane-bound prenyltransferases for linear and/or angular furanocoumarin biosynthesis[J]. New Phytol,2016,211 (1):332−344. doi: 10.1111/nph.13899
[43] Venugopala KN,Rashmi V,Odhav B. Review on natural coumarin lead compounds for their pharmacological activity[J]. Biomed Res Int,2013,2013:963248.
[44] Karamat F,Olry A,Munakata R,Koeduka T,Sugiyama A,et al. A coumarin-specific prenyltransferase catalyzes the crucial biosynthetic reaction for furanocoumarin formation in parsley[J]. Plant J,2014,77 (4):627−638. doi: 10.1111/tpj.12409
[45] Munakata R,Inoue T,Koeduka T,Karamat F,Olry A,et al. Molecular cloning and characterization of a geranyl diphosphate-specific aromatic prenyltransferase from lemon[J]. Plant Physiol,2014,166 (1):80−90. doi: 10.1104/pp.114.246892
[46] Munakata R,Kitajima S,Nuttens A,Tatsumi K,Takemura T,et al. Convergent evolution of the UbiA prenyltransferase family underlies the independent acquisition of furanocoumarins in plants[J]. New Phytol,2020,225 (5):2166−2182. doi: 10.1111/nph.16277
[47] Munakata R,Olry A,Takemura T,Tatsumi K,Ichino T,et al. Parallel evolution of UbiA superfamily proteins into aromatic O-prenyltransferases in plants[J]. Proc Natl Acad Sci USA,2021,118 (17):e2022294118. doi: 10.1073/pnas.2022294118
[48] Li N,Liu X,Zhang ML,Zhang ZK,Zhang BB,et al. Characterization of a coumarin C-/O-prenyltransferase and a quinolone C-prenyltransferase from Murraya exotica[J]. Org Biomol Chem,2022,20 (28):5535−5542. doi: 10.1039/D2OB01054B
[49] Li HX,Ban ZN,Qin H,Ma LY,King AJ,Wang GD. A heteromeric membrane-bound prenyltransferase complex from hop catalyzes three sequential aromatic prenylations in the bitter acid pathway[J]. Plant Physiol,2015,167 (3):650−659. doi: 10.1104/pp.114.253682
[50] Fiesel T,Gaid M,Müller A,Bartels J,El-Awaad I,et al. Molecular cloning and characterization of a xanthone prenyltransferase from Hypericum calycinum cell cultures[J]. Molecules,2015,20 (9):15616−15630. doi: 10.3390/molecules200915616
[51] Nagia M,Gaid M,Biedermann E,Fiesel T,El-Awaad I,et al. Sequential regiospecific gem-diprenylation of tetrahydroxyxanthone by prenyltransferases from Hypericum sp.[J]. New Phytol,2019,222 (1):318−334. doi: 10.1111/nph.15611
[52] Akinwumi BC,Bordun KAM,Anderson HD. Biological activities of stilbenoids[J]. Int J Mol Sci,2018,19 (3):792. doi: 10.3390/ijms19030792
[53] Yang TH,Fang LL,Sanders S,Jayanthi S,Rajan G,et al. Stilbenoid prenyltransferases define key steps in the diversification of peanut phytoalexins[J]. J Biol Chem,2018,293 (1):28−46. doi: 10.1074/jbc.RA117.000564
[54] Zhong ZH,Zhu W,Liu SZ,Guan QJ,Chen X,et al. Molecular characterization of a geranyl diphosphate-specific prenyltransferase catalyzing stilbenoid prenylation from Morus alba[J]. Plant Cell Physiol,2018,59 (11):2214−2227.
[55] Munakata R,Takemura T,Tatsumi K,Moriyoshi E,Yanagihara K,et al. Isolation of Artemisia capillaris membrane-bound di-prenyltransferase for phenylpropanoids and redesign of artepillin C in yeast[J]. Commun Biol,2019,2:384. doi: 10.1038/s42003-019-0630-0
[56] Saeki H,Hara R,Takahashi H,Iijima M,Munakata R,et al. An aromatic farnesyltransferase functions in biosynthesis of the anti-HIV meroterpenoid daurichromenic acid[J]. Plant Physiol,2018,178 (2):535−551. doi: 10.1104/pp.18.00655
[57] Luo XZ,Reiter MA,D’Espaux L,Wong J,Denby CM,et al. Complete biosynthesis of cannabinoids and their unnatural analogues in yeast[J]. Nature,2019,567 (7746):123−126. doi: 10.1038/s41586-019-0978-9
[58] Marsafari M,Samizadeh H,Rabiei B,Mehrabi A,Koffas M,Xu P. Biotechnological production of flavonoids:an update on plant metabolic engineering,microbial Host selection,and genetically encoded biosensors[J]. Biotechnol J,2020,15 (8):1900432. doi: 10.1002/biot.201900432
[59] Levisson M,Araya-Cloutier C,de Bruijn WJC,van Der Heide M,Salvador López JM,et al. Toward developing a yeast cell factory for the production of prenylated flavonoids[J]. J Agric Food Chem,2019,67 (49):13478−13486. doi: 10.1021/acs.jafc.9b01367
-
期刊类型引用(12)
1. 张琦,蒲婷婷,王艺儒,胡银凤,李华威,韩卫娟,索玉静,傅建敏. 不同柿种质果实品质多样性评价. 中国农业大学学报. 2025(02): 61-71 .  百度学术
百度学术
2. 罗赛男,张文. 我国猕猴桃质量安全标准体系现状及建议. 食品安全质量检测学报. 2025(01): 243-249 .  百度学术
百度学术
3. 王宝趁. 不同猕猴桃品种果实品质比较研究. 现代农业科技. 2024(02): 140-143+164 .  百度学术
百度学术
4. 张慧艺,汪丽霞,吴伊静,毕旭灿,王刚,赵四清,徐昌杰,陈昆松. 18份胡柚种质果实品质分析与综合评价. 果树学报. 2024(06): 1033-1043 .  百度学术
百度学术
5. 李艺潇,陈建华,闫芳芳,焦子源,李纪军,马培芳. 韭菜种质资源营养与感官品质评价. 中国瓜菜. 2024(08): 109-116 .  百度学术
百度学术
6. 孔令硕,樊丁宇,靳娟,杨磊,郝庆,田嘉. 灰枣及其芽变品种果实品质评价. 现代农业科技. 2024(16): 119-123+131 .  百度学术
百度学术
7. 傅一凡,王周倩,邱栋梁,黄文俊,钟彩虹. 防雹网对‘东红’猕猴桃果实耐贮性和品质的影响. 植物科学学报. 2024(04): 533-542 .  本站查看
本站查看
8. 黄文俊,王周倩,杨洁,张琦,钟彩虹. 不同干物质含量对金艳猕猴桃果实品质和感官评价的影响. 落叶果树. 2024(05): 20-25 .  百度学术
百度学术
9. 汪梦诗,祁雅楠,赵沁雨,兰天,鲍诗晗,孙翔宇,马婷婷. 五种陕西主栽猕猴桃品种营养品质与香气特征解析. 食品工业科技. 2024(23): 272-281 .  百度学术
百度学术
10. 贺迪,钟彩虹,朱佳慧,潘慧,李文艺,杨洁,黄跃,刘普,李黎. 山梨与中华猕猴桃种间杂交群体的抗病性评价及抗病机制的研究. 果树学报. 2024(11): 2235-2249 .  百度学术
百度学术
11. 唐玉凤,朱世银,蔡荣靖,胡志芳,田虹,张小龙,王世敏,蒙元燕,全勇. 威信县5个主要猕猴桃品种的果实品质比较. 云南农业科技. 2024(05): 1-5 .  百度学术
百度学术
12. 公旭晨,毛积鹏,高磊,林孟飞,卢玉鹏,王小玲,高柱. 6个猕猴桃品种果实发育期品质变化规律分析. 中国南方果树. 2024(06): 216-224+232 .  百度学术
百度学术
其他类型引用(6)
-
其他相关附件
-
DOCX格式
落艳娇附图1-2 点击下载(980KB)
-




 下载:
下载: