Genome-wide identification and characterization of MADS-box gene family of Rhododendron griersonianum Balf. f. et Forrest
-
摘要:
MADS-box是在植物生长发育过程中扮演重要角色的一类转录因子,特别是花器官形成、开花时间控制及果实发育与成熟等过程。本研究基于朱红大杜鹃(Rhododendron griersonianum Balf. f. et Forrest)全基因组测序数据,利用生物信息学方法从中鉴定出 81 个 MADS-box 基因,并进行了分析。根据系统发育关系和蛋白结构将 MADS-box分为两类:Type-Ⅰ型包含 24 个基因,Type-Ⅱ型包含57个基因,这些基因不均匀地分布于12条染色体上;81个MADS-box 基因中,存在 6 对片段复制和 1 对串联复制基因,且它们经历了纯化选择作用;同时,基因启动子区域含有光响应、植物生长、激素反应和胁迫响应等顺式作用元件。综上,本研究鉴定了朱红大杜鹃的MADS-box家族转录因子,为深入研究其 MADS-box 蛋白的生物学功能提供了基础。
-
关键词:
- 朱红大杜鹃 /
- MADS-box 基因家族 /
- 生物信息学 /
- 基因结构
Abstract:MADS-box genes play an important role in plant growth and development, especially in processes such as floral organ formation, flowering time regulation, and fruit development and ripening. Based on whole-genome sequencing data of Rhododendron griersonianum Balf. f. et Forrest, 81 MADS-box genes were identified and analyzed using bioinformatics methods. Phylogenetic analysis and protein structure classification divided these genes into two classes, including 24 genes in Type-Ⅰand 57 genes in Type-Ⅱ. The MADS-box genes were unevenly distributed across 12 chromosomes, with no genes located on chromosome 2. Among the 81 MADS-box genes, six pairs showed segmental duplications and one pair showed tandem duplication, all of which have undergone purifying selection. The promoter regions of the MADS-box genes contained elements involved in light response, plant growth, hormone response, and stress response. Overall, the identification of MADS-box gene family members provides a reliable reference for further studies on the biological functions of MADS-box proteins in R. griersonianum
-
Keywords:
- Rhododendron griersonianum /
- MADS-box gene family /
- Bioinformatics /
- Gene structure
-
朱红大杜鹃(Rhododendron griersonianum Balf. f. et Forrest)是杜鹃花属(Rhododendron)朱红大杜鹃亚族的唯一物种,也是中国特有物种。全世界杜鹃花品种中超过10%的亲本都与其有着直接或间接的关系,是名副其实的明星物种。然而,该物种自 1924 年被发表后,在野外几乎销声匿迹,直到 1964 年再次被发现[1]。由于人类活动频繁,其生境遭到严重破坏。2020 年,经本课题组多次实地调查发现,其仅残存 322 株于云南腾冲市,为典型的极小种群野生植物[2]。该物种已被列入 The Red List of Rhododendrons[3]和中国高等植物受威胁物种名录[4]中,均被评为极度濒危(CR)等级。朱红大杜鹃具有株形优美、花色鲜艳、花期长且易驯化等优点,研究其花器官分化具有重要意义,而目前与其相关的研究仅限于全基因组测序与组装等方面,对其花发育方面的研究尚未见报道。通常,花器官的分化主要由 MADS-box 基因家族控制,且该基因家族在果实发育中也发挥作用,但迄今为止,尚未见朱红大杜鹃 MADS-box 基因家族的相关报道。
MADS-box 基因家族最初被鉴定为同源异型基因,是植物中研究最广泛的转录因子家族之一[5, 6],同时也广泛存在于动物和真菌中[7],其命名来自酵母(Saccharomyces cerevisiae)MCMI 基因、拟南芥(Arabidopsis thaliana (L.) Heynh.)AGAMOUS 基因、金鱼草 (Antirrhinum majus L.)DEFTCI 基因及人类 SRF基因的首字母缩写[8]。MADS-box 转录因子的主要特征为具有 58~60 个氨基酸的 DNA 结合结构域,位于蛋白质的 N-端区域,称为 MADS 结构域[9]。根据其蛋白的结构域特征和系统发育关系,可将植物 MADS-box 家族划分成两大类,即 Type-Ⅰ型和 Type-Ⅱ型[10]。基于 SRF-like 结构域的差异,Type-Ⅰ型可进一步划分成 Mα、Mβ 和 Mγ 等 3 个亚族[11],这些基因的 N 端都包含一个约180 bp的MADS-box保守区域,且明确缺少 K-box 结构域(Keratin-like domain);Type-Ⅱ型不仅含 MEF2-like MADS 结构域,还含有约 70 个氨基酸的 K-box 结构域、约 30 个氨基酸的 I 结构域(Intervening domain)和长度可变的 C 结构域(Carboxy-terminal domain),故又被称为 MIKC 型 MADS-box 基因[12];基于Ⅱ型结构域所对应的基因结构以及K-box结构域的不同,可将MIKC划分为 MIKC* 型和 MIKCC型[13-15]。此外,K 结构域介导 MADS-box 蛋白的二聚化,并已被证明参与高级复合物的形成;I 结构域在 DNA 结合二聚体的形成中提供特异性;C 结构域是一个高度多样的区域,但已有研究证实 C 结构域在 MADS-box 蛋白相互作用和转录激活中具有关键作用[16]。多项研究发现,大多数植物中的Ⅰ型 MADS-box 基因比Ⅱ型基因更快地经历了基因复制过程,这可能是由于节段基因复制的频率更高和纯化选择更弱所致[9]。
MADS-box 基因家族在植物生长发育过程中承担多种功能,在参与调控植物非生物胁迫响应、开花时间控制、分生组织和花器官确定、果实成熟、胚发育以及根和叶等营养器官发育中具有突出功能[17-21],且在植物生长发育过程中,Ⅰ型基因作用一般弱于Ⅱ型[22]。在植物中发现的第一个MADS-box 基因是参与花发育的Ⅱ型基因,Ⅱ型基因还控制开花时间[23]。目前,许多物种的全基因组测序已完成,对 MADS-box 基因功能的理解已更加深刻。在拟南芥[11]、茄子(Solanum melongena L.) [24]、小麦(Triticum aestivum L.)[25]、水稻(Oryza sativa L.)[26]、毛果杨 (Populus trichocarpa Torr. & Gray)[27]和菠萝(Ananas comosus (L.) Merr.)[28]等植物中,MADS-box基因已得到广泛研究,如 Ruelens 等[29]研究发现,二穗短柄草(Brachypodium distachyum (L.) Beauv.)的 MADS-box在调控花发育的“ABCDE 模型”中扮演着非常重要的角色,E 类基因 (SEP1/2、SEP3 和 SEP4)可以形成所有花器官发育所必需的 MADS-box 蛋白复合物;Ferrario 等[30]在圆叶牵牛(Pharbitis purpurea (L.) Voisgt)的研究中发现,FBP2 基因能调控开花时间,且可以调控花萼片的转化。拟南芥 FLC 亚族基因不仅可以调节其开花时间,还可以改变或消除不同生态型的春化作用[23];高玮林等[31]在对枣(Ziziphus jujuba Mill.)E 类 MADS-box 基因的研究中发现,SEP1/2、SEP3 和 SEP4 同源基因在花发育的整个过程中均有表达,但不同基因在功能和调控模式上均有略微差异,表现出一定的功能分化。在拟南芥中,MADS-box 家族的 AGAMOUS-LIKE6(AGL6)基因在调控其侧生器官发育和开花时间中起关键作用,且在花发育过程中,在所有花器官的近端区域和发育的胚珠中均能检测到 AGL6[20]。孙福辉等[32]对马银花(Rhododendron ovatum (Lindl.) Planch. ex Maxim.)MADS-box 基因家族的研究结果表明,AP1、AP3/PI、AG、SEP 和 MIKC*分支基因均在花器官中特异表达,并构建了花的发育模型。
本研究基于课题组前期获得的朱红大杜鹃全基因组数据[2],使用生物信息学方法,对其 MADS-box 基因的理化性质、基因结构、染色体定位、共线性和系统进化关系等进行分析,研究结果旨在为 MADS-box 基因家族成员的生物学功能分析奠定基础。同时,理解朱红大杜鹃 MADS-box 基因分子的进化特点,为杜鹃花科植物的花型遗传改良和育种工作提供参考。
1. 材料与方法
1.1 朱红大杜鹃 MADS-box 基因的鉴定及理化性质分析
利用 BLAST和HMMER search 方法鉴定 MADS-box 基因。朱红大杜鹃基因组数据来源于本课题组的全基因组测序数据[2],拟南芥基因组数据下载自 TAIR 网站(https://www.arabidopsis.org/)。从 Pfam 网站(http://pfam.xfam.org/)下载与 MADS 结构域(PF00319)对应的隐马尔可夫模型(Hidden Markov model,HMM)文件,用 HMMER search 方法在朱红大杜鹃的蛋白序列中检索 MADS-box 蛋白序列,得出一次结果,然后以拟南芥的 MADS-box 蛋白序列为索引,利用 TBtools[33] 软件进行本地 BLAST 比对。将两种方法鉴定的结果合并取交集,将比对后的MADS-box基因家族蛋白序列,利用NCBI的CDD 数据库(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)和SMART数据库(https://www.ncbi.nlm.nih.gov/Structure/),对其保守结构域进行逐个鉴定分析,剔除结构域不完整、重复的序列,最终获得81个MADS-box转录因子序列。通过在线软件 ExPasy(https://web.expasy.org/protparam/)分析其编码蛋白序列的相对分子质量、理论等电点等基本理化性质。
1.2 系统进化树构建、蛋白基序及保守结构域分析
根据文献获得拟南芥 MADS-box 蛋白序列的登录号[11],在 TAIR 网站下载拟南芥 107 个MADS-box蛋白序列文件。利用 MAGE 11 软件中的 Clustal W 程序,对朱红大杜鹃和拟南芥的MADS-box蛋白序列进行多序列比对。采用邻近法(Neighbor-joining method,NJ)构建系统发育树,参数设置为:1 000 Bootstrap method、P-distance 模型和 Pairwise deletion,使用 Itol 在线工具(https://itol.embl.de/upload.cgi)对进化树进行美化。将朱红大杜鹃的 MADS-box基因家族全部进行提取,采用MEME在线工具(http://meme-suit.org/meme/tools/meme),分析其蛋白基序,基序的最大数目设置为10。利用 NCBI 数据库的 Batch CD-Search 工具,分析 MADS-box蛋白序列的保守结构域,最后使用 TBtools 软件的 Gene structure view(Advanced)插件对蛋白基序及保守结构域进行可视化。
1.3 MADS-box基因的结构分析
在朱红大杜鹃的 GFF3 文件中提取MADS-box基因序列的注释信息,使用 TBtools 的 Amazing optional gene viewer 模块分析内含子与外显子的组成情况,利用TBtools 软件的 Gene structure view(Advanced)插件进行可视化处理。
1.4 MADS-box基因的染色体分布
在全基因组文件中提取 MADS-box基因在染色体上的定位信息,利用 TBtools 软件的 Gene density profile 插件对染色体颜色进行填充,使用 Map Gene2 Chromosome v2(http://mg2c.iask.in/mg2c_v2.0/)在线工具绘制 MADS-box 基因在染色体上的位置。
1.5 MADS-box 启动子顺式作用元件分析
使用 TBtools 软件,在基因组文件中提取 MADS-box 基因转录起始位点上游2 000 bp的序列作为基因的启动子区,利用 Plant CARE在线工具(http://bioinformatics.psb.ugent.be/webtools/plantcare/html)进行分析,预测启动子区中的顺式作用元件,并对分析结果进行统计,将整理后的数据利用 TBtools 软件进行可视化。
1.6 MADS-box的共线性分析
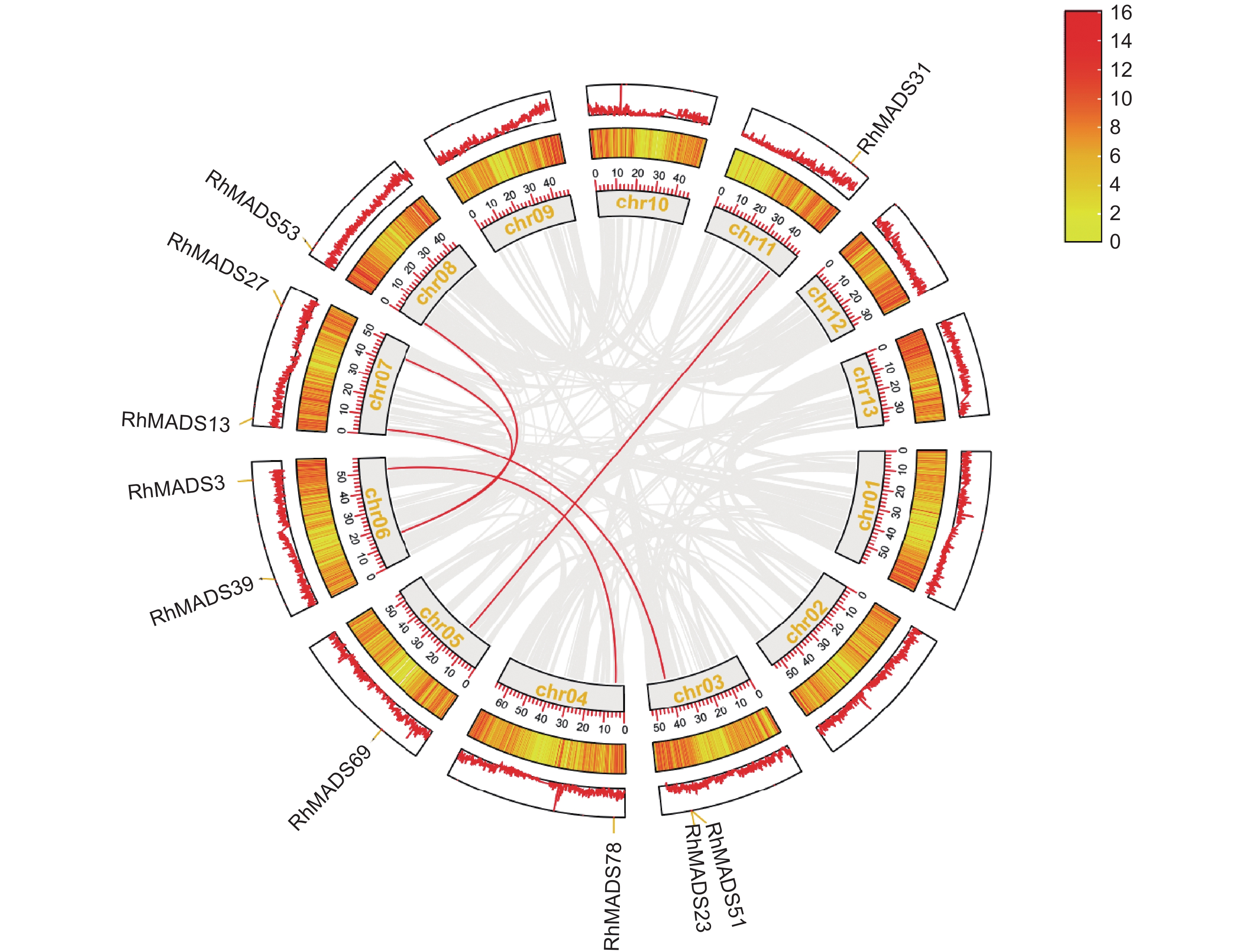
利用基因组数据库 TAIR 和植物数据库 Ensembl Plants 分别下载拟南芥和毛果杨的MADS-box基因组数据,使用TBtools软件的多重共线性扫描工具包(One step MCScanX)对3种植物进行全基因组共线性分析,获得 ctl、gff 和 collinearity 文件。使用TBtools软件获得朱红大杜鹃各染色体长度文件,找出 MADS-box基因中的高亮基因及所在染色体位置,利用Advanced Circos软件绘制其种内共线性关系图;根据朱红大杜鹃、拟南芥及毛果杨的MADS-box基因 ID,从共线性结果中筛选出 三 者间的共线性基因对,利用 TBtools 软件的Dual systeny plot程序绘制种间共线性图。
1.7 MADS-box的基因复制事件
根据 MADS-box 基因两两之间的 BLASTP 比对结果,获取基因复制的同源事件,利用 MCScanX 进行全基因组共线性分析,鉴定片段复制,然后利用 TBtools 软件计算复制基因的非同义替换率(Ka)和同义替换率(Ks),并进一步计算 Ka/Ks 的比值,分析环境选择压力。判断基因复制事件有两个条件:一是两个基因序列相似性大于 75%,二是相似序列长度大于较长序列的 75%[34]。
2. 结果与分析
2.1 MADS-box 的鉴定及理化性质分析
基于朱红大杜鹃的全基因组数据,进行初步筛选,获得 93 个 MADS-box 基因家族候选基因。利用 NCBI 的CDD 库和SMART对保守结构域进行进一步验证,剔除了 12 个不含 MADS-box 结构域或重复的基因,最终获得 81 个 MADS-box基因家族成员,将其命名为RhMADS1~RhMADS81(附表1
1 ))。对 81 个MADS-box的蛋白序列进行理化性质分析,发现不同序列之间差异较大(附表1)。MADS-box蛋白序列的长度为67~575 aa,最长的是RhMADS66,最短的是RhMADS30,平均为 225 aa。理论等电点在4.76~10.66,平均等电点为 8.12,等电点最大的为RhMADS35,最小的为 RhMADS49,其中58 个蛋白的等电点大于 7。蛋白分子量在 7 769.95~
64848.69 Da,最大的为 RhMADS66,最小的为 RhMADS30。在蛋白稳定性方面,不稳定系数小于40,则该蛋白具有稳定性。在 81 个 MADS-box 蛋白序列中,只有 11 个蛋白的不稳定系数小于 40,其余的均大于 40。脂肪系数最大的是RhMADS2,为118.08,最小的是RhMADS26,为56.50。在亲水性方面,平均亲水系数大于0,表示蛋白有一定的疏水性,小于0,则表示具有亲水性;本研究发现,除了3个蛋白序列的平均亲水性大于 0,其余MADS-box蛋白均小于 0,为亲水性蛋白。2.2 MADS-box 系统发育树的构建
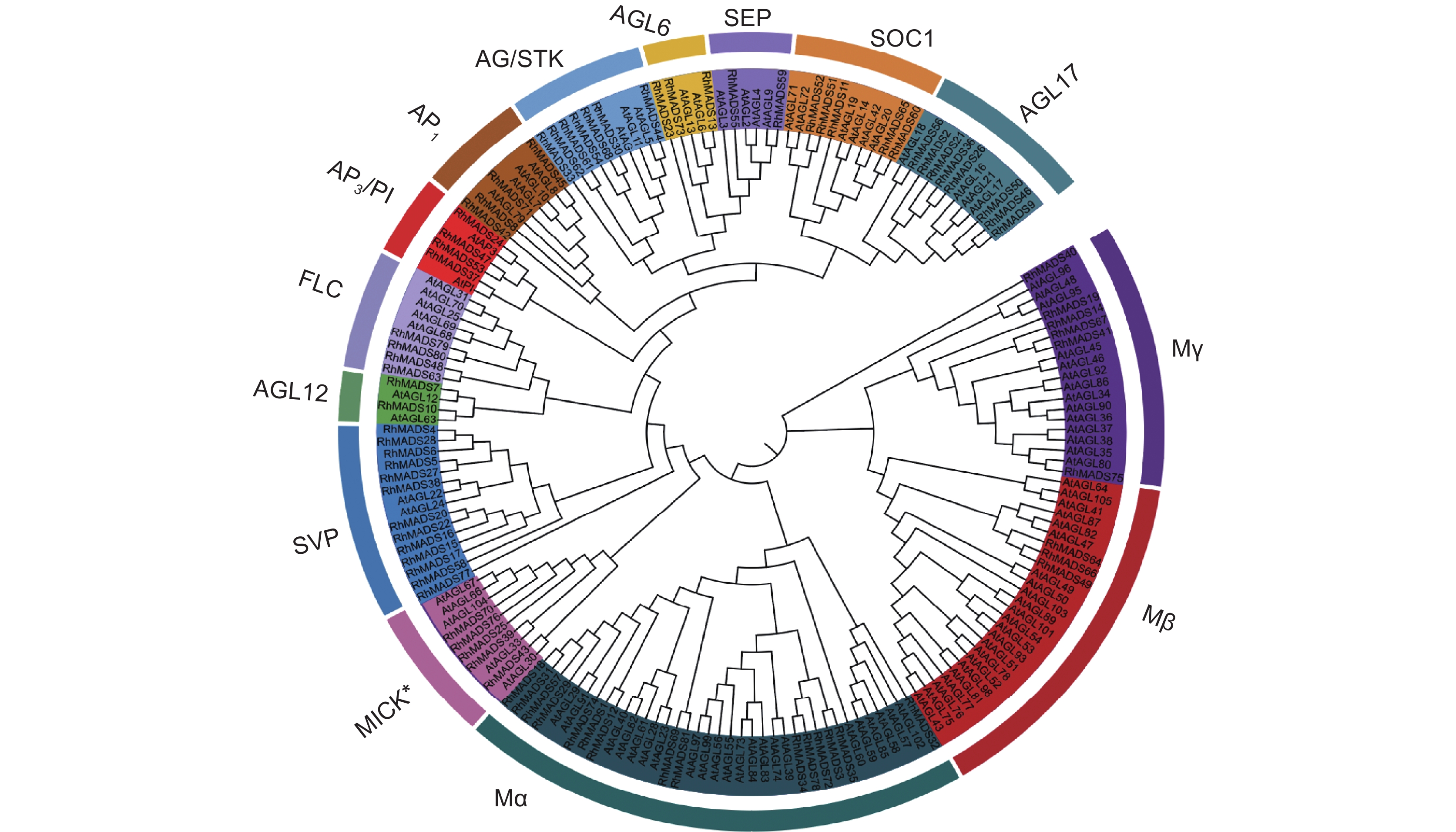
对朱红大杜鹃和拟南芥的 MADS-box蛋白序列进行比对,采用邻接法,构建系统进化树,并用 Itol 工具进行美化。结果显示,81个基因中,有 24个是Ⅰ型基因,其中15个为Mα亚族,3个为Mβ 亚族,6个为 Mγ 亚族;Ⅱ型MADS-box 基因有57 个,其中MICK* 型亚族有5 个,而 MICKC型又可划分为13个亚族,除了OsMADS32、AGL15和FLC 外,其他亚族均含有 MADS-box基因,其中SVP亚族含有的成员最多,为13 个,AGL12和SEP 最少,均为 2 个(图1)。由于 OsMADS32 亚族为单子叶植物所特有,本研究在朱红大杜鹃中未发现。
2.3 MADS-box蛋白基序及保守结构域分析
本研究从朱红大杜鹃 MADS-box 蛋白共鉴定出 10 个保守基序(附图1
2 )),命名为 Motif 1~10。Motif 1 是 MADS 域最明显的特征,仅 RhMADS63不含该基序。Ⅱ型中仅 RhMADS11和RhMADS77 不含 Motif 2;Motif 3 和Motif 4是Ⅱ型 MADS-box的典型结构;而 Motif 5~7和 Motif 10 仅存在于Ⅱ型 MADS-box蛋白序列中,Motif 8和Motif 9仅存在于Ⅰ型蛋白序列中。同一亚族的MADS-box 具有相似的Motif结构,而Ⅱ型的结构组成更加多样化,且存在一些亚家族特异性的保守基序,如 SVP 蛋白序列特有的Motif 5(附图1)。对MADS-box蛋白的保守结构域进行分析发现,所有蛋白序列均含有MADS、MADS_SRF或 MADS_MEF2 结构域(附图1),而 K-box 结构域仅存在于Ⅱ型蛋白序列中。此外,朱红大杜鹃 MADS-box 蛋白还含有其他少数几个结构域。2.4 MADS-box基因结构分析
对朱红大杜鹃MADS-box基因的 CDS 序列和基因组 DNA 序列进行分析发现(附图2
3 )),不同基因的长度和外显子的数目及长度差异较大。外显子数目为 1~12 个,其中,RhMADS39 的外显子最多,为 12 个;Ⅰ型MADS-box 基因有 23 个不含内含子,仅 RhMADS14 含有 1 个内含子;Ⅱ型MADS-box 基因中有 21 个具有内含子,其中 RhMADS43 内含子最多,为11个;其余基因均没有内含子。2.5 MADS-box基因的染色体分布
染色体分布分析结果表明(附图3
4 )),朱红大杜鹃MADS-box 基因家族81 个成员不均匀地分布在 12 条染色体上。其中,第 7 号染色体上分布数量最多,为 14 个,而 2 号染色体上没有分布。染色体长度与 MADS-box 基因的分布没有相关性,最长的第 4 号染色体上仅分布有 5 个基因,而最短的第 12 号染色体上有 8 个。2.6 MADS-box启动子顺式作用元件分析
本研究提取MADS-box转录因子起始位点上游 2 000 bp 的序列作为基因的启动子区,共鉴定出48种顺式作用元件(附图4
4 ) 和附表24 ))。根据其功能注释,可分为:光响应作用元件(23 种)、激素反应作用元件(11 种)、胁迫响应作用元件(6 种)以及植物生长作用元件(8 种)。在MADS-box基因启动子中共鉴定出 876 个光响应元件,主要包括 G-box、Box4、3-AF1 binding site、Chs-Unit 1 m1 等元件,其中,G-box和 Box4 最多,且每个MADS-box至少含有161个与光响应相关的元件;同时,在MADS-box基因启动子中共鉴定出 637 个植物激素反应元件,包括赤霉素、脱落酸、茉莉酸甲酯和水杨酸等相关作用元件。此外,还鉴定出 403 个胁迫响应元件以及 98 个植物生长相关元件。2.7 MADS-box 的共线性分析
种内共线性分析结果表明,共线性 MADS-box 基因分布于3~8 和 11 号染色体上(图2),共检测出 6 对片段重复基因和 1 对串联重复基因 (RhMADS23 与 RhMADS51),这可能是基因重复事件所致,朱红大杜鹃 MADS-box基因在进化过程中可能发生了基因扩张事件。
物种间共线性分析发现, 朱红大杜鹃25个MADS-box基因与拟南芥的基因存在共线性关系(附图5
5 )),有69个基因与毛果杨的基因存在共线性关系。除了在 5~8、11和12号染色体与拟南芥存在共线性基因对外,其余染色体均与拟南芥 MADS-box基因不存在共线性基因对。与毛果杨的共线性分析中,在 3、5~8、11~13 号染色体上存在共线性基因对,其余染色体均无共线性基因对。2.8 MADS-box基因复制事件
Ka/Ks<1,表明基因在进化过程中受到纯化选择;Ka/Ks=1,表明基因受到中性选择;Ka/Ks>1,则表示基因受到正向选择。本研究结果显示,存在共线性基因对的 Ka/Ks 值均小于1(附表3
6 )),表明朱红大杜鹃 MADS-box 基因在进化过程中受到纯化选择作用,基因结构稳定保守,在其基因功能上并无较大差异的分化。3. 讨论
3.1 MADS-box基因家族的鉴定与特征
本研究基于朱红大杜鹃的全基因组测序数据,利用生物信息学方法鉴定出 81 个 MADS-box 基因。根据拟南芥中的分类方法,将其分为 Type-Ⅰ(24个)和 Type-Ⅱ 型(57个)两类。两种类型的成员数目显著差异,可能Ⅱ型基因较Ⅰ型功能复杂,且进化程度更高。目前,在拟南芥[11]、大豆(Glycine max (L.) Merr.)[35]、烟草(Nicotiana tabacum L.)[36]、番茄(Lycopersicon esculentum Mill.) [37]、马银花[32]、萝卜(Raphanus sativus L.) [38]、菊花(Dendranthema morifolium (Ramat.) Tzvel.)[39]、中华猕猴桃(Actinidia chinensis Planch.) [40]和芝麻(Sesamum indicum L.)[41]等植物中分别鉴定出了107、95、183、131、77、144、98、68 和 37 个MADS-box家族成员,各物种 MADS-box 基因的数量有差异,可能与其基因组大小及全基因组复制水平不同有关。理化性质分析结果显示,不同家族成员的蛋白序列长度、等电点、亲水性等差异均比较大,可能与其相对应的功能有关。本研究对朱红大杜鹃 MASD-box的理化性质、系统进化、基因结构、Motif、保守结构域及顺式作用元件等方面进行了分析,研究结果可为进一步探讨该基因家族的功能提供参考。
MADS-box是植物转录因子中研究最为广泛的家族之一,在植物生长发育、胁迫反应以及次生代谢调控等方面起着重要作用[42],尤其是在植物花序、花发育和果实成熟过程中表达最为明显。在植物花器官发育的“ABCDE”模型中,所涉及的基因多为MADS-box 基因家族,这一家族在调控花器官的形成与分化过程中扮演着至关重要的角色,其中不同花序中的特定表达过程受Ⅱ型基因调节[28]。譬如拟南芥的 SVP 亚族基因是花完整发育的网络调控中心,SOC1 亚族基因可以决定开花时间,SOC1 亚族中的 MADS-box 基因为植物开花信号通路的整合因子,AGL17亚族在调控花发育过程中也发挥重要作用,但拟南芥 SVP 亚族仅有两个成员[43]。本研究发现,朱红大杜鹃的 SVP亚族成员多达13个,AGL17和SOC1亚族成员分别为9个和5个。因此,朱红大杜鹃的SVP亚族可能涉及更加复杂的花发育调控,而AGL17和SOC1亚族则可能协同调控花发育和开花时间。拟南芥的AGL6亚族基因在其侧生器官发育和开花时间的调控中同样不可或缺,其在茎生叶原基的远轴和近端区域以及花苞片中均有表达[20]。朱红大杜鹃的AGL6亚族仅有3个成员,可能在进化过程中发生了基因丢失。由于 MADS-box 转录因子在控制植物开花过程中非常重要,研究该基因家族可能有助于理解朱红大杜鹃濒危的原因。此外,MADS-box基因启动子存在较多的光反应、激素反应及胁迫适应相关的顺式作用元件,进一步表明该家族基因在植物的整个生命周期中,特别是在其生长发育阶段以及面对各种环境胁迫时,发挥着不可或缺的关键作用。
3.2 MADS-box家族的基因结构及保守基序
内含子的数量越多、长度越大,基因剪接的方式就越多样化,因而更易影响基因表达,从而调控蛋白质的活性。同时,内含子丢失的基因具有较高的同义替换率,且丢失的内含子比现存的内含子具有更高的突变率[44, 45]。朱红大杜鹃Ⅰ型MADS-box的结构较为简单,其中23个成员没有内含子,仅有RhMADS14含有1个内含子,这可能是MADS-box基因家族多样化过程中经历了多个内含子的丢失所致;而Ⅱ型具有较多的内含子。蛋白基序分析结果表明,同一组中含有最保守的基序,且这些保守基序在群体特异性功能中发挥着重要作用。MADS-box基因均含有1个MADS保守结构域,而Ⅱ型成员还含有1个独有的 K-box 结构域,意味着Ⅱ型基因功能更复杂,与拟南芥[11]、茄子[24]和马铃薯(Solanum tuberosum L.)[46]中的研究结果相似,可为朱红大杜鹃MADS-box的进化关系提供更多的线索。顺式作用元件分析结果表明,启动子区主要的作用元件可分为4大类型:光响应元件、胁迫响应元件、激素反应和植物生长元件。
3.3 MADS-box基因家族的进化分析
在进化过程中,基因复制产生的新基因可能发挥着新的功能,同时也提高植物适应新环境的能力[47]。本研究中,片段重复(6 对)和串联重复(1 对)基因对均属于Ⅱ型和Mα亚家族,属于Ⅱ型亚家族的基因占 60%,这与浙江红山茶(Camellia chekiangoleosa Hu)[48]相似,Ⅱ型 MADS-box的片段重复比Ⅰ型频率高,可能是Ⅱ型基因出现和分化的时间更早,且Ⅱ型基因的结构也更加保守。MADS-box在7号染色体上分布最多,共有14个基因,这可能是由串联重复所致。基因间的共线性是理解两者亲缘关系的重要途径,朱红大杜鹃与拟南芥之间的共线性基因为 25 对,而与毛果杨间的共线性基因多达 69 对,表明朱红大杜鹃与毛果杨的共线性和同源性更高,亲缘关系可能更近。Ka/Ks 值被认为是研究蛋白质编码基因的选择压力或强度的重要指标[34]。本研究中,朱红大杜鹃中存在共线性基因对的MADS-box基因的Ka/Ks 值均小于 1,表明这些基因在进化过程中受到纯化选择作用。
4. 结论
本研究基于课题组前期建立的朱红大杜鹃全基因组数据库,对其 MADS-box 家族进行了全基因组鉴定,并对其理化性质、系统进化、基因结构、保守结构域、顺式作用元件以及种内与种间共线性等进行了分析,研究结果将有助于理解MADS-box家族在朱红大杜鹃花分化过程中的作用,并为其他植物的相关基因家族研究提供参考。
1 1~2)如需查阅附件内容请登录《植物科学学报》网站(http://www.plantscience.cn)查看本期文章。2 3 4 1~4)如需查阅附件内容请登录《植物科学学报》网站(http://www.plantscience.cn)查看本期文章。5 1~2)如需查阅附件内容请登录《植物科学学报》网站(http://www.plantscience.cn)查看本期文章。6 -
-
[1] Liu DT,Sun WB,Ma YP,Fang ZD. Rediscovery and conservation of the critically endangered Rhododendron griersonianum in Yunnan,China[J]. Oryx,2019,53(1):14. doi: 10.1017/S0030605318001278
[2] Ma H,Liu YB,Liu DT,Sun WB,Liu XF,et al. Chromosome-level genome assembly and population genetic analysis of a critically endangered Rhododendron provide insights into its conservation[J]. Plant J,2021,107(5):1533−1545. doi: 10.1111/tpj.15399
[3] Gibbs D,Chamberlain D,Argent G. The Red List of Rhododendrons[M]. Richmond:Botanic Gardens Conservation International,2011:51.
[4] 覃海宁,杨永,董仕勇,何强,贾渝,等. 中国高等植物受威胁物种名录[J]. 生物多样性,2017,25(7):696−744. doi: 10.17520/biods.2017144 Qin HN,Yang Y,Dong SY,He Q,Jia Y,et al. Threatened species list of China’s higher plants[J]. Biodiversity Science,2017,25(7):696−744. doi: 10.17520/biods.2017144
[5] De Folter S,Angenent GC. Trans meets cis in MADS science[J]. Trends Plant Sci,2006,11(5):224−231. doi: 10.1016/j.tplants.2006.03.008
[6] Nam J,dePamphilis CW,Ma H,Nei M. Antiquity and evolution of the MADS-box gene family controlling flower development in plants[J]. Mol Biol Evol,2003,20(9):1435−1447. doi: 10.1093/molbev/msg152
[7] Theissen G,Becker A,Di Rosa A,Kanno A,Kim JT,et al. A short history of MADS-box genes in plants[J]. Plant Mol Biol,2000,42(1):115−149. doi: 10.1023/A:1006332105728
[8] Shan HY,Zhang N,Liu CJ,Xu GX,Zhang J,et al. Patterns of gene duplication and functional diversification during the evolution of the AP1/SQUA subfamily of plant MADS-box genes[J]. Mol Phylogenet Evol,2007,44(1):26−41. doi: 10.1016/j.ympev.2007.02.016
[9] Nam J,Kim J,Lee S,An G,Ma H,Nei M. TypeⅠMADS-box genes have experienced faster birth-and-death evolution than TypeⅡMADS-box genes in angiosperms[J]. Proc Natl Acad Sci USA,2004,101(7):1910−1915. doi: 10.1073/pnas.0308430100
[10] Alvarez-Buylla ER,Pelaz S,Liljegren SJ,Gold SE,Burgeff C,et al. An ancestral MADS-box gene duplication occurred before the divergence of plants and animals[J]. Proc Natl Acad Sci USA,2000,97(10):5328−5333. doi: 10.1073/pnas.97.10.5328
[11] Par̆enicová L,de Folter S,Kieffer M,Horner DS,Favalli C,et al. Molecular and phylogenetic analyses of the complete MADS-Box transcription factor family in Arabidopsis:new openings to the MADS world[J]. Plant Cell,2003,15(7):1538−1551. doi: 10.1105/tpc.011544
[12] Qin Y,Zhu GP,Li FD,Wang L,Chen C,Zhao H. MIKC-Type MADS-box gene family discovery and evolutionary investigation in Rosaceae plants[J]. Agronomy,2023,13(7):1695. doi: 10.3390/agronomy13071695
[13] Henschel K,Kofuji R,Hasebe M,Saedler H,Münster T,Theißen G. Two ancient classes of MIKC-Type MADS-box genes are present in the moss Physcomitrella patens[J]. Mol Biol Evol,2002,19(6):801−814. doi: 10.1093/oxfordjournals.molbev.a004137
[14] Kwantes M,Liebsch D,Verelst W. How MIKC* MADS-box genes originated and evidence for their conserved function throughout the evolution of vascular plant gametophytes[J]. Mol Biol Evol,2012,29(1):293−302. doi: 10.1093/molbev/msr200
[15] Kofuji R,Sumikawa N,Yamasaki M,Kondo K,Ueda,K,et al. Evolution and divergence of the MADS-box gene family based on genome-wide expression analyses[J]. Mol Biol Evol,2003,20(12):1963−1977. doi: 10.1093/molbev/msg216
[16] Masiero S,Colombo L,Grini PE,Schnittger A,Kater MM. The emerging importance of TypeⅠMADS box transcription factors for plant reproduction[J]. Plant Cell,2011,23(3):865−872. doi: 10.1105/tpc.110.081737
[17] Alvarez-Buylla ER,Liljegren SJ,Pelaz S,Gold SE,Burgeff C,et al. MADS-box gene evolution beyond flowers:expression in pollen,endosperm,guard cells,roots and trichomes[J]. Plant Journal,2000,24(4):457−466.
[18] Chanderbali AS,Berger BA,Howarth DG,Soltis DE,Soltis PS. Evolution of floral diversity:genomics,genes and gamma[J]. Philos Trans R Soc B Biol Sci,2017,372(1713):20150509. doi: 10.1098/rstb.2015.0509
[19] Grimplet J,Martínez-Zapater JM,Carmona MJ. Structural and functional annotation of the MADS-box transcription factor family in grapevine[J]. BMC Genomics,2016,17(1):80. doi: 10.1186/s12864-016-2398-7
[20] Koo SC,Bracko O,Park MS,Schwab R,Chun HJ,et al. Control of lateral organ development and flowering time by the Arabidopsis thaliana MADS-box gene AGAMOUS-LIKE6[J]. Plant J,2010,62(5):807−816. doi: 10.1111/j.1365-313X.2010.04192.x
[21] Qiu YC,Köhler C,Sverlgel L. Endosperm evolution by duplicated and neofunctionalized TypeⅠMADS-box transcription factors[J]. Mol Biol Evol,2022,39(1):msab355. doi: 10.1093/molbev/msab355
[22] Schwarz-Sommer Z,Huijser P,Nacken W,Saedler H,Sommer H. Genetic control of flower development by homeotic genes in Antirrhinum majus[J]. Science,1990,250(4983):931−936. doi: 10.1126/science.250.4983.931
[23] Amasino RM,Michaels SD. The timing of flowering[J]. Plant Physiol,2010,154(2):516−520. doi: 10.1104/pp.110.161653
[24] Chen Q,Li J,Yang FJ. Genome-wide analysis of the MADS-box transcription factor family in Solanum melongena[J]. Int J Mol Sci,2023,24(1):826. doi: 10.3390/ijms24010826
[25] Arora R,Agarwal P,Ray S,Singh AK,Singh VP,et al. MADS-box gene family in rice:genome-wide identification,organization and expression profiling during reproductive development and stress[J]. BMC Genomics,2007,8(1):242. doi: 10.1186/1471-2164-8-242
[26] Wei B,Zhang RZ,Guo JJ,Liu DM,Li AL,et al. Genome-wide analysis of the MADS-box gene family in Brachypodium distachyon[J]. PLoS One,2014,9(1):e84781. doi: 10.1371/journal.pone.0084781
[27] Leseberg CH,Li AL,Kang H,Duvall M,Mao L. Genome-wide analysis of the MADS-box gene family in Populus trichocarpa[J]. Gene,2006,378:84−94. doi: 10.1016/j.gene.2006.05.022
[28] Zhang XD,Fatima M,Zhou P,Ma Q,Ming R. Analysis of MADS-box genes revealed modified flowering gene network and diurnal expression in pineapple[J]. BMC Genomics,2020,21(1):8. doi: 10.1186/s12864-019-6421-7
[29] Ruelens P,Zhang ZC,van Mourik H,Maere S,Kaufmann K,Geuten K. The origin of floral organ identity quartets[J]. Plant Cell,2017,29(2):229−242. doi: 10.1105/tpc.16.00366
[30] Ferrario S,Immink RGH,Shchennikova A,Busscher-Lange J,Angenent GC. The MADS box gene FBP2 is required for SEPALLATA function in petunia[J]. Plant Cell,2003,15(4):914−925. doi: 10.1105/tpc.010280
[31] 高玮林,张力曼,薛超玲,张垚,刘孟军,赵锦. 枣E类MADS基因在花和果中的表达及其蛋白互作研究[J]. 园艺学报,2022,49(4):739−748. Gao WL,Zhang LM,Xue CL,Zhang Y,Liu MJ,Zhao J. Expression of E-Type MADS-box genes in flower and fruits and protein interaction analysis in Chinese jujube[J]. Acta Horticulturae Sinica,2022,49(4):739−748.
[32] 孙福辉,方慧仪,温小蕙,张亮生. 马银花MADS-box基因家族系统进化与表达分析[J]. 植物学报,2023,58(3):404−416. Sun FH,Fang HY,Wen XH,Zhang LS. Phylogenetic and expression analysis of MADS-box gene family in Rhododendron ovatum[J]. Chinese Bulletin of Botany,2023,58(3):404−416.
[33] Chen CJ,Chen H,Zhang Y,Thomas HR,Frank MH,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant,2020,13(8):1194−1202. doi: 10.1016/j.molp.2020.06.009
[34] Zhang Z,Li J,Zhao XQ,Wang J,Wong GKS,Yu J. KaKs_calculator:calculating Ka and Ks through model selection and model averaging[J]. Genomics Proteomics Bioinformatics,2006,4(4):259−263. doi: 10.1016/S1672-0229(07)60007-2
[35] Shu YJ,Yu DS,Wang D,Guo DL,Guo CH. Genome-wide survey and expression analysis of the MADS-box gene family in soybean[J]. Mol Biol Rep,2013,40(6):3901−3911. doi: 10.1007/s11033-012-2438-6
[36] 潘志演,唐博希,田慧源,范吴蔚,彭剑涛,刘国琴. 烟草MADS-box基因家族鉴定及其腋芽发育相关基因表达分析[J]. 分子植物育种,2022,20(7):2217−2232. Pan ZY,Tang BX,Tian HY,Fan WW,Peng JT,Liu GQ. Identification of MADS-box gene family and expression analysis of genes related to axillary bud development in Nicotiana tabacum[J]. Molecular Plant Breeding,2022,20(7):2217−2232.
[37] Wang YS,Zhang JL,Hu ZL,Guo XH,Tian SB,Chen GP. Genome-wide analysis of the MADS-box transcription factor family in Solanum lycopersicum[J]. Int J Mol Sci,2019,20(12):2961. doi: 10.3390/ijms20122961
[38] Li C,Wang Y,Xu L,Nie SS,Chen YL,et al. Genome-wide characterization of the MADS-box gene family in radish (Raphanus sativus L.) and assessment of its roles in flowering and floral organogenesis[J]. Front Plant Sci,2016,7:1390.
[39] Won SY,Jung JA,Kim JS. Genome-wide analysis of the MADS-box gene family in Chrysanthemum[J]. Comput Biol Chem,2021,90:107424. doi: 10.1016/j.compbiolchem.2020.107424
[40] 高欢,郑珂昕,廖光联,王海令,陈璐,等. 中华猕猴桃全基因组MADS-box基因家族鉴定及表达分析[J]. 果树学报,2023,40(11):2307−2324. Gao H,Zheng KX,Liao GL,Wang HL,Chen L,et al. Genome-wide identification and expression analysis of the MADS-box gene family in Actinidia chinensis[J]. Journal of Fruit Science,2023,40(11):2307−2324.
[41] Wei X,Wang LH,Yu JY,Zhang YX,Li DH,Zhang XR. Genome-wide identification and analysis of the MADS-box gene family in sesame[J]. Gene,2015,569(1):66−76. doi: 10.1016/j.gene.2015.05.018
[42] 王溪唯,陈璨,王大玮. 云南栘[木衣]MADS-box基因家族鉴定与表达分析[J]. 生物工程学报,2023,39(7):2897−2913. Wang XW,Chen C,Wang DW. Identification and expression analysis of MADS-box gene family in Docynia delavayi (Franch.) Schneid[J]. Chinese Journal of Biotechnology,2023,39(7):2897−2913.
[43] Li D,Liu C,Shen LS,Wu Y,Chen HY,et al. A repressor complex governs the integration of flowering signals in Arabidopsis[J]. Dev Cell,2008,15(1):110−120. doi: 10.1016/j.devcel.2008.05.002
[44] Yang YF,Zhu T,Niu DK. Association of intron loss with high mutation rate in Arabidopsis:implications for genome size evolution[J]. Genome Biol Evol,2013,5(4):723−733. doi: 10.1093/gbe/evt043
[45] Wu JY,Xiao JF,Wang LP,Zhong J,Yin HY,et al. Systematic analysis of intron size and abundance parameters in diverse lineages[J]. Sci China Life Sci,2013,56(10):968−974. doi: 10.1007/s11427-013-4540-y
[46] Gao HH,Wang ZM,Li SL,Hou ML,Zhou Y,et al. Genome-wide survey of potato MADS-box genes reveals that StMADS1 and StMADS13 are putative downstream targets of tuberigen StSP6A[J]. BMC Genomics,2018,19(1):726. doi: 10.1186/s12864-018-5113-z
[47] Airoldi CA,Davies B. Gene duplication and the evolution of plant MADS-box transcription factors[J]. J Genet Genomics,2012,39(4):157−165. doi: 10.1016/j.jgg.2012.02.008
[48] Zhou PY,Qu YS,Wang ZW,Huang B,Wen Q,et al. Gene structural specificity and expression of MADS-box gene family in Camellia chekiangoleosa[J]. Int J Mol Sci,2023,24(4):3434. doi: 10.3390/ijms24043434
-
其他相关附件
-
PDF格式
柳文-附件 点击下载(1390KB)
-



 下载:
下载: