Comparative chloroplast genomic and phylogenetic analysis of Aralia and related species
-
摘要:
分别对楤木属(Aralia)和羽叶参属(Pentapanax)各两个物种进行叶绿体基因组测序、组装和注释,并进行结构分析;结合NCBI下载的近缘类群叶绿体组序列,进行系统发育分析。结果显示,4个物种的叶绿体基因组均为环状四分体结构,长度为155 744 ~ 156 201 bp,GC含量为38.1%,均包含132个基因,其中蛋白质编码基因87个,rRNA基因8个,tRNA基因37个。边界分析发现IR(Inverted repeat)区均未发生收缩和扩张。SSR(Simple sequence repeat)序列数量在39 ~ 43个,多为单核苷酸和二核苷酸重复,位置多在非编码区。序列差异多出现在LSC(Large single copy)和SSC(Small single copy)区的非编码区。最大似然树揭示出两个高度支持的单系分支:第1分支包括羽叶参属、浓紫龙眼独活(Aralia atropurpurea Franch.)、食用土当归(Aralia cordata Thunb.)和东北土当归(Aralia continentalis Kitagawa);第2分支则均由楤木属物种组成。总体来说,楤木属和羽叶参属植物的叶绿体基因组序列比较保守;系统发育分析结果支持羽叶参属置入楤木属内部以及楤木属的各个种成单系。
Abstract:The chloroplast genomes of two species of Aralia and Pentapanax were sequenced, assembled, annotated, and structurally analyzed. Furthermore, combined with chloroplast genome sequences of related species downloaded from the NCBI database, phylogenetic analysis was conducted. The chloroplast genomes of the four species all exhibited a quadripartite circular structure with a length of 155 744 – 156 201 bp and GC content of 38.1%. Both genera contained 132 genes, including 87 protein-coding genes, eight ribosomal RNA (rRNA) genes, and 37 transfer RNA (tRNA) genes. Boundary analysis found that neither contraction nor expansion occurred in the inverted repeat regions. The number of simple sequence repeat (SSR) loci ranged from 39 to 43, most of which were mononucleotide and dinucleotide repeats, and mostly located in non-coding regions. Sequence differences mainly occurred in the non-coding regions of the large single copy (LSC) and small single copy (SSC) regions. The maximum-likelihood tree revealed two highly supported monophyletic branches: the first composed of Pentapanax, Aralia atropurpurea Franch., Aralia cordata Thunb., and Aralia continentalis Kitagawa and the second composed of other Aralia species. In general, the chloroplast genome sequences of Aralia and Pentapanax plants were relatively conserved. The phylogenetic results supported the placement of Pentapanax into the genus Aralia, with each Aralia species forming a monophyly.
-
Keywords:
- Aralia /
- Pentapanax /
- Chloroplast genome /
- Phylogeny
-
楤木属(Aralia)隶属于五加科楤木族,全世界约40种,我国有29种,是楤木属植物资源最为丰富的国家[1],其中20种可供药用,主要以根、根皮入药,主要药用成分为三萜皂苷类、黄酮类和有机酸等[2],可用于肝炎、胃痛、风湿关节痛和跌打损伤等多种疾病的治疗。黄毛楤木(Aralia chinensis L. )、食用土当归(Aralia cordata Thunb. )、辽东楤木(Aralia elata var. glabrescens (Franchet & Savatier) Pojarkova)等还具有抗肿瘤作用[3]。羽叶参属(Pentapanax)为楤木属同族植物,大约有18 ~ 22个种,中国有16种,主要分布在中国-喜马拉雅地区[4]。
楤木属和羽叶参属在分类上长期存在分歧。李湘萍等[5]根据花瓣、花梗等特征认为楤木属和羽叶参属关系十分密切,不排除共同起源的可能。向其柏[6]根据刺、托叶、苞片等形态特征将圆叶楤木(Aralia caesia Hand. -Mazz.)、西南楤木(Aralia wilsonii Harms)和羽叶楤木(Aralia plumosa Li)划归为羽叶参属中。Wen等[7]利用ITS序列对五加科进行系统发育分析,结果表明羽叶参属和楤木属聚为一个单系成为楤木属-羽叶参属复合群,但树的支持率较低。基于ITS序列,Wen[8]提出将整个羽叶参属置入楤木属内部作为羽叶参组(Aralia sect. Pentapanax),并在后续研究中利用ITS序列和叶绿体基因组片段等分子标记进行了验证[9, 10],但由于分子标记分辨率不足等原因,该结论并没有得到很好的支持。综上,基于形态性状和分子系统学对楤木属及其近缘类群开展的分类研究已有一些报道,但这些形态特征、分子标记的分辨率较低,导致楤木属及其近缘类群的界限仍然不明晰。
叶绿体基因组作为植物系统发育研究的有效分子标记,通常具有双链、环状结构等特征。完整的叶绿体基因组由大单拷贝区、小单拷贝区和两个反向重复区组成,被子植物基因组大小通常在115 ~ 165 Kb,包含110 ~ 130个基因[11]。叶绿体基因组在大多数植物中表现出母系遗传,重组较少,进化速度较慢,这与核基因有很大不同,这些特征使其在组成和结构上高度保守[12]。总之,基于叶绿体基因组构建属、种间的关系已被广泛应用于被子植物的系统发育研究中[13]。
本文报道了羽叶参属物种和棘茎楤木(Aralia echinocaulis Hand. -Mazz.)的叶绿体基因组,并结合NCBI发布的其他楤木属植物叶绿体基因组序列,开展比较基因组和系统发育分析,以揭示两属物种叶绿体基因组的基本特征和系统关系。本研究从基因组水平探究楤木属与其近缘类群羽叶参属间的亲缘关系,研究结果旨在为楤木属植物药用资源的分子鉴定、保护和开发奠定基础。
1. 材料与方法
1.1 实验材料
楤木属的两个物种(共3个个体)和羽叶参属的两个物种(共4个个体),分别采自河南南阳、云南维西、云南富民县和云南香格里拉(表1,原始测序数据已上传至SRA数据库,编号为SRR20849639 ~ SRR20849645)。新鲜叶片材料采集后迅速放置于硅胶中干燥保存,凭证标本存放于河南农业大学标本馆。7个楤木属和1个马蹄参(Diplopanax stachyanthus Hand. -Mazz.)叶绿体基因组序列来源于NCBI。
表 1 楤木属和羽叶参属物种采样信息Table 1. Sampling information of Aralia and Pentapanax species编号
No.中文名
Chinese name拉丁名
Latin name采集地
Collection places纬度
Latitude经度
LongitudeGenBank 登录号
GenBank No.FM1 毛梗寄生羽叶参 Pentapanax parasiticus var. khasianus
C. B. Clarke云南富民县 25°15'25"N 102°26'13"E ON493678 FM4 毛梗寄生羽叶参 P. parasiticus var. khasianus C. B. Clarke 云南富民县 25°15'25"N 102°26'13"E ON493679 NY1 楤木 Aralia elata (Miq.) Seem. 河南南阳 33°37'48"N 111°41'13"E ON493673 WX1 棘茎楤木 A. echinocaulis Hand. -Mazz. 云南维西 39°54'39"N 116°24'48"E ON493674 WX6 棘茎楤木 A. echinocaulis Hand. -Mazz. 云南维西 39°54'39"N 116°24'48"E ON493675 XG5 圆叶羽叶参 P. caesius (Handel-Mazzetti) C. B. Shang 云南香格里拉 27°50'87"N 99°42'26"E ON493676 XG15 圆叶羽叶参 P. caesius (Handel-Mazzetti) C. B. Shang 云南香格里拉 27°50'87"N 99°42'26"E ON493677 1.2 基因组DNA提取与测序
使用改良的CTAB方法提取基因组总DNA,为了去除植物叶片中的多糖、多酚等成分,使用Plant DNAZol快速提取试剂(Invitrogen, 美国)代替传统的CTAB裂解液进行DNA提取。将提取的高质量DNA送往苏州金唯智科技有限公司,采用Illumina Hiseq2500测序平台进行文库构建和测序。
1.3 叶绿体基因组的组装和注释
将测序的原始数据利用FASTQC v0.11.3软件[14]进行质控,去掉低质量区域和冗余片段。完整的叶绿体基因组通过GetOrganelle v1.7.5.3软件[15]进行组装,以楤木的叶绿体基因组序列(KT153023.1)作为参考序列,通过在线工具PGA[16]对基因组进行注释,最后使用Geneious v11.1.4软件进行手动修正注释。叶绿体基因组图谱通过在线软件OGDraw[17]进行绘制。
1.4 数据统计和比较叶绿体组分析
使用Geneious v11.1.4软件统计叶绿体基因组的基因信息和GC含量等基本特征。通过在线工具IRscope[18]生成基因边界分布图。此外,利用MISA v2.1软件[19]鉴定叶绿体基因组中的SSR位点,分别将单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸以及六核苷酸重复的最小重复数设为10、5、4、3、3、3。以楤木为参考序列,使用在线工具mVISTA[20]选择Shuffle-LAGAN模式将7条叶绿体基因组序列进行可视化差异分析。使用DNAsp v6.12.03软件[21]将窗口大小设置为600,步长设置为200,进行叶绿体基因组片段多态性分析。
1.5 系统发育树构建
基于去除一个IR区的15个叶绿体基因组序列,以马蹄参叶绿体组序列(NC_029750)作为外类群推断楤木属及其近缘类群之间的系统发育关系。首先使用Mafft v7.310软件[22]进行序列比对,然后基于ModelFinder v1.6.8软件[23]筛选出核苷酸最佳替代模型,通过Phylosuite v1.2.2软件[24]中的IQ-Tree将Bootstrap设置为1000,基于最大似然法构建系统发育树。
2. 结果与分析
2.1 叶绿体基因组的基本特征及分类
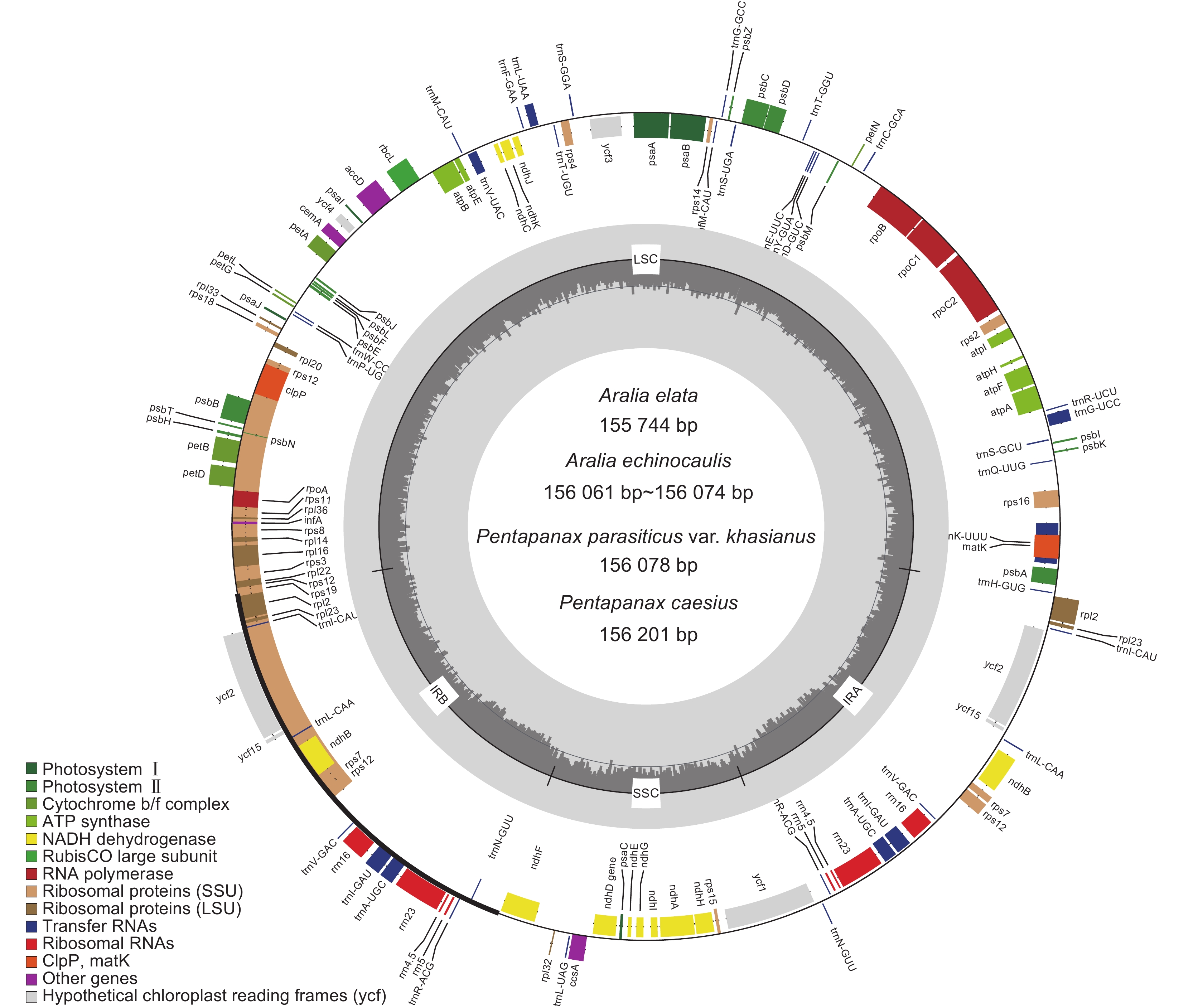
楤木属和羽叶参属的叶绿体基因组长度为155 744 ~ 156 201 bp(图1),呈典型的环状四分体结构,由1个大单拷贝区(LSC,85 799 ~ 86 295 bp)、1个小单拷贝区(SSC,18 055 ~ 18 121 bp)和2个反向重复区(IR,25 923 ~ 25 941 bp)组成,GC含量均为38.1%(表2)。7个个体通过注释均得到132个基因,其中蛋白质编码基因(CDS)87个,rRNA 8个,tRNA 37个,包含1个内含子的基因为15个,包含2个内含子的基因为3个(表3)。rps12为反式剪接基因,第1个外显子位于LSC区,第2、3个外显子均位于IR区。
表 2 7个叶绿体基因组的基本特征Table 2. Basic characteristics of seven chloroplast genomes编号
No.长度Length / bp GC含量
GC / %基因数量 Gene number 基因组大小
Genome size大单拷贝区
LSC小单拷贝区
SSC反向重复区
IRrRNA tRNA CDS 总基因数
TotalFM1 156 078 86 171 18 055 25 926 38.10 37 8 87 132 FM4 156 078 86 171 18 055 25 926 38.10 37 8 87 132 NY1 155 744 85 799 18 099 25 923 38.10 37 8 87 132 WX1 156 074 86 071 18 121 25 941 38.10 37 8 87 132 WX5 156 061 86 058 18 121 25 941 38.10 37 8 87 132 XG5 156 201 86 295 18 060 25 923 38.10 37 8 87 132 XG15 156 201 86 295 18 060 25 923 38.10 37 8 87 132 表 3 7个叶绿体基因组的基因组成Table 3. Gene composition of seven chloroplast genomes基因分组 Gene group 基因名称 Gene name 核糖体大亚基 rpl2a ( × 2)、rpl14、rpl16a、rpl20、rpl22、rpl23 ( × 2)、rpl32、rpl33、rpl36 核糖体小亚基 rps2a、rps3、rps4、rps7 ( × 2)、rps8、rps11、rps12b ( × 2)、rps14、rps15、rps16a、rps18、rps19 RNA聚合酶 rpoA、rpoB、rpoC1a、rpoC2 rRNA基因 rrn4.5 ( × 2)、rrn5 ( × 2)、rrn16 ( × 2)、rrn23 ( × 2) tRNA基因 trnA-UGCa ( × 2)、trnC-GCA、trnD-GUC、trnE-UUC、trnF-GAA、trnfM-CAU ( × 2)、trnG-GCC、trnG-UCCa、trnH-GUG、trnI-CAU ( × 2)、trnI-GAUa、trnK-UUUa、trnL-CAA ( × 2)、trnL-UAAa、trnL-UAG、trnM-CAU、trnN-GUU ( × 2)、trnP-UGG、trnQ-UUG、trnR-ACG ( × 2)、trnR-UCU、trnS-GCU、 trnS-GGA、trnS-UGA、trnT-GGU、trnT-UGU、trnV-GAC ( × 2)、trnV-UACa、trnW-CCA、trnY-GUA 光系统Ⅰ psaA、psaB、psaC、psaI、psaJ 光系统Ⅱ psbA、psbB、psbC、psbD、psbE、psbF、psbH、psbI、psbJ、psbK、psbL、psbM、psbN、psbT、psbZ NADH脱氢酶 ndhAa、ndhBa ( × 2)、ndhC、ndhE、ndhF、ndhG、ndhH、ndhI、ndhJ、ndhK 细胞色素b/f 复合物 petA、petBa、petDa、petG、petL、petN ATP合成酶 atpA、atpB、atpE、atpFa、atpH、atpI 二磷酸核酮糖羧化酶大亚基 rbcL 成熟酶 matK 转录启动因子 infA 蛋白酶 clpPb 被膜蛋白 cemA 乙酰辅酶A羧化酶 accD 细胞色素C的合成基因 ccsA 保守假定的叶绿体开放阅读框 ycf1、ycf2 ( × 2)、ycf3b、ycf4、ycf15 ( × 2) 注:“a”表示基因含有1个内含子;“b”表示基因含有2个内含子;( × 2) 表明基因在IR 区复制。 Notes: “a” indicates that gene contains one intron; “b” indicates that gene contains two introns; ( × 2) indicates that gene was replicated in IR region. 2.2 边界区比较分析
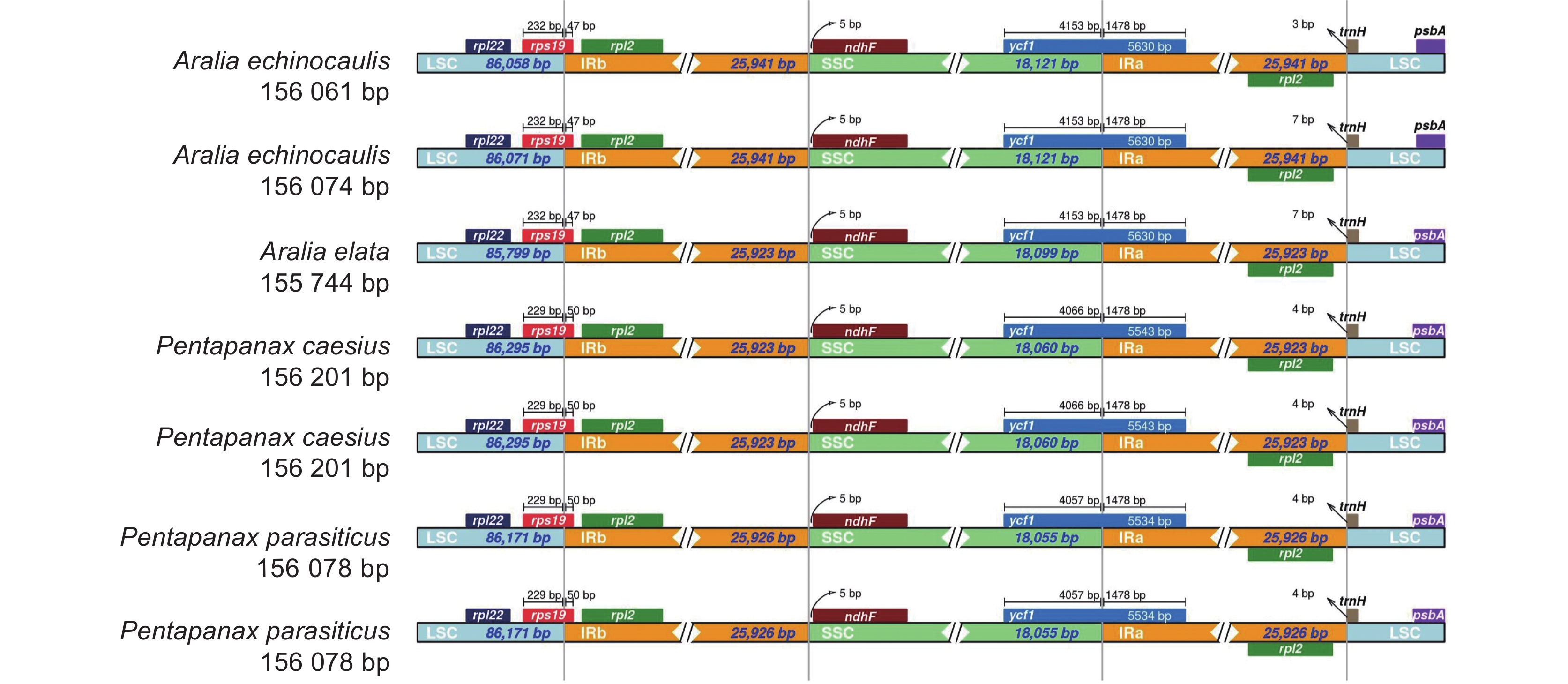
7个叶绿体基因组的边界区比较保守,均未发生收缩和扩张(图2)。rps19基因处于LSC/IRb的边界,有229 ~ 232 bp位于LSC区,47 ~ 50 bp位于IRa区。ndhF基因位于SSC区,距离IRb/SSC边界有5 bp。ycf1基因位于SSC/IRa边界,有4057 ~ 4153 bp位于SSC区,1478 bp位于IRa区。rpl2基因和trnH基因分别位于IRa/LSC边界两侧。
2.3 简单重复序列(SSR)分析
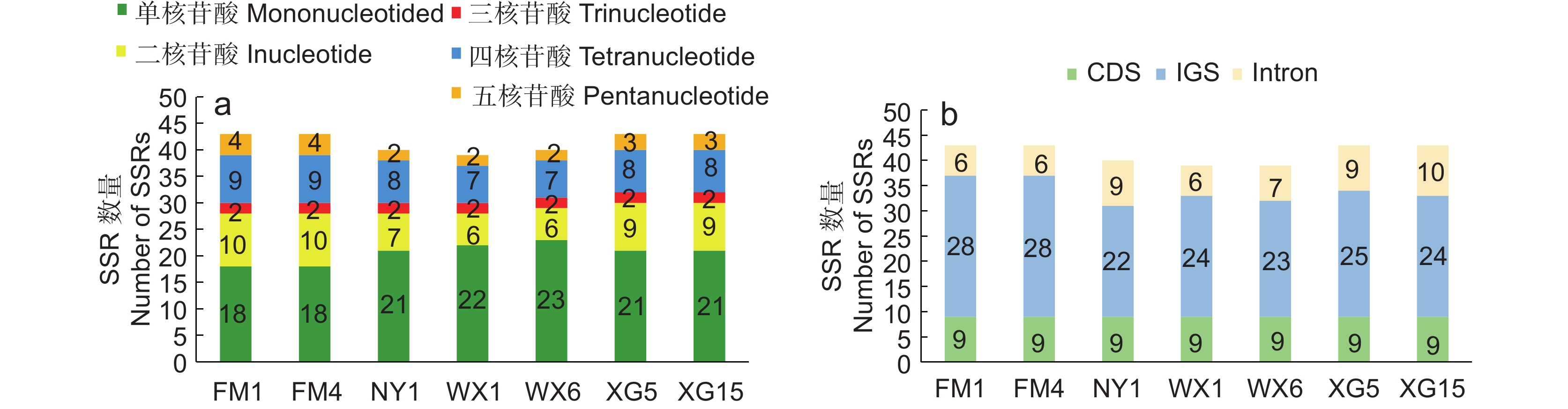
在7个个体的叶绿体基因组中,单核苷酸和二核苷酸重复的数量要多于其他核苷酸重复,且均未发现六核苷酸重复。鉴定到的SSR为39 ~ 43个,其中单核苷酸重复在18 ~ 23条,二核苷酸重复在6 ~ 10条,三核苷酸重复均为2条,四核苷酸重复在7 ~ 9条,五核苷酸重复为2 ~ 4条。7个个体的SSR序列多分布于IGS(Intergenic spacer)区,其次是CDS(Protein-coding sequences)区,分布在Intron区的最少(图3)。
2.4 基因组对比及核苷酸多样性分析
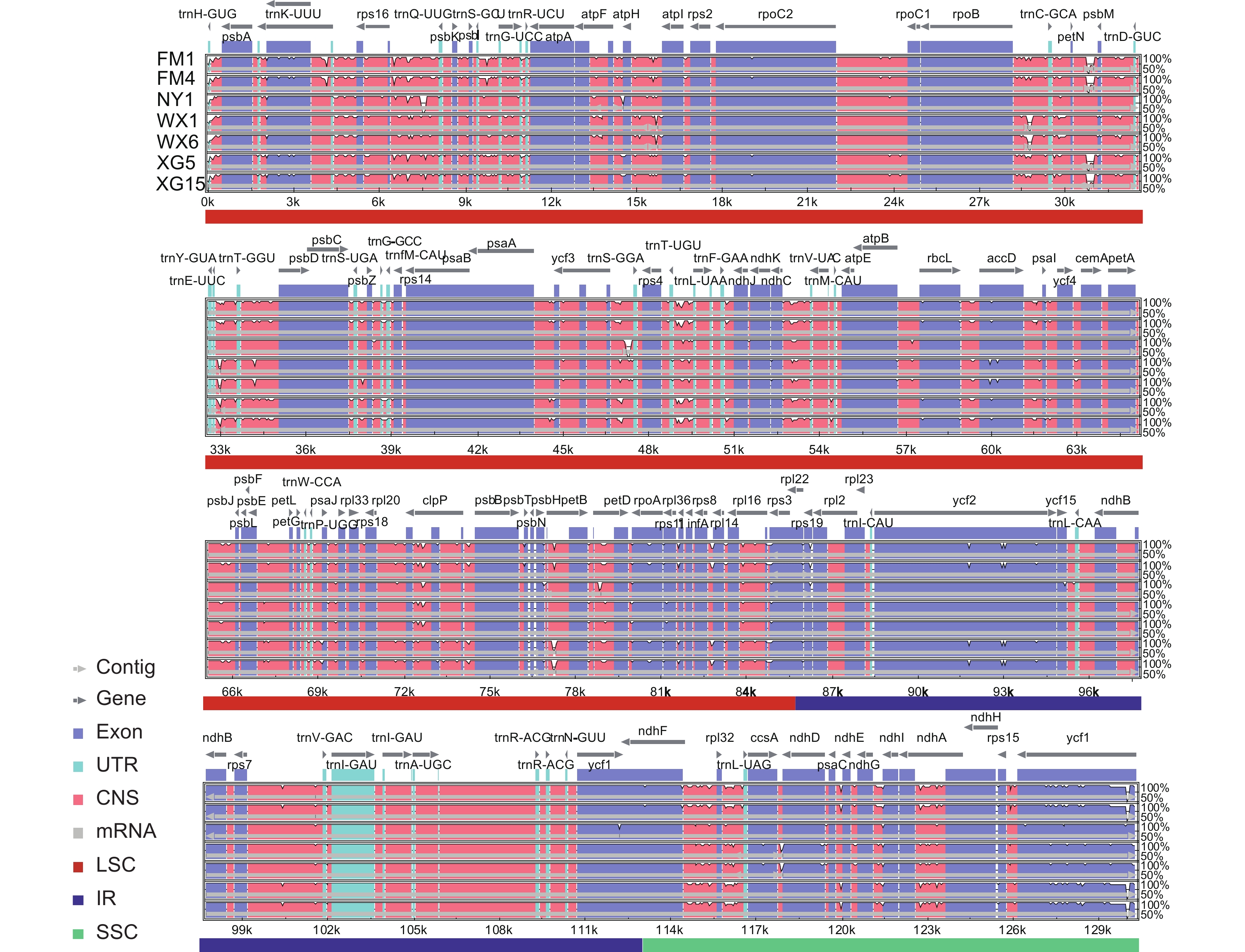
根据序列整体特征,去掉1个IR区的7个叶绿体基因组总体表现出保守状态,但在部分区域,LSC区和SSC区的变异程度要高于IR区,且非编码序列变异程度高于编码序列。个体间的变异主要体现在rps16-trnQ-UGU、petN-psbM、ycf3-trnS-GGA、petB、ycf1等基因区域(图4)。
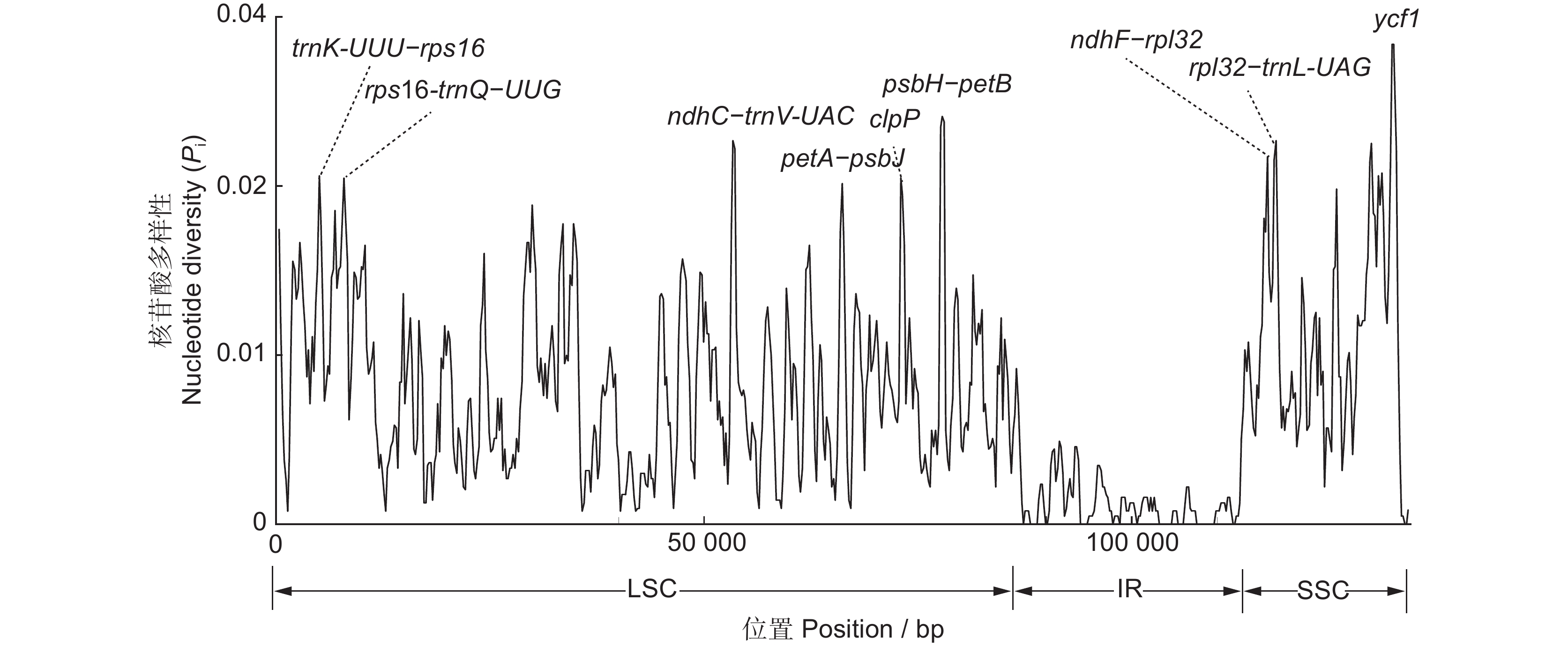
根据核苷酸多样性分析,7个基因组的Pi(Nucleotide diversity)值在0 ~ 0.0284,差异多出现在LSC和SSC区,而IR区相对保守,trnK-UUU-rps16、rps16-trnQ-UUG、ndhC-trnV-UAC、petA-psbJ、clpP、psbH-petB、dnhF-rpl32、rpl32-trnL-UAG、ycf1等片段Pi值大于0.02,核苷酸变异程度较高,且多位于基因间隔区(图5)。
2.5 系统发育分析
基于去掉一个IR区的15个叶绿体基因组序列和最佳核苷酸代替模型(K3Pu + F + R2)所构建的最大似然树,主要包括两个高度支持的单系分支(支持率均为100%),第1分支由羽叶参属的两个物种、楤木属的浓紫龙眼独活、食用土当归和东北土当归组成,第2分支由楤木(Aralia elata (Miq.) Seem.)和棘茎楤木(Aralia echinocaulis Hand. -Mazz.)组成(图6)。
3. 讨论
大多数被子植物叶绿体基因组的结构为环状四分体结构[11],本研究中楤木属和羽叶参属4个物种的叶绿体基因组均包括1个LSC区、1个SSC区和两个IR区,基因组大小在155 744 ~ 156 201 bp,GC含量均为38.1%,且均注释得到132个基因,与被子植物叶绿体基因组特征基本吻合,且与之前报道过的楤木属叶绿体基因组特征相似[25]。值得注意的是,rps12是一个反式剪接基因,其第1个外显子在LSC区,第2和第3个外显子均在IR区,与荚蒾属(Viburnum)、接骨木属(Sambucus)[26]、藜芦属(Veratrum)[27]、高良姜(Alpinia officinarum Hance)[28]和楤木属的浓紫龙眼独活[29]、食用土当归[30]等物种一致。4个rRNA基因(rrn4.5、rrn5、rrn16、rrn23)比较保守,因都处于IR区,所以每个rRNA都发生了复制。7个叶绿体基因组的整体结构比较稳定,没有发生大的变化,具有较高保守性。
IR区的收缩和扩张在叶绿体基因组中十分常见,这是导致其基因数量和长度变化的主要因素[31]。在本研究中,rps19基因均处于LSC/IRb边界处;ycf1基因处在SSC/IRa边界上,rpl2和trnH基因均位于IRa/LSC两侧,IR区边界有较高保守性,没有发生明显的收缩和扩张,这与人参属(Panax)[32]和五加科其他植物[33]的研究结果相似。通过mVISTA全基因组比对分析和核苷酸多态性分析发现,一些基因如rps16-trnQ-UGU、petN-psbM、ycf3-trnS-GGA、psbH-petB、ycf1等在个体间变异较大,这些差异多发生在LSC和SSC的基因间隔区,类似现象在五加科的波缘楤木(Aralia undulata Hand. -Mazz.)、浅裂罗伞(Brassaiopsis hainla (Buch. -Ham.) Seem.)、刺五加(Eleutherococcus senticosus (Ruprecht & Maximowicz) Maximowicz)、刺楸(Kalopanax septemlobus (Thunb.) Koidz.)、梁王茶(Metapanax delavayi (Franchet) J. Wen & Frodin)、穗序鹅掌柴(Schefflera delavayi (Franch.) Harms ex Diels)等植物中也有发现[34]。这些片段或许可为整个五加科植物的分子鉴定提供参考。在质体基因组中,ycf1基因通常处于SSC/IRa边界上[35],有研究表明其变异程度和分辨率比常用作条形码的matK片段更高[35]。在本研究中,ycf1基因也处于SSC/IRa边界上,且具有较大程度的变异,根据核苷酸多态性分析结果,此片段在7个个体中变异程度最高,在后续的楤木属物种鉴定中可考虑将其作为重要条形码之一。
SSR标记具有操作简单、成本较低、分辨率高等特点,被广泛应用于物种的分类和鉴定等研究中[36]。本研究结果表明,SSR序列中单核苷酸重复数量多于其他核苷酸重复数量,且多分布于IGS区,这与藜芦属[27]、暴马丁香(Syringa reticulata subsp. amurensis (Rupr.) P. S. Green & M. C. Chang)[37]和乳苣(Mulgedium tataricum (L.) DC.)等植物的研究结果相似[38]。这些SSR位点对今后楤木属和羽叶参属植物的遗传多样性、群体结构及分子鉴定等研究具有一定的参考价值。
叶绿体基因组作为研究系统发育的重要工具,其优点在于含有大量的遗传信息、检测方法简便、结构和含量比较保守等[39]。本研究利用7个测序材料和NCBI发布的8个楤木属叶绿体基因组序列,基于最大似然法构建系统发育树,结果高度支持将羽叶参属置入楤木属内成为羽叶参组,这与Wen[8]的研究结果一致,且在两属内部,各个种的单系地位都得到了强烈支持。此外,在楤木属内部,草本类群(浓紫龙眼独活、食用土当归和东北土当归)和木本类群(楤木和棘茎楤木)各自聚为两个单系。李湘萍和向其柏[5]认为楤木属草本物种与木本物种在形态上存在着明显差别,有必要建立两个亚属,即木本亚属楤木亚属(Aralia subgen. Aralia)和草本亚属土当归亚属(Aralia subgen. Paralia)。许旭东等[40]根据是否含有二萜皂甙也证实了上述两个亚属的分化。本研究同样支持楤木属草本亚属和木本亚属两亚属的划分,且从系统发育分析结果来看,羽叶参属似乎与楤木属中的草本类群关系更近。叶绿体基因组在解析楤木属及其近缘类群间以及楤木属下物种间的系统发育关系方面具有强大效力,但本研究涉及的相关类群太少,未来应基于此技术,加大类群覆盖面,全面重建楤木属及其近缘类群的系统演化关系,以期为将来的资源开发利用奠定坚实的理论基础。
-
表 1 楤木属和羽叶参属物种采样信息
Table 1 Sampling information of Aralia and Pentapanax species
编号
No.中文名
Chinese name拉丁名
Latin name采集地
Collection places纬度
Latitude经度
LongitudeGenBank 登录号
GenBank No.FM1 毛梗寄生羽叶参 Pentapanax parasiticus var. khasianus
C. B. Clarke云南富民县 25°15'25"N 102°26'13"E ON493678 FM4 毛梗寄生羽叶参 P. parasiticus var. khasianus C. B. Clarke 云南富民县 25°15'25"N 102°26'13"E ON493679 NY1 楤木 Aralia elata (Miq.) Seem. 河南南阳 33°37'48"N 111°41'13"E ON493673 WX1 棘茎楤木 A. echinocaulis Hand. -Mazz. 云南维西 39°54'39"N 116°24'48"E ON493674 WX6 棘茎楤木 A. echinocaulis Hand. -Mazz. 云南维西 39°54'39"N 116°24'48"E ON493675 XG5 圆叶羽叶参 P. caesius (Handel-Mazzetti) C. B. Shang 云南香格里拉 27°50'87"N 99°42'26"E ON493676 XG15 圆叶羽叶参 P. caesius (Handel-Mazzetti) C. B. Shang 云南香格里拉 27°50'87"N 99°42'26"E ON493677 表 2 7个叶绿体基因组的基本特征
Table 2 Basic characteristics of seven chloroplast genomes
编号
No.长度Length / bp GC含量
GC / %基因数量 Gene number 基因组大小
Genome size大单拷贝区
LSC小单拷贝区
SSC反向重复区
IRrRNA tRNA CDS 总基因数
TotalFM1 156 078 86 171 18 055 25 926 38.10 37 8 87 132 FM4 156 078 86 171 18 055 25 926 38.10 37 8 87 132 NY1 155 744 85 799 18 099 25 923 38.10 37 8 87 132 WX1 156 074 86 071 18 121 25 941 38.10 37 8 87 132 WX5 156 061 86 058 18 121 25 941 38.10 37 8 87 132 XG5 156 201 86 295 18 060 25 923 38.10 37 8 87 132 XG15 156 201 86 295 18 060 25 923 38.10 37 8 87 132 表 3 7个叶绿体基因组的基因组成
Table 3 Gene composition of seven chloroplast genomes
基因分组 Gene group 基因名称 Gene name 核糖体大亚基 rpl2a ( × 2)、rpl14、rpl16a、rpl20、rpl22、rpl23 ( × 2)、rpl32、rpl33、rpl36 核糖体小亚基 rps2a、rps3、rps4、rps7 ( × 2)、rps8、rps11、rps12b ( × 2)、rps14、rps15、rps16a、rps18、rps19 RNA聚合酶 rpoA、rpoB、rpoC1a、rpoC2 rRNA基因 rrn4.5 ( × 2)、rrn5 ( × 2)、rrn16 ( × 2)、rrn23 ( × 2) tRNA基因 trnA-UGCa ( × 2)、trnC-GCA、trnD-GUC、trnE-UUC、trnF-GAA、trnfM-CAU ( × 2)、trnG-GCC、trnG-UCCa、trnH-GUG、trnI-CAU ( × 2)、trnI-GAUa、trnK-UUUa、trnL-CAA ( × 2)、trnL-UAAa、trnL-UAG、trnM-CAU、trnN-GUU ( × 2)、trnP-UGG、trnQ-UUG、trnR-ACG ( × 2)、trnR-UCU、trnS-GCU、 trnS-GGA、trnS-UGA、trnT-GGU、trnT-UGU、trnV-GAC ( × 2)、trnV-UACa、trnW-CCA、trnY-GUA 光系统Ⅰ psaA、psaB、psaC、psaI、psaJ 光系统Ⅱ psbA、psbB、psbC、psbD、psbE、psbF、psbH、psbI、psbJ、psbK、psbL、psbM、psbN、psbT、psbZ NADH脱氢酶 ndhAa、ndhBa ( × 2)、ndhC、ndhE、ndhF、ndhG、ndhH、ndhI、ndhJ、ndhK 细胞色素b/f 复合物 petA、petBa、petDa、petG、petL、petN ATP合成酶 atpA、atpB、atpE、atpFa、atpH、atpI 二磷酸核酮糖羧化酶大亚基 rbcL 成熟酶 matK 转录启动因子 infA 蛋白酶 clpPb 被膜蛋白 cemA 乙酰辅酶A羧化酶 accD 细胞色素C的合成基因 ccsA 保守假定的叶绿体开放阅读框 ycf1、ycf2 ( × 2)、ycf3b、ycf4、ycf15 ( × 2) 注:“a”表示基因含有1个内含子;“b”表示基因含有2个内含子;( × 2) 表明基因在IR 区复制。 Notes: “a” indicates that gene contains one intron; “b” indicates that gene contains two introns; ( × 2) indicates that gene was replicated in IR region. -
[1] 程莹,李根有,夏国华,黄晌决,黄宇锋. 楤木属植物组织培养研究综述[J]. 浙江农林大学学报,2011,28(6):968−972. Cheng Y,Li GY,Xia GH,Huang SJ,Huang YF. Review on tissue culture of Aralia plants[J]. Journal of Zhejiang A & F University,2011,28 (6):968−972.
[2] 郑玲玲,裴凌鹏. 楤木属植物研究进展[J]. 中国民族医药杂志,2010,16(6):57−59. [3] 张广美,王春梅. 楤木属植物及其活性成分药理学研究进展[J]. 中华中医药学刊,2011,29(8):1715−1717. Zhang GM,Wang CM. Review of pharmacotogical effects of plants of Araliaceae and bioactive compounds[J]. Chinese Archives of Traditional Chinese Medicine,2011,29 (8):1715−1717.
[4] Wu ZY, Raven PH, Hong DY. Flora of China: Vol.13[M]. Beijing: Science Press & St. Louis: Missouri Botanical Garden Press, 2007: 480−489.
[5] 李湘萍,向其柏. 中国楤木属的研究[J]. 南京林业大学学报,1992,16(2):17−24. Li XP,Xiang QB. Studies on the genus Aralia Linn.[J]. Journal of Nanjing Forestry University,1992,16 (2):17−24.
[6] 向其柏. 五加科植物的新分类群及某些修订[J]. 南京林学院学报,1985,28(2):15−28. Xiang QB. New taxa and some revisions about the Araliaceae of China[J]. Journal of Nanjing Institute of Forestry,1985,28 (2):15−28.
[7] Wen J,Plunkett GM,Mitchell AD,Wagstaff SJ. The evolution of Araliaceae:a phylogenetic analysis based on ITS sequences of nuclear ribosomal DNA[J]. Syst Bot,2001,26 (1):144−167.
[8] Wen J. Revision of Aralia Sect. Pentapanax (Seem. ) J. Wen (Araliaceae)[M]. Fuzhou: Cathaya, 2002: 1−116.
[9] Wen J. Systematics and Biogeography of Aralia L. (Araliaceae): Revision of Aralia Sects. Aralia, Humiles, Nanae, and Sciadodendron[M]. Washington D. C: Contributions from the United States National Herbarium, 2011: 1−172.
[10] Li R,Wen J. Phylogeny and diversification of Chinese Araliaceae based on nuclear and plastid DNA sequence data[J]. J Syst Evol,2016,54 (4):453−467. doi: 10.1111/jse.12196
[11] Jansen RK,Raubeson LA,Boore JL,Depamphilis CW,Chumley TW,et al. Methods for obtaining and analyzing whole chloroplast genome sequences[J]. Methods Enzymol,2005,395:348−384.
[12] Alzahrani DA. Complete chloroplast genome of Abutilon fruticosum:genome structure,comparative and phylogenetic analysis[J]. Plants,2021,10 (2):270. doi: 10.3390/plants10020270
[13] 樊守金,郭秀秀. 植物叶绿体基因组研究及应用进展[J]. 山东师范大学学报(自然科学版),2022,37(1):22−31. Fan SJ,Guo XX. Advances in research and application of plant chloroplast genome[J]. Journal of Shandong Normal University (Natural Science)
,2022,37 (1):22−31. [14] Brown J,Pirrung M,McCue LA. FQC dashboard:integrates FastQC results into a web-based,interactive,and extensible FASTQ quality control tool[J]. Bioinformatics,2017,33 (19):3137−3139. doi: 10.1093/bioinformatics/btx373
[15] Jin JJ,Yu WB,Yang JB,Song Y,de Pamphilis CW,et al. GetOrganelle:a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome Biol,2020,21 (1):241. doi: 10.1186/s13059-020-02154-5
[16] Qu XJ,Moore MJ,Li DZ,Yi TS. PGA:a software package for rapid,accurate,and flexible batch annotation of plastomes[J]. Plant Methods,2019,15 (1):50. doi: 10.1186/s13007-019-0435-7
[17] Greiner S,Lehwark P,Bock R. OrganellarGenomeDRAW (OGDRAW) version 1.3.1:expanded toolkit for the graphical visualization of organellar genomes[J]. Nucleic Acids Res,2019,47 (W1):W59−W64. doi: 10.1093/nar/gkz238
[18] Amiryousefi A,Hyvönen J,Poczai P. IRscope:an online program to visualize the junction sites of chloroplast genomes[J]. Bioinformatics,2018,34 (17):3030−3031. doi: 10.1093/bioinformatics/bty220
[19] Beier S,Thiel T,Münch T,Scholz U,Mascher M. MISA-web:a web server for microsatellite prediction[J]. Bioinformatics,2017,33 (16):2583−2585. doi: 10.1093/bioinformatics/btx198
[20] Frazer KA,Pachter L,Poliakov A,Rubin EM,Dubchak I. VISTA:computational tools for comparative genomics[J]. Nucleic Acids Res,2004,32 (S2):W273−W279.
[21] Librado P,Rozas J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,2009,25 (11):1451−1452. doi: 10.1093/bioinformatics/btp187
[22] Katoh K,Misawa K,Kuma KI,Miyata T. MAFFT:a novel method for rapid multiple sequence alignment based on fast Fourier transform[J]. Nucleic Acids Res,2002,30 (14):3059−3066. doi: 10.1093/nar/gkf436
[23] Kalyaanamoorthy S,Minh BQ,Wong TKF,von Haeseler A,Jermiin LS. ModelFinder:fast model selection for accurate phylogenetic estimates[J]. Nat Methods,2017,14 (6):587−589. doi: 10.1038/nmeth.4285
[24] Zhang D,Gao FL,Jakovlić I,Zou H,Zhang J,et al. PhyloSuite:an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Mol Ecol Resour,2020,20 (1):348−355. doi: 10.1111/1755-0998.13096
[25] Kim K,Nguyen VB,Dong JZ,Wang Y,Park JY,et al. Evolution of the Araliaceae family inferred from complete chloroplast genomes and 45S nrDNAs of 10 Panax-related species[J]. Sci Rep,2017,7 (1):4917. doi: 10.1038/s41598-017-05218-y
[26] Ran H,Liu YY,Wu C,Cao YN. Phylogenetic and comparative analyses of complete chloroplast genomes of Chinese Viburnum and Sambucus (Adoxaceae)[J]. Plants,2020,9 (9):1143. doi: 10.3390/plants9091143
[27] 田星,刘莹莹,张颖敏,杨从卫,钱子刚,李国栋. 藜芦属药用植物的叶绿体基因组比较分析和系统发育研究[J]. 中草药,2022,53(4):1127−1137. Tian X,Liu YY,Zhang YM,Yang CW,Qian ZG,Li GD. Comparative and phylogeny analysis of four Veratrum medicinal plants complete chloroplast genomes[J]. Chinese Traditional and Herbal Drugs,2022,53 (4):1127−1137.
[28] 黄琼林. 高良姜叶绿体基因组测序与特征分析[J]. 热带作物学报,2021,42(1):1−6. Huang QL. Complete sequencing and analysis of chloroplast genome from Alpinia officinarum Hance[J]. Chinese Journal of Tropical Crops,2021,42 (1):1−6.
[29] Liu J,Wen J. The complete chloroplast genome of Aralia atropurpurea (Araliaceae,the ginseng family) from the Sino-Himalayan region,China[J]. Mitochondrial DNA Part B Resour,2019,4 (2):2773−2774. doi: 10.1080/23802359.2019.1643805
[30] Kim CK,Kim YK. The complete chloroplast genome of Aralia cordata (Apiales:Araliaceae)[J]. Mitochondrial DNA Part B Resour,2019,4 (1):211−212. doi: 10.1080/23802359.2018.1546140
[31] Abdullah N,Henriquez CL,Mehmood F,Carlsen MM,Islam M,et al. Complete chloroplast genomes of Anthurium huixtlense and Pothos scandens (Pothoideae,Araceae):unique inverted repeat expansion and contraction affect rate of evolution[J]. J Mol Evol,2020,88 (7):562−574. doi: 10.1007/s00239-020-09958-w
[32] Liu CK,Yang ZY,Yang LF,Yang JB,Ji YH. The complete plastome of Panax stipuleanatus:comparative and phylogenetic analyses of the genus Panax (Araliaceae)[J]. Plant Divers,2018,40 (6):265−276. doi: 10.1016/j.pld.2018.11.001
[33] 宋菊,龙月红,林丽梅,尹峰,邢朝斌. 五加科植物叶绿体基因组结构与进化分析[J]. 中草药,2017,48(24):5070−5075. Song J,Long YH,Lin LM,Yin F,Xing ZB. Analysis on structure and phylogeny of chloroplast genomes in Araliaceae species[J]. Chinese Traditional and Herbal Drugs,2017,48 (24):5070−5075.
[34] Li R,Ma PF,Wen J,Yi TS. Complete sequencing of five Araliaceae chloroplast genomes and the phylogenetic implications[J]. PLoS One,2013,8 (10):e78568. doi: 10.1371/journal.pone.0078568
[35] Dong WP,Xu C,Li CH,Sun JH,Zuo YJ,et al. ycf1,the most promising plastid DNA barcode of land plants[J]. Sci Rep,2015,5:8348. doi: 10.1038/srep08348
[36] 郭栋梁,王静,韩冬梅,潘学文,李建光. 龙眼顶芽转录组简单重复序列(SSR)标记信息分析及分子标记开发[J]. 植物生理学报,2018,54(5):863−871. Guo DL,Wang J,Han DM,Pan XW,Li JG. Analysis on simple sequence repeat (SSR) information in apical transcriptome and development of molecular markers in Dimocarpus longan[J]. Plant Physiology Journal,2018,54 (5):863−871.
[37] 王久利,郑旭,邓因子. 暴马丁香的叶绿体基因组特征分析[J]. 阜阳师范大学学报 (自然科学版),2022,39(1):55−64. Wang JL,Zheng X,Deng YZ. Characterization of chloroplast genome of Syringa reticulata subsp. amurensis[J]. Journal of Fuyang Normal University (Natural Science)
,2022,39 (1):55−64. [38] 苏玥,刘娟娟,完斌,张鹏举,陈正根,等. 乳苣叶绿体基因组特征及其系统发育分析[J]. 中国农业科技导报,2021,23(6):33−42. Su Y,Liu JJ,Wan B,Zhang PJ,Chen ZG,et al. Chloroplast genome structure characteristic and phylogenetic analysis of Mulgedium tataricum[J]. Journal of Agricultural Science and Technology,2021,23 (6):33−42.
[39] 张韵洁,李德铢. 叶绿体系统发育基因组学的研究进展[J]. 植物分类与资源学报,2011,33(4):365−375. Zhang YJ,Li DZ. Advances in phylogenomics based on complete chloroplast genomes[J]. Plant Diversity and Resources,2011,33 (4):365−375.
[40] 许旭东,杨峻山,朱兆仪. 楤木属植物三萜皂甙研究进展[J]. 药学学报,1997,32(9):711−720. -
期刊类型引用(4)
1. 刘天付,辛培尧. 叶绿体基因组及其在樟科植物系统发育研究中的应用. 寒旱农业科学. 2024(03): 197-202 .  百度学术
百度学术
2. 马海霞,张亚颖,张玉婉,益西卓玛,梁焯森,李欣冉,郭云涛,封烁. 角蒿属药用植物叶绿体基因组比较及系统发育分析. 中草药. 2024(17): 5972-5981 .  百度学术
百度学术
3. 陈帅,陈邦,陈宇凡,陈福长,钱海珊,何红平,李宝晶. 白背叶楤木芽苞乙酸乙酯部位中芳香族化合物及其体外抗炎活性. 中南药学. 2024(10): 2596-2601 .  百度学术
百度学术
4. 刘巧珍,代江鹏,朱鹏坚,林月霞,高晓霞,朱爽. 栽培奇楠沉香叶绿体基因组特征及系统发育分析. 中国中药杂志. 2023(20): 5531-5539 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载: