Construction of two-hybrid library of yeast and screening of NnWRKY40 interacting proteins in Nelumbo nucifera Gaertn. ‘Baihuajian’
-
摘要:
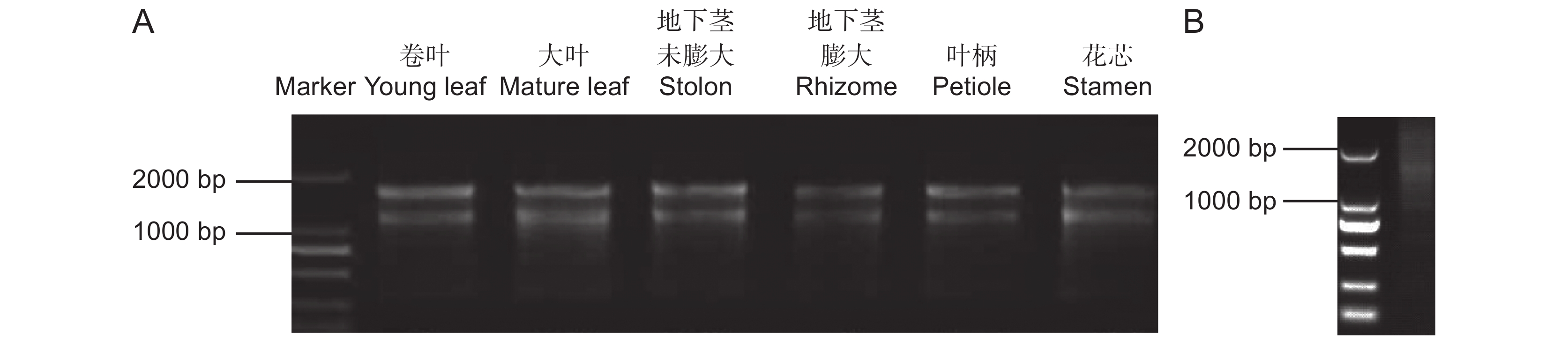
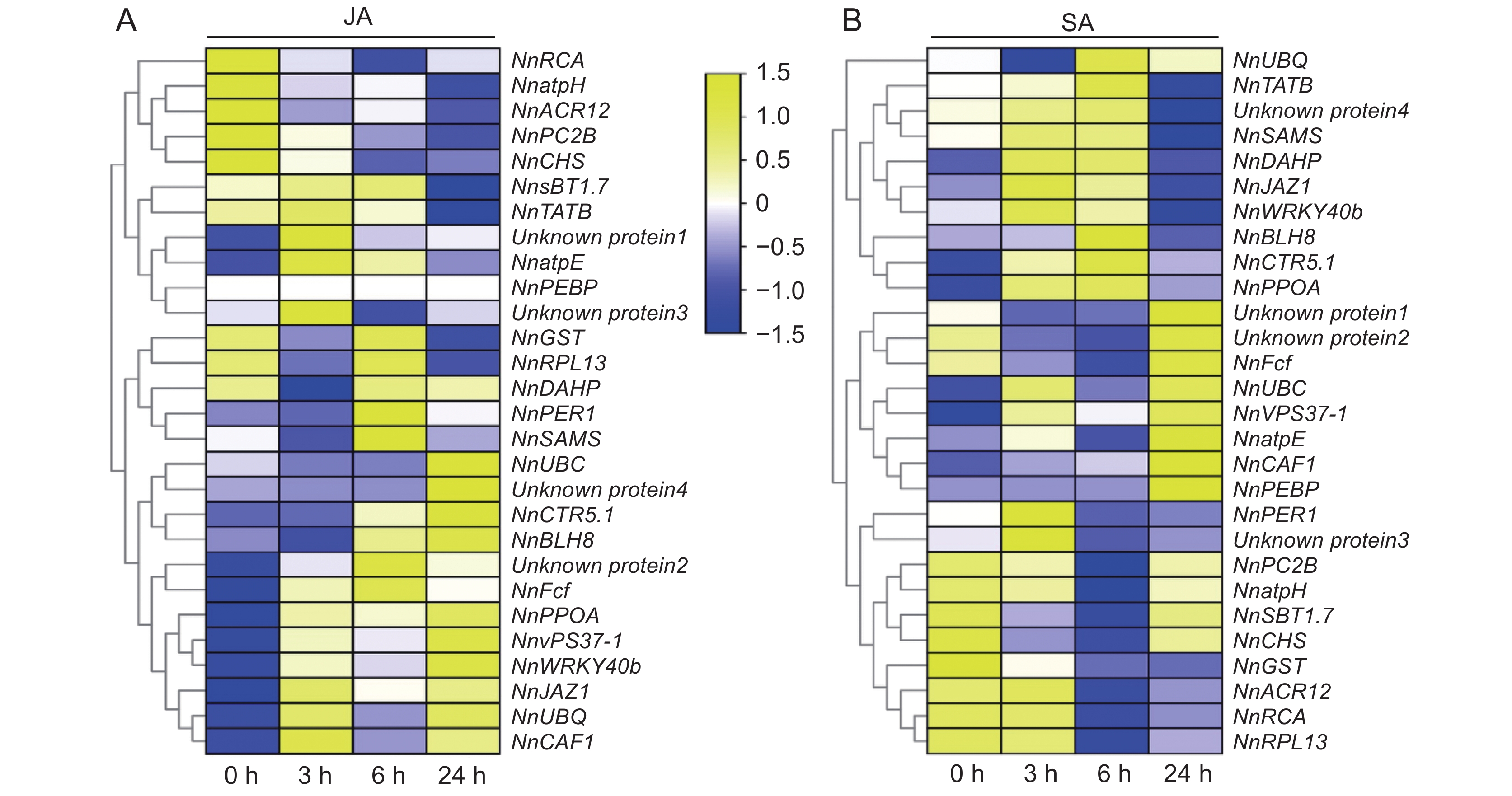
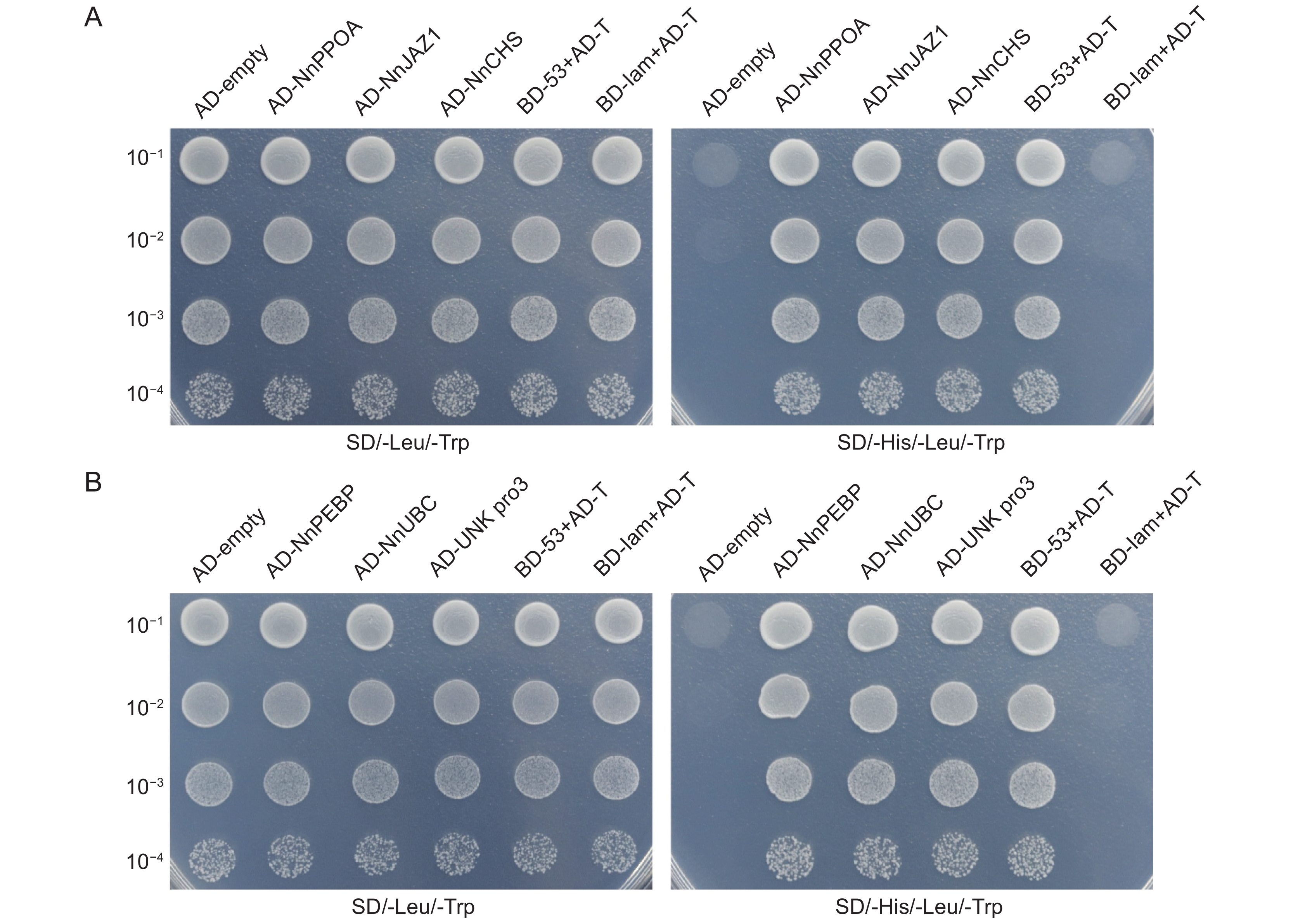
通过构建莲(Nelumbo nucifera Gaertn.)品种 ‘白花建’不同组织的混合cDNA文库,筛选与莲转录因子NnWRKY40互作的蛋白,探究NnWRKY40参与调控次级代谢物生物碱合成的可能机制。结果显示,混合cDNA文库的库容为1.2 × 107 CFU,重组率为100%,插入片段平均长度大于1000 bp。NnWRKY40包含两个同源基因NnWRKY40a和NnWRKY40b,利用NnWRKY40b构建诱饵载体pGBKT7-NnWRKY40b,通过共转化方法,从文库中筛选到27个与NnWRKY40b 互作的蛋白。这些互作蛋白可分为生长发育及抗逆、激素调控和次级代谢、未知蛋白3类。选取6个代表性互作蛋白(NnUBC、NnPEBP、NnPPOA、NnCHS、NnJAZ1和Unknown protein 3)进行一对一验证,发现其中JAZ蛋白与次生代谢物生物碱合成相关,提示NnWRKY40b转录因子可能与茉莉酸(JA)介导的生物碱合成调控密切相关。
Abstract:To explore the potential mechanism of the NnWRKY40 transcription factor in lotus (Nelumbo nucifera Gaertn.) for regulating the synthesis of secondary metabolite alkaloids, a mixed cDNA library from different lotus tissues was constructed, and the proteins interacting with NnWRKY40 were screened. Total RNA of different tissues was extracted from ‘Baihuajian’, and a mixed cDNA library was established. The library capacity was 1.2 × 107 CFU, recombinant rate was 100%, and average length of the inserted fragments was >1 000 bp. NnWRKY40 contains two homologous genes, NnWRKY40a and NnWRKY40b. As NnWRKY40b is reported to play a leading role in transcriptional activation of alkaloid synthesis genes, we used NnWRKY40b to construct the bait vector PGBKT7-NnWRKY40b. In total, 27 proteins interacting with NnWRKY40b were screened from the library using the co-transformation method. These interacting proteins could be divided into three categories: i.e., growth and development and stress response, hormone regulation and secondary metabolism, and unknown proteins. Six representative proteins, including NnUBC, NnPEBP, NnPPOA, NnCHS, NnJAZ1, and unknown protein 3, were selected for one-to-one verification, among which the JAZ protein was associated with alkaloid synthesis, suggesting that the NnWRKY40b transcription factor may be closely related to jasmonic acid (JA)-mediated regulation of alkaloid synthesis in N. nucifera.
-
Keywords:
- Nelumbo nucifera /
- Yeast two-hybrid /
- NnWRKY40 /
- Protein interaction
-
1999年,Nicholson等[1]提出了代谢组学(Metabonomics)的概念,认为代谢组学能够弥补基因组学和蛋白质组学所忽略的生物体的整个动态代谢状态,可以定量测定生命系统对病理生理刺激或基因修饰的动态多参数代谢反应。Fiehn[2]认为代谢组学可以定义为对限定条件下的特定生物样品中所有代谢组分的定性和定量,对代谢物进行全面定量的分析。该团队开创性地利用气相色谱-质谱联用(GC-MS)技术,自动定量了拟南芥(Arabidopsis thaliana (L.) Heynh)提取物中的326种代谢物,并确定了其中149个代谢物的化学结构[3]。
代谢组学技术是继蛋白质组和基因组学技术发展起来的新兴科学技术,是系统生物学的重要组成部分,在临床研究、天然产物化学等研究领域中有着举足轻重的作用。其采用高通量的色谱、质谱和核磁共振等技术对生物体代谢产物的多维数据信息与数学模型建立联系,从而实现对复杂代谢物的分析,建立完整的代谢网络[4]。同时,根据液相色谱质谱联用技术的固有特性,要求研究者使用多元变量统计方法对原始数据进行分析。与差异倍数分析、显著性检验等比较传统的单变量分析相比,多元变量统计分析,如无监督分析(主成分分析等)、监督分析(偏最小二乘判别分析等)等更加注重代谢物之间的关系,以及它们在生物过程中的促进/拮抗关系。
植物初生代谢与次生代谢产物成分复杂、种类繁多、体系庞大,传统分析方法很难检测出其所含的全部成分,代谢组学的出现能很好地解决农林业、天然产物业等领域的相关问题,如中药材或中药饮片中发挥主要疗效的活性成分的模糊性、未知性等问题[5]。代谢组学相关技术能够从根本上全方位地对天然植物所含的化学成分进行系统分析,从而获得活性物质的指纹图谱,通过多元统计分析对化学物质进行检测鉴定,从而获得该天然产物所含的化学成分种类,为其应用提供思路[6]。目前,代谢组学技术在植物表型评价、辅助育种、活性成分分析以及植物胁迫/抗逆分子机制研究等方面发挥着重要功能。
1. 代谢组学相关分析技术
1.1 代谢组学的技术分析平台
代谢组学的目标是整合广泛而复杂的数据信息集,通过可靠的技术平台,进行数据集成,并利用先进的生物统计方法,得到生物代谢过程中的动态变化和代谢通路的相关机制信息[7]。代谢组学实验旨在表征来自各种样品类型(如血清、细胞提取物、尿液等)的不同类别小分子,收集小分子样本,选择合适的平台进行分析。因其可靠性高,质谱(MS)和核磁共振(NMR)是目前代谢组学实验中最常用的技术,核磁共振光谱法操作简便,基于质谱的代谢组学方法则提供了一个高灵敏度与高选择性相结合的平台,两者在代谢组学分析中的优越性和适应性各具特色,具体如图1所示。
MS作为代谢组学中最常用的分析工具之一,具有灵敏度高 (通常是pg级)、可重复性好、多功能性强以及数据采集速度快等特点,能测量分子的质量,确定物质片段,可检测超过1000种内源性代谢物的特征,已成为极性和非极性小分子分析的领先技术。同时MS配合其他分离分析技术,更能适应对不同性质小分子代谢物的分析检测,如气相-质谱联用技术(GC-MS)可用于分析低沸点、低极性、易挥发的衍生物;而液相-质谱联用技术(LC-MS)可用于高沸点、热稳定性差的代谢物的分离分析,是目前在代谢组学领域应用范围较广的分析检测技术。同时,随着科学的发展,更高层次、高精度、高分辨率的分析检测技术如高效液相色谱-质谱联用技术与超高效液相色谱-质谱联用技术的问世,相比于LC-MS,具有灵敏度更高、峰容量更好、通量更高等优势。其次,用于检测离子型代谢物的毛细管电泳-质谱技术(CE-MS)已被证明是生物样品中极性代谢物分析的强大手段。超临界流体色谱-质谱联用技术(SFC-MS)相较于前述检测技术,可以对代谢组进行全面分析,提高了代谢组的覆盖率,并进一步提高了分析通量,在代谢组学领域具有光明的应用前景[8]。
NMR是近年来在代谢组学研究领域中能提供最丰富数据的优秀分析平台之一,在生命科学领域通常采用氢谱(1H NMR)、碳谱(13C NMR)及磷谱(31P NMR)[9],一维光谱学本质上是定量的,无需单独的代谢物标准品即可轻松用于代谢物定量,二维光谱最常用于鉴定代谢物,但也可以定量[10],其作为代谢组学研究的强大工具,可提供高度可重复的定量分析。虽然与MS和质谱相比,NMR的检测灵敏度较低,但可以提供直接定量、鉴定和简单的样品处理,且相较于MS检测过程中存在的基质干扰和离子化程度问题,NMR具有基本无偏倚性的特点,其对所有物质的检测灵敏度一致,同时,经过NMR技术检测过的样品基本无损伤,并可以设计多种编辑方法,使实验进行得更加灵活。
1.2 数据处理
1.2.1 数据预处理
代谢组学分析中使用各种分析平台,获取样品的代谢物数据,要从这些数据中提取有用的信息还需要进行一系列的数据处理,将原始数据转化为可用于进一步统计分析的形式。数据处理过程一般包括NMR或MS原始数据的获得、数据预处理(包含去噪、基线校正、去同位素、峰对齐、分箱、峰识别、均一化、数据缩放等)。通过数据库对质谱数据进行采集及目标化合物定性分析工作,通常GC-MS数据选择NIST数据库,LC-MS选择METLIN数据库或者Fiehn数据库等。然后,使用自撰写R程序包或相关软件结合自建数据库完成提峰、注释等工作。接着对原始数据进行准备和处理,最后进行数据标准化处理,对数据进行优化,获得代谢物列表。
1.2.2 统计分析
基于代谢组学数据算法框架应运而生的软件(如XCMS、MZmine 2),可用于对原始数据进行峰注释、峰对齐等相关处理,从而获得具有代谢物特征的数据模式,以便进行后续的数据挖掘和统计分析[11]。通过整理,可将枯燥的数据可视化,转化为更易分析的形式。利用数据处理软件获得需要的展现形式,如主成分分析图,能直观地表示组内差异及组间差异,判定数据质量如何,有没有离散样本,结果是否可信,且在后续分析中可以去掉离散样品,提高最终结果的可靠度[12]。偏最小二乘法分析图和正交偏最小二乘法分析图则采用变量重要性投影(VIP)值结合t检验P值对代谢物进行统计分析,以VIP > 1.2且P < 0.05为标准,进行组间差异代谢物的筛选[13]。数据处理的最后一步是对差异代谢物进行分析,通过各类代谢组学数据库对筛选的代谢物进行鉴定及通路分析,进行代谢物聚类,获得差异代谢物聚类热图等结果。
1.3 代谢组学联合分析
代谢组学联合其他组学研究模式在植物领域也已经广泛使用,为系统阐述植物相关作用机制提供了巨大帮助。目前已有转录组学-代谢组学联合分析、蛋白质组学-代谢组学联合分析、基因组学-代谢组学联合分析及微生物组学-代谢组学联合分析等。代谢组是生物体表型的直接体现者,代谢物是生物体的最终产物,是生物体表型的物质基础。结合基因组或转录组,从“原因”与“结果”两个角度,从不同层次对生物体的内在变化进行分析,鉴定关键基因靶点、代谢物及代谢通路,构建核心调控网络,能够系统地阐述植物体生长发育、疾病发生、中药材药效作用等机制,从整体上理解植物产生内源性变化的机理。
目前还出现了基于代谢物的全基因组关联分析(mGWAS)、数量性状位点关联分析(mQTL)、长非编码RNA关联分析(lncRNA)等研究手段。利用全谱代谢组检测队列样本的代谢组数据,基于SNP重测序、转录组测序数据等,把代谢物作为表型与基因型数据进行关联分析,有利于批量定位调控代谢物的候选基因,挖掘调控产量、品质和环境应答等相关代谢通路,深入了解植物代谢物合成调控的遗传机制,从而解释生物学问题。目前,联合分析技术已应用于植物发育时空代谢谱的解析以及植物应激反应机制解析等,在深入研究植物代谢合成调控机制中发挥了重要作用。
2. 代谢组学在植物次生代谢调控研究中的应用
由于代谢组学技术所提供的众多优秀平台及数据整合分析系统,其在植物研究领域也发挥着极其重要的作用。各类植物所含的化学物质千变万化,具有多样性、不稳定性以及成分结构复杂等特点。因此,采用传统方法进行提取分离、纯化鉴定以及活性评价等准确性较低,且过程复杂,而代谢组学技术能够进行高通量检测、鉴定、定量以及代谢通路分析,可用于捕获植物化学复杂性的快照,能够快速测量其累积的植物化学物质,并揭示其代谢特征与响应遗传或环境变化的潜在关系[14],植物代谢组分析流程见图2。
2.1 环境胁迫相关研究
2.1.1 生物胁迫相关研究
代谢组学技术被越来越多地用于研究更全面的植物-病原菌相互作用机制。植物在生长发育过程中,会遭受到有害生物的侵染,如细菌、真菌、病毒、线虫等。在受到病原生物侵染时,植物可以通过调节体内的代谢反应,产生一系列的初、次级代谢产物。通过代谢组学技术分析病菌入侵前后代谢物谱图之间的差异,可以分析物质的变化,从而寻找抗病相关的代谢物,同时有效识别参与代谢过程的重要基因[15],这些抗病代谢物或基因可能给植物抗性育种带来新的研究思路和方向。
代谢组学技术已经用于分析核桃(Juglans regia L.)[16]、红枣(Ziziphus jujuba Mill.)[17]、柑橘(Citrus reticulata Blanco)[18, 19]、藜麦(Chenopodium quinoa Willd.)[20]、小麦(Triticum aestivum L.)[21, 22]、人参(Panax ginseng C. A. Meyer)[23]、葡萄(Vitis vinifera L.)[24]的防御机制,并揭示了防御过程中的重要反应,相关研究见表1。这些研究加深了对植物抗病虫害相关机制的理解,也为后期开发天然抗性药物提供了参考。
表 1 植物生物胁迫代谢组研究列表Table 1. List of metabolomics studies related to plant disease control物种
Species侵染病害
Infectious disease相关代谢物
Related metabolites代谢组学分析
Metabolomics analysis参考文献
Reference核桃
Juglans regia L.胶孢炭疽菌 花青素B1/2/3、咖啡酸、没食子酸、柚皮素等 探讨了炭疽菌的发生机制 [16] 红枣
Ziziphus jujuba Mill.链格孢菌、细极链格孢菌 链格孢酚、细交链孢菌酮酸;天冬氨酸、甲硫氨酸和赖氨酸 阐释了黑斑病变过程中链格孢毒素的产生机理 [17] 柑橘
Citrus reticulata Blanco柑橘绿霉菌 糖类、脂质、有机酸 揭示柑橘采后绿脓杆菌的侵染机制 [18] 藜麦
Chenopodium quinoa Willd.霜霉病菌 蔗糖、生物胺、天冬氨酸、ɣ-氨基丁酸等氨基酸 揭示了毒素产生机理 [20] 小麦
Triticum aestivum L.禾谷镰刀菌 氨基酸 优化代谢物提取方法,探究染菌发生机制 [21] 小麦
Triticum aestivum L.腥黑粉菌 葫芦酸、十八碳三烯酸 揭示了籽粒腥黑穗病菌侵染前后代谢谱差异 [22] 柑橘
Citrus reticulata Blanco黄龙病 氨基酸、植物激素、嘌呤、水杨酸 证明了耐黄龙病品种的抗性策略 [19] 人参
Panax ginseng C. A. Meyer人参锈根症状 木质素、脂质、生物碱 揭示了病害的潜在分子机制 [23] 葡萄
Vitis vinifera L.中华蚱蜢、灰葡萄孢菌 脯氨酸等氨基酸、天冬氨酰-L、谷胱甘肽和一些脂肪酸 揭示了代谢组水平的复杂网络调控 [24] 植物在生长过程中不可避免地会遭受到多种自然灾害的危害,其中病虫害尤为普遍和严重,可能会使植株生长发育不良,严重的还可造成植株品种退化、植物死亡等,影响农业、林业及药材行业的经济效益和社会效益。而代谢组学技术在预防病虫害、掌握植物发病机理、研发防治病虫害药物方面发挥着巨大作用。
2.1.2 植物非生物胁迫代谢组研究
植物在生长发育过程中会受到生物(如真菌、细菌、虫害等)以及非生物的胁迫,非生物胁迫主要是环境带来的,包括光照、水分、盐分、紫外线、重金属、温度等因素。植物也会根据当前的应激条件对代谢网络进行配置,维持必要的代谢,采用新的稳态来适应逆境[25]。逆境胁迫下代谢组的重塑很大程度上反映了植物体对逆境胁迫的响应和防御,而代谢组学技术为研究不同逆境胁迫下代谢物的重塑提供了可靠手段。
在植物栽培繁育方面,工作人员需要从土壤特性、光照习性、温度等方面考虑植物生长发育的适宜性。而土壤中的化学元素会影响植物发育,如盐分能抑制不喜盐植物的生长。利用代谢组学技术可探究植物对盐的响应机制[26-28],为提高植物的抗盐能力奠定基础。同时随着农业环境的恶化,土壤中的重金属元素超标,如铅[25]等对植物的生长发育造成了严重的干扰,揭示植物对重金属胁迫的耐受策略就显得十分重要。除此之外,代谢组学技术对于植物所受的水分[29]、温度[30, 31]等其他非生物胁迫也可起到分析、监测的作用,相关研究见表2。
表 2 植物非生物胁迫代谢组研究列表Table 2. List of metabolomics studies related to plant abiotic stress物种
Species非生物胁迫因素
Abiotic stress factors相关代谢物
Related metabolites代谢组学分析
Metabolomics analysis参考文献
Reference蓝莓 Vaccinium spp. 盐 甘氨酸、苹果酸、十八烷酸、L-苏糖酸等 耐盐性机理 [26] 水稻 Oryza sativa L. 铅 脂质、类二十烷酸 根际微环境与重金属胁迫耐受性 [25] 白刺 Nitraria tangutorum Bobr. 盐 氨基酸、糖、脂肪酸 白刺对盐胁迫的代谢响应 [27] 党参 Codonopsis pilosula (Franch.) Nannf. 干旱 脂类、糖类 干旱胁迫后产物 [29] 黑麦草 Lolium perenne L. 盐 糖类、氨基酸及其中间代谢物 耐盐代谢通路 [28] 早熟禾 Poa annua L. 低温 糖和糖醇、氨基酸、有机酸 适应低温的主要机制 [30] 橡胶树 Hevea brasiliensis (Willd. ex A. Juss.)Muell. 低温 糖类、氨基酸、脂质类 低温胁迫代谢物响应机制 [31] 2.2 自身因素
代谢组学技术在植物研究中的应用包括但不限于探究植物的代谢途径和代谢网络、环境对植物化学成分的影响、植物化学成分的作用及相关价值等方面。我国横跨寒带、温带、亚热带等气候类型,且地貌丰富,因此物种丰富度高,植物种类多样。不同种属植物或同种植物在不同生长环境、不同组织、不同发育阶段其所含代谢物差异明显,从而造成植物表型的差异,植株的功能也会发生改变。对于中医药产业来说,中药材的化学成分发生变化,在临床上可能会产生不同的疗效。代谢组学技术的出现弥补了中医药行业无法可视化的短板,有助于推动中医药行业的可持续发展,以及向国际化发展。同时,为了寻找植株产生差异的原因,促进植物资源的可持续利用及绿色发展,需要对该方面进行深层次的研究,新兴的代谢组学技术结合其他组学技术已经在该领域做出了重要贡献,具体内容见附表1
1 )。2.2.1 品种代谢物差异
植物代谢组学通过对代谢物的全面分析,可以鉴别农作物、树木以及中药材,结合多元统计分析,还可进一步进行质量控制。在代谢组学分析中,无监督分析能够最大程度地保留数据的原始状态,适合分析差异较小的品种。监督型分析适合差异较大的物种鉴别。不同种属的植物,其化学成分、品质、活性、药性等不同,且对周围环境产生的作用各异,因此,植物种属鉴定对于后续的应用至关重要。
在农业、林业、中药材等行业中,尤其是中药材行业,假冒伪劣产品防不胜防,除去人为因素,同种中药材,商品规格、品种等不同也会造成产品的差异性。如中药皂荚(Gleditsia sinensis Lam.)[32]、忍冬(Lonicera japonica Thunb.)[33]、百合(Lilium)[34]、柴胡(Bupleurum)[35]、黄精属(Polygonatum)[36]、人参属(Panax)[37]等植物均有两种及以上药材选择,每种药材的表型性状及其代谢物、功效等存在一定的差异,这就需要对其进行质量控制。代谢组学技术在区分不同种属药材、质量控制以及临床应用方面已较为成熟,且得到广泛应用。Jiang等[38]对5个品种荔枝(Litchi chinensis Sonn.)中所含的多酚类物质进行了代谢组学研究,结果发现品种间差异显著,且品种成熟度与酚类物质的组成及含量存在一定的相关性。同时,代谢组学技术对不同茶叶品种[39,40]及其所含活性物质等方面、农作物栽培育种、功能活性、营养价值评价研究[41-45]等方面也提供了支持。
2.2.2 组织代谢物差异
对于植物整体而言,不同组织[46, 47]的代谢物、功效等存在差异,将一种材料与其突变体或基因改造植物的代谢产物进行比较,对于筛选优良品种或基因、评价基因改造效果及进行相关功能基因组学研究有重要意义。
2.2.3 环境引起的代谢物差异
植物的生长繁衍需要特定的环境条件,不同的环境因子对植物物种的空间分布具有一定的影响,植物的组成和分布是环境与植物相互作用的结果[48]。环境因子如营养条件、地理位置、种植方式等的差异,会造成植物体初生及次生代谢的差异;环境因素如施肥条件[49, 50]、种植密度[51]、栽培环境[52, 53]以及产地[54, 55]等也会影响植株的代谢物、代谢水平以及代谢途径。代谢组学与多元统计分析相结合,可在环境造成的植物代谢物差异性研究方面提供新的方法与手段,促进资源的合理利用。
2.2.4 不同发育阶段的代谢物差异
植物从种子到植株的整个生长发育阶段,体内的代谢物不断转化,化学物质经过不同生物合成途径转变成自身所需的成分。了解植株在整个生长发育过程中的动态变化,在植物育种,特别是药材良种培育方面极其重要。
比较不同发育阶段有助于确定最佳收获时间,提高农业经济效益,并可揭示生长发育阶段的调控机制以及活性物质的积累机制等。代谢组学技术已应用于植物种子萌发[56-59]、开花[60-62]、果实发育[63-65]等阶段的差异代谢物检测,以及活性物质基础、发育过程中的动态变化检测等方面。
利用代谢组学技术对植物次生代谢物的变化特征进行分析,找出植物自身变化与代谢物的联系,从而发掘出与表型相关的代谢产物,对于揭示植物病理变化、品种差异以及进行质量评价等领域的研究意义重大。
3. 小结与展望
代谢组学技术在植物研究领域中占据重要位置,植物的次生代谢物在不同领域也发挥着重要作用。因此,利用代谢组学技术对植物体内的次生代谢物进行定性定量,同时结合数据处理软件对植物整体进行相关分析,能够直观地掌握植株之间的差异性,揭示植物次生代谢途径,阐明代谢网络调控机制。近年来,代谢组联合其他组学技术在揭示植物生长发育、生物及非生物胁迫、色味变化、活性成分、作用机理以及合成通路等方面的研究中发挥了越来越重要的作用。已有最新报道展现了代谢组学联合其他组学技术在研究植物生物防治、自交不亲和机制、代谢物空间分布等方面的优势。
随着对天然产物研究的深入,揭示植物生长发育过程中的代谢物及其代谢途径越来越重要。但目前对天然产物的取样存样以及定性定量分析还存在瓶颈,相关数据库还不够完善,通过代谢组学数据库鉴定的代谢物和代谢通路还较少,代谢物识别能力较为有限。同时,目前代谢物质的判断方法也比较单一,代谢物的分布状态无法反映代谢途径的状态,非靶向的代谢组学技术只能对代谢物进行半定量,这些不足之处尚需攻克。植物代谢组学可以结合其他组学技术以及其他学科(如微生物学,环境资源学,药理学等),进行多方向的探索,将代谢物与微生物、环境因子、基因、蛋白质等相结合,以共同揭示植物生物学信息。相信组学联合技术将会成为今后植物研究领域的热点,使学者解决植物领域研究中的难题成为可能。
-
表 1 互作蛋白点对点验证所用引物
Table 1 Primers used in one-to-one verification of interacting proteins
引物名
Primer name正向序列(5′–3′)
Sequence of forward primer反向序列(5′–3′)
Sequence of reverse primerpGBKT7-NnWRKY40b CATGGAGGCCGAATTCATGGAGTC
GACTTGGTTGGATACGCAGGTCGACGGATCCTCACCA
TTTCTGCACTGTTGAATGpGADT7-NnJAZ1 CAGATTACGCTCATATGATGTCAA
GAGCGCCGGACCTTGCTTGGGTGGAATTCCTACTGT
GGAGATCGAGCTTGTpGADT7-NnUBC CAGATTACGCTCATATGATGGCGA
ACAGCAATCTACCCTGCTTGGGTGGAATTCTCAGGCA
CCACTTGCATATAGpGADT7-NnCHS CAGATTACGCTCATATGATGGTGA
CCGTGGAAGACATCTGCTTGGGTGGAATTCCTAGGCA
GCGATACTGTGAAGpGADT7-NnPEBP CAGATTACGCTCATATGATGGCGA
GTGACGAGTTTAGGTTGCTTGGGTGGAATTCTTAGGCT
GGGAAAAGTCGGATCpGADT7-NnPPOA CAGATTACGCTCATATGATGGCA
TCGCTTTCTCCCTTGATGCTTGGGTGGAATTCTCACGAA
GCGAACACTATCTTGpGADT7-Unknown protein3 CAGATTACGCTCATATGATGCAT
TCCCTGAGCTTAAAACTTGCTTGGGTGGAATTCTTAGACG
ATATCCGTATCATCTC表 2 NnWRKY40b互作蛋白筛选及其功能预测
Table 2 Screening and functional prediction of NnWRKY40b interacting proteins
分类
Classification蛋白号
Protein ID基因号
Gene ID蛋白名称
Protein name相关蛋白功能预测
Protein function prediction生长发育及
抗逆XP_010271938.1 LOC104607876 枯草杆菌蛋白酶,NnSBT1.7 种皮发育相关 XP_010266914.1 LOC104604316 质体蓝素,NnPC2B 参与光合作用 XP_010279114.1 LOC104613113 泛素结合酶,NnUBC DNA修复,光周期,抗逆胁迫响应,降解生长素,延缓植物衰老,调控ABA信号途径 YP_009093956.1 LOC20834983 ATP合成酶CF1亚基,NnatpE 光合作用,细胞代谢 XP_010265991.1 LOC104603626 类似谷胱甘肽S-转移酶U17, NnGST 抗逆反应,植物修复 XP_010269750.1 LOC104606314 铜转运蛋白5.1,NnCTR5.1 光合作用,呼吸作用,细胞壁代谢,氧化应激反应 XP_010255313.1 LOC104596029 非依赖性蛋白转位酶蛋白,NnTATB 细胞内运输、分泌和囊泡转运 XP_010248208.1 LOC104591115 二磷酸核酮糖羧化酶/加氧酶激活酶,NnRCA 光合作用,叶片衰老,响应非生物胁迫 XP_010270928.1 LOC104607108 半胱氨酸过氧化物氧还蛋白,NnPER1 细胞氧化还原稳态,细胞氧化剂解毒 XP_010269352.1 LOC104606034 60S核糖体蛋白L13e,NnRPL13 翻译、核糖体结构与生物发生 XP_010270872 LOC104607076 ADP-核糖基化因子,NnBLH8 细胞内运输、分泌和囊泡转运 XP_010275748.1 LOC104610704 核糖核酸酶,NnCAF 1 RNA降解 XP_010241640.1 LOC104586181 液泡蛋白分选相关蛋白,NnVPS37-1 盐胁迫响应 XP_010264580.1 LOC104602549 磷脂酰乙醇胺结合蛋白,NnPEBP 植物生长发育,几种信号通路的调节,如MAP激酶通路 XP_010251283.1 LOC104593218 类似Fcf2蛋白,NnFcf 胚成熟,花瓣分化,叶片衰老 XP_010263125.1 LOC104601478 ATP合成酶, NnatpH 光合作用,细胞代谢 XP_010244725.1 LOC104588480 类ACR12蛋白,NnACR12 光合电子传递,冷响应,光响应 激素调控和
次级代谢XP_010258950.1 LOC104598530 泛素蛋白,NnUBQ 蛋白降解,茉莉酸信号途径,细胞周期 XP_010251469.1 LOC104593386 类似TIFY 10A蛋白,NnJAZ1 抑制JA信号传导,响应盐胁迫,
花的发育,茎叶的发育NP_001305084.1 LOC104602160 查尔酮合成酶,NnCHS 类黄酮的生物合成,生长素运输的调节,根系向重力性的调节 XP_010273014.1 LOC104608661 DAHP合成酶,NnDAHP 分支酸合成 ADC92563.1 LOC104588895 多酚氧化酶,NnPPOA 类黄酮、木质素、原花青素生物合成过程 XP_010270953.1 LOC104607120 S-腺苷甲硫氨酸合酶5,NnSAMS 木质素生物合成过程,蛋氨酸代谢过程,冷反应 未知 XP_010261469.1 LOC104600297 未表征蛋白1 未知 XP_010260316.1 LOC104599465 未表征蛋白2 未知 XP_010248518.1 LOC104591415 未表征蛋白3 未知 XP_010276554.1 LOC104611264 未表征蛋白4 未知 -
[1] 陈强,张华,沙玫,刘永静. 一测多评法同时测定荷叶中4种生物碱含量[J]. 福建中医药,2020,51(6):29−32. doi: 10.3969/j.issn.1000-338X.2020.06.011 Chen Q,Zhang H,Sha M,Liu YJ. Simultaneous determination of four alkaloids in nelumbinis folium by quantitative analysis of multi-components by single marker[J]. Fujian Journal of Traditional Chinese Medicine,2020,51 (6):29−32. doi: 10.3969/j.issn.1000-338X.2020.06.011
[2] Wan Y,Xia J,Xu JF,Chen L,Yang Y,et al. Nuciferine,an active ingredient derived from lotus leaf,lights up the way for the potential treatment of obesity and obesity-related diseases[J]. Pharmacol Res,2022,175:106002. doi: 10.1016/j.phrs.2021.106002
[3] Abdallah BM,Ali EM. Green synthesis of silver nanoparticles using the Lotus lalambensis aqueous leaf extract and their anti-candidal activity against oral candidiasis[J]. ACS Omega,2021,6 (12):8151−8162. doi: 10.1021/acsomega.0c06009
[4] Tong YL,Li ZW,Wu YK,Zhu SL,Lu KK,He Z. Lotus leaf extract inhibits ER- breast cancer cell migration and metastasis[J]. Nutr Metab,2021,18 (1):20. doi: 10.1186/s12986-021-00549-0
[5] Van der Fits L,Memelink J. ORCA3,a jasmonate-responsive transcriptional regulator of plant primary and secondary metabolism[J]. Science,2000,289 (5477):295−297. doi: 10.1126/science.289.5477.295
[6] Kato N,Dubouzet E,Kokabu Y,Yoshida S,Taniguchi Y,et al. Identification of a WRKY protein as a transcriptional regulator of benzylisoquinoline alkaloid biosynthesis in Coptis japonica[J]. Plant Cell Physiol,2007,48 (1):8−18. doi: 10.1093/pcp/pcl041
[7] Suttipanta N,Pattanaik S,Kulshrestha M,Patra B,Singh SK,Yuan L. The transcription factor CrWRKY1 positively regulates the terpenoid indole alkaloid biosynthesis in Catharanthus roseus[J]. Plant Physiol,2011,157 (4):2081−2093. doi: 10.1104/pp.111.181834
[8] Agarwal P,Pathak S,Lakhwani D,Gupta P,Asif MH,Trivedi PK. Comparative analysis of transcription factor gene families from Papaver somniferum:identification of regulatory factors involved in benzylisoquinoline alkaloid biosynthesis[J]. Protoplasma,2016,253 (3):857−871. doi: 10.1007/s00709-015-0848-8
[9] Zhou ML,Memelink J. Jasmonate-responsive transcription factors regulating plant secondary metabolism[J]. Biotechnol Adv,2016,34 (4):441−449. doi: 10.1016/j.biotechadv.2016.02.004
[10] Tripathi S,Sangwan RS,Mishra B,Jadaun JS,Sangwan NS. Berry transcriptome:insights into a novel resource to understand development dependent secondary metabolism in Withania somnifera(Ashwagandha)[J]. Physiol Plant,2020,168 (1):148−173. doi: 10.1111/ppl.12943
[11] Hao XL,Xie CH,Ruan QY,Zhang XC,Wu C,et al. The transcription factor OpWRKY2 positively regulates the biosynthesis of the anticancer drug camptothecin in Ophiorrhiza pumila[J]. Hortic Res,2021,8 (1):7. doi: 10.1038/s41438-020-00437-3
[12] Eulgem T,Rushton PJ,Robatzek S,Somssich IE. The WRKY superfamily of plant transcription factors[J]. Trends Plant Sci,2000,5 (5):199−206. doi: 10.1016/S1360-1385(00)01600-9
[13] Ishiguro S,Nakamura K. Characterization of a cDNA encoding a novel DNA-binding protein,SPF1,that recognizes SP8 sequences in the 5′ upstream regions of genes coding for sporamin and β-amylase from sweet potato[J]. Mol Gen Genet,1994,244 (6):563−571. doi: 10.1007/BF00282746
[14] 向小华,吴新儒,晁江涛,杨明磊,杨帆,等. 普通烟草WRKY基因家族的鉴定及表达分析[J]. 遗传,2016,38(9):840−856. doi: 10.16288/j.yczz.16-016 Xiang XH,Wu XR,Chao JT,Yang ML,Yang F,et al. Genome-wide identification and expression analysis of the WRKY gene family in common tobacco (Nicotiana tabacum L. )[J]. Hereditas,2016,38 (9):840−856. doi: 10.16288/j.yczz.16-016
[15] Cormack RS,Eulgem T,Rushton PJ,Köchner P,Hahlbrock K,Somssich IE. Leucine zipper-containing WRKY proteins widen the spectrum of immediate early elicitor-induced WRKY transcription factors in parsley[J]. BBA-Gene Struct Expr,2002,1576 (1-2):92−100. doi: 10.1016/S0167-4781(02)00298-1
[16] 郑超,郑二松,王栩鸣,李冬月,杨勇,等. 水稻WRKY转录调控因子研究进展[J]. 生物技术通讯,2018,29(2):286−294. doi: 10.3969/j.issn.1009-0002.2018.02.026 Zheng C,Zheng ES,Wang XM,Li DY,Yang Y,et al. Research progress on rice WRKY transcription factors[J]. Letters in Biotechnology,2018,29 (2):286−294. doi: 10.3969/j.issn.1009-0002.2018.02.026
[17] 杨致荣,王兴春,薛金爱,孟令芝,李润植. 药用植物长春花WRKY转录因子的鉴定及表达谱分析[J]. 生物工程学报,2013,29(6):785−802. doi: 10.13345/j.cjb.2013.06.006 Yang ZR,Wang XC,Xue JA,Meng LZ,Li RZ. Identification and expression analysis of WRKY transcription factors in medicinal plant Catharanthus roseus[J]. Chinese Journal of Biotechnology,2013,29 (6):785−802. doi: 10.13345/j.cjb.2013.06.006
[18] Yamada Y,Nishida S,Shitan N,Sato F. Genome-wide profiling of WRKY genes involved in benzylisoquinoline alkaloid biosynthesis in California Poppy (Eschscholzia californica)[J]. Front Plant Sci,2021,2:699326.
[19] Wei HW,Chen SY,Niyitanga S,Liu T,Qi JM,Zhang LW. Genome-wide identification and expression analysis response to GA3 stresses of WRKY gene family in seed hemp (Cannabis sativa L. )[J]. Gene,2022,822:146290. doi: 10.1016/j.gene.2022.146290
[20] Mishra S,Triptahi V,Singh S,Phukan UJ,Gupta MM,et al. Wound induced tanscriptional regulation of benzylisoquinoline pathway and characterization of wound inducible PsWRKY transcription factor from Papaver somniferum[J]. PLoS One,2013,8 (1):e52784. doi: 10.1371/journal.pone.0052784
[21] He J,Bouwmeester HJ,Dicke M,Kappers IF. Transcriptional and metabolite analysis reveal a shift in direct and indirect defences in response to spider-mite infestation in cucumber (Cucumis sativus)[J]. Plant Mol Biol,2020,103 (4-5):489−505. doi: 10.1007/s11103-020-01005-y
[22] 代红洋,柏旭,李晓岗,张兴开,罗霖,等. 植物激素在三萜类化合物生物合成中的作用及调控机制研究进展[J]. 中草药,2021,52(20):6391−6402. Dai HY,Bai X,Li XG,Zhang XK,Luo L,et al. Research progress on roles of phytohormone in biosynthesis of triterpenoids and their regulatory mechanisms[J]. Chinese Traditional and Herbal Drugs,2021,52 (20):6391−6402.
[23] Wasternack C,Song SS. Jasmonates:biosynthesis,metabolism,and signaling by proteins activating and repressing transcription[J]. J Exp Bot,2017,68 (6):1303−1321.
[24] Yang J,Duan GH,Li CQ,Liu L,Han GY,et al. The crosstalks between jasmonic acid and other plant hormone signaling highlight the involvement of jasmonic acid as a core component in plant response to biotic and abiotic stresses[J]. Front Plant Sci,2019,10:1349. doi: 10.3389/fpls.2019.01349
[25] Ming R,VanBuren R,Liu YL,Yang M,Han YP,et al. Genome of the long-living sacred lotus (Nelumbo nucifera Gaertn. )[J]. Genome Biol,2013,14 (5):R41. doi: 10.1186/gb-2013-14-5-r41
[26] Zhang Y,Rahmani RS,Yang XY,Chen JM,Shi T. Integrative expression network analysis of microRNA and gene isoforms in sacred lotus[J]. BMC Genomics,2020,21 (1):429. doi: 10.1186/s12864-020-06853-y
[27] Li J,Xiong YC,Li Y,Ye SQ,Yin Q,et al. Comprehensive analysis and functional studies of WRKY transcription factors in Nelumbo nucifera[J]. Int J Mol Sci,2019,20 (20):5006. doi: 10.3390/ijms20205006
[28] Ferrer JL,Austin MB,Stewart C Jr,Noel JP. Structure and function of enzymes involved in the biosynthesis of phenylpropanoids[J]. Plant Physiol Biochem,2008,46 (3):356−370. doi: 10.1016/j.plaphy.2007.12.009
[29] Ren GH,Wang BJ,Zhu XD,Mu Q,Wang C,et al. Cloning,expression,and characterization of miR058 and its target PPO during the development of grapevine berry stone[J]. Gene,2014,548 (2):166−173. doi: 10.1016/j.gene.2014.07.021
[30] 王馨雨,杨绿竹,王婷,王蓉蓉,刘洁,等. 植物多酚氧化酶的生理功能、分离纯化及酶促褐变控制的研究进展[J]. 食品科学,2020,41(9):222−237. doi: 10.7506/spkx1002-6630-20190411-145 Wang XY,Yang LZ,Wang T,Wang RR,Liu J,et al. Recent progress toward understanding the physiological function,purification,and enzymatic browning control of plant polyphenol oxidases[J]. Food Science,2020,41 (9):222−237. doi: 10.7506/spkx1002-6630-20190411-145
[31] 田娇,刘园,房敏峰. 外源茉莉酸类激素对药用植物次生代谢的影响研究[J]. 天然产物研究与开发,2015,27(1):185−190. doi: 10.16333/j.1001-6880.2015.01.037 Tian J,Liu Y,Fang MF. Review on the influence of exogenous jasmonates on medicinal plant secondary metabolism[J]. Natural Product Research and Development,2015,27 (1):185−190. doi: 10.16333/j.1001-6880.2015.01.037
[32] 王金利,史胜青,贾利强,江泽平. 植物泛素结合酶E2功能研究进展[J]. 生物技术通报,2010(4):7−10. doi: 10.13560/j.cnki.biotech.bull.1985.2010.04.002 Wang JL,Shi SQ,Jia LQ,Jiang ZP. Progress on functions of ubiquitin-conjugating enzyme (E2) in plants[J]. Biotechnology Bulletin,2010 (4):7−10. doi: 10.13560/j.cnki.biotech.bull.1985.2010.04.002
[33] 李兴芬,苗雅慧,孙永江,张孟娟,张凌云. 青杄PwPEBP基因及其启动子序列的克隆与表达分析[J]. 北京林业大学学报,2019,41(4):8−20. Li XF,Miao YH,Sun YJ,Zhang MJ,Zhang LY. Cloning and expression analysis of PwPEBP gene and promoter sequence in Picea wilsonii[J]. Journal of Beijing Forestry University,2019,41 (4):8−20.
[34] 王寻,高凝,张富军,韩月彭,王小非,等. 苹果磷脂酰乙醇胺结合蛋白PEBP家族基因的鉴定与比较分析[J]. 植物生理学报,2021,57(10):1996−2010. doi: 10.13592/j.cnki.ppj.2020.0395 Wang X,Gao N,Zhang FJ,Han YP,Wang XF,et al. Identification and comparative analysis of phosphatidyl ethanolamine binding protein (PEBP) family gene in apple[J]. Plant Physiology Journal,2021,57 (10):1996−2010. doi: 10.13592/j.cnki.ppj.2020.0395
[35] 祝一文,车永梅,赵方贵,朱丹,刘新. 碱胁迫下H2S参与活性氧代谢和水稻幼苗生长的调控[J]. 农业生物技术学报,2018,26(7):1124−1131. Zhu YW,Che YM,Zhao FG,Zhu D,Liu X. H2S functions in growth regulation in rice (Oryza sativa) seedling and metabolism modulating of reactive oxygen under alkaline stress[J]. Journal of Agricultural Biotechnology,2018,26 (7):1124−1131.
[36] 张金梅,白雪,李玥莹,张颖. WRKY响应植物逆境的“角色”[J]. 安徽农业科学,2020,48(12):5−8. doi: 10.3969/j.issn.0517-6611.2020.12.002 Zhang JM,Bai X,Li YY,Zhang Y. WRKY’s “Role” in response to plant adversity[J]. Journal of Anhui Agricultural Sciences,2020,48 (12):5−8. doi: 10.3969/j.issn.0517-6611.2020.12.002
[37] 魏昕,刘雨恒,刘宇阳,殷晓浦,谢恬,等. 植物JAZ蛋白家族研究进展[J]. 植物生理学报,2021,57(5):1039−1046. doi: 10.13592/j.cnki.ppj.2020.0532 Wei X,Liu YH,Liu YY,Yin XP,Xie T,et al. Advances of JAZ family in plants[J]. Plant Physiology Journal,2021,57 (5):1039−1046. doi: 10.13592/j.cnki.ppj.2020.0532
[38] Chini A,Fonseca S,Fernández G,Adie B,Chico JM,et al. The JAZ family of repressors is the missing link in jasmonate signalling[J]. Nature,2007,448 (7154):666−671. doi: 10.1038/nature06006
[39] Thines B,Katsir L,Melotto M,Niu YJ,Mandaokar A,et al. JAZ repressor proteins are targets of the SCFCOI1 complex during jasmonate signalling[J]. Nature,2007,448 (7154):661−665. doi: 10.1038/nature05960
[40] Chen XZ,Li JR,Liu YT,Wu DD,Huang HL,et al. PatSWC4,a methyl jasmonate-responsive MYB (v-myb avian myeloblastosis viral oncogene homolog)-related transcription factor,positively regulates patchoulol biosynthesis in Pogostemon cablin[J]. Ind Crops Prod,2020,154:112672. doi: 10.1016/j.indcrop.2020.112672
-
期刊类型引用(7)
1. 符梅,李桂花,罗文龙,骆善伟,郭巨先. 基于广泛靶向代谢组学的芋头代谢物和抗氧化酶活性的差异分析. 中国农学通报. 2025(06): 118-125 .  百度学术
百度学术
2. 邓煜,马正政,李正潇,侯佳. 基于代谢组学分析LED光质对山丹组培苗的生长代谢调控. 江苏农业科学. 2024(11): 150-159 .  百度学术
百度学术
3. 崔萌,段宝忠,程蕾,张满常,和福美,周萍. 基于近红外光谱结合化学计量学方法的山里红产地溯源分析. 中草药. 2024(14): 4897-4906 .  百度学术
百度学术
4. 申树林,李洪勋,黄兰,张昆鹏,朱勇,张龙,陈德慧,尹能文,曲存民,彭华伟,林叶春. 有机肥对烟叶次生代谢物影响分析. 安徽农业科学. 2024(20): 141-144+154 .  百度学术
百度学术
5. 贾延莎,张雪佳,谭宁华. 中药材中化学成分时空特异性的研究进展. 中草药. 2024(24): 8580-8588 .  百度学术
百度学术
6. 张凤,于翠,董朝霞,朱志贤,胡兴明,张成. 桑树代谢组学研究进展. 蚕业科学. 2024(06): 567-576 .  百度学术
百度学术
7. 武美卿,廖易,陆顺教,殷涵泰,余文刚,李崇晖. 基于广泛靶向代谢组学技术的不同花色秋石斛中花青素差异分析. 热带作物学报. 2023(11): 2167-2178 .  百度学术
百度学术
其他类型引用(8)




 下载:
下载: