Nitrogen sensing and root development in plants
-
摘要:
氮素是构成生物体的主要成分,也是植物体所需的重要营养元素。为应对土壤中氮素含量的变化,植物体需要通过一系列复杂的通路来精密调控根部构型,以达到最优的氮吸收效率。本文综述了植物根部接收氮素信号的感受器及其调控根系发育的具体途径,详细阐述了氮信号通路、植物激素以及其他分子间的交互对植物根系发育的协同调控,并展望了该领域研究的应用方向,以期为今后植物根系与氮素信号互作机制的探索提供新的研究思路。
Abstract:Nitrogen is a major component of living organisms and an essential nutrient for plant growth. To adapt to changes in nitrogen availability in the soil, plants employ complex signaling pathways to finely regulate root system architecture to optimize nitrogen uptake efficiency. This review focuses on the receptors involved in perceiving nitrogen signals in plant roots and the specific mechanisms governing root development. It also elaborates on the interplay between nitrogen signaling pathways, plant hormones, and other molecules that coordinately regulate root growth and development. This review aims to provide valuable insight into how plant roots perceive and respond to nitrogen signals.
-
Keywords:
- Nitrogen signal /
- Root development /
- Phytohormone
-
自然界中的生命体都有和外部环境进行物质和能量交换的能力,在这一过程中,机体通过感知外界环境信息,与自身内源信号相整合,从而调控自身生长发育和繁殖等一系列生命进程。对于大多数陆生植物而言,土壤除了能帮助其固着生长,也为其提供水分和养分。因此,土壤环境变化直接影响植物生长发育。土壤中的重要营养元素——氮,在土壤中的浓度、分布状况,是植物根系感知的重要信号。为适应土壤中复杂的氮信号环境,植物通过改变根系结构以实现最高的氮利用效率(Nitrogen-use efficiency,NUE)。在水稻(Oryza sativa L.)、拟南芥(Arabidopsis thaliana (L.) Heynh)、玉米(Zea mays L.)等植物中,普遍存在这种根系构型响应土壤氮素结构变化的现象,且大部分植物体中氮信号诱导的根系结构重塑途径与植物激素通路相互交联。本文总结了近年来植物根系感知及响应外源氮信号途径的研究进展以及亟待解决的问题,并展望了该领域研究的应用方向,以期为今后植物根系与土壤氮信号互作机制的研究提供理论基础。
1. 植物根际氮信号的感知
在自然条件下,为应对不同土壤条件,植物需要不断优化根系构型,氮供应不足或分布不均会诱导植物根觅食反应(Foraging response)[1],通过增大整体或局部的根表面积来增强对氮的吸收。而高氮环境下,由于氮素营养充足,植物则倾向于削弱根部生长,将更多营养转运到地上部[2, 3]。硝酸盐和铵盐是植物利用的主要无机氮源,其中硝酸盐是大多数生长于富氧土壤中植物的主要氮源,铵盐是生长在淹水或酸性土壤条件下植物的主要氮源[4]。
在土壤环境中,硝酸根离子浓度存在范围较大的波动,为适应这一自然现象,高等植物体内演化出两套转运系统,分别是适应低硝酸盐浓度(<0.5 mmol/L)的高亲和性转运系统和适应高硝酸盐浓度(>0.5 mmol/L)的低亲和性转运系统。共涉及到4类已知的硝酸盐转运家族[5-7],分别为氯离子通道家族(CLC)、硝酸盐转运体2家族(NRT2)、硝酸盐转运体1/小肽转运体家族NPF (NRT1/PTR)和S-型阴离子通道及其同系物(SLAC/SLAH)[8]。其中,在根部表达量较高且参与根系对土壤氮信号感知的主要为NPF家族和NRT2家族的成员(表1)。拟南芥中,AtNRT1.1是NPF家族的重要成员,也是一种双亲和转运体,在土壤氮水平改变时,AtNRT1.1的101位苏氨酸残基会在磷酸化和去磷酸化两种状态间转变,以此来改变其对硝酸盐的转运亲和力。有趣的是,该基因的T-DNA插入突变体chl1-5表现为硝酸盐信号严重不敏感和硝酸盐摄取缺陷,而点突变转基因材料chl1-9仅表现硝酸盐摄取障碍,这暗示硝酸盐摄取与下游氮信号响应过程可能相互独立[9]。水稻中OsNPF2.2、2.4、7.2、8.9都属于NPF家族的硝酸盐低亲和性转运蛋白,其中OsNPF2.2、OsNPF2.4依赖于跨膜pH梯度转运硝酸根离子,主要表达于木质部薄壁细胞的细胞膜,参与硝酸根离子在木质部的装载,这暗示它们可能参与硝酸盐向地上部的转运,帮助完成植物体内氮素营养的再分配[10, 11]。水稻中,在根组织中大量表达的OsNRT1.1b作为同属于NPF家族的双亲和性转运体,也可以调控硝酸盐的摄取以及根部向地上部的运输[12]。OsNRT2家族的OsNRT2.1和OsNRT2.2作为高亲和力转运体, 在低硝酸盐条件下与伴侣蛋白OsNAR2.1共同作用调控硝酸盐的吸收,同家族的OsNRT2.3a作为低亲和力转运蛋白,主要负责硝酸盐从地下部到地上部的转运[13]。
表 1 拟南芥和水稻根中表达的氮转运蛋白Table 1. N transport proteins expressed in Arabidopsis thaliana and Oryza sativa roots转运蛋白
Transport protein表达区域
Expression pattern主要功能
Description参考文献
ReferencesAtNRT1.1 根、保卫细胞 硝酸根吸收,生长素累积 [16] AtNRT1.2 根 根部硝酸盐吸收 [17] AtNRT1.5 根 硝酸根地下到地上部的转运 [18] AtNRT1.8 根、地上部 木质部硝酸盐转运 [18] AtNRT1.9 根部伴胞 硝酸根木质部到韧皮部的转运 [19] AtNRT1.11 根茎韧皮部 木质部硝酸盐转运 [19] AtNRT1.12 根茎韧皮部 木质部硝酸盐转运 [19] AtNRT2.1 根表皮及皮层细胞 硝酸盐吸收 [20] AtNRT2.2 根 硝酸盐的吸收和转运 [21] AtNRT2.3 根、茎 硝酸盐转运 [21] AtNRT2.4 根部表皮质膜 硝酸盐转运 [23] AtNRT2.5 根表皮及皮层细胞、根毛 表达受硝酸盐抑制 [22] AtNRT2.6 根、茎 硝酸盐转运 [21] AtAMT1;1 根表皮及皮层细胞 铵根离子吸收 [23] AtAMT1;2 根皮层根茎内皮层细胞 铵根离子从质外体向维管束转运 [23] AtAMT1;3 根表皮及皮层细胞 铵根离子吸收 [23] AtAMT2;1 根茎木质部 铵根离子地下到地上部的转运 [23] OsNRT1.1b 根表皮、根毛、微管组织、叶鞘、叶片和茎 双亲和力硝酸根转运蛋白 [12] OsNPF2.4 根表皮、木质部薄壁组织、
韧皮部伴细胞、 叶片韧皮部低亲和力硝酸根转运蛋白,
硝酸根地下到地上部的转运[11] OsNPF7.2 根系的厚壁组织、皮层和中柱 低亲和力硝酸根转运蛋白 [24] OsNPF7.9 根系、叶片和根茎结合处 硝酸根地下到地上部的转运 [25] OsNPF8.9 根系的外层、根表皮和根毛 低亲和力硝酸根转运蛋白 [26] OsNRT2.1 根、地上部 高亲和力硝酸根转运蛋白 [13] OsNRT2.2 根 高亲和力转运蛋白 [13] OsNRT2.3a 根部的微管组织和根颈 低亲和力硝酸根转运蛋白,硝酸根地下到地上部的转运 [13] OsNRT2.3b 微管组织、根系和地上部韧皮部 高亲和硝酸根转运蛋白,能够感知细胞溶质内pH的变化 [13] OsNRT2.4 侧根原基和地上部微管组织 假定的高亲和硝酸根转运蛋白 [13] OsAMT1.1 根表皮、微管组织、根颈、维管束、叶肉细胞 铵根离子地下到地上部的转运 [27] OsAMT1.2 微管组织、初生根根尖的内皮层和中柱鞘 铵根离子转运蛋白 [15] OsAMT1.3 根系维管束组织和侧根原基 铵根离子转运蛋白 [15] OsAMT2.1 根系、地上部、叶片、叶鞘 低亲和铵根离子转运蛋白 [28] 植物吸收利用的另一无机氮源,即铵盐,同样能够调控植物根系形态发育。与局部硝酸盐供应引起的根伸长作用不同,局部铵盐供应会诱导植物产生高度分支短根的表型。植物中,根据其演化特征,铵转运蛋白被分为AMT1和AMT2两类(表1)。拟南芥AMT家族共包含6个成员,其中AtAMT1;1、AtAMT1;2、AtAMT1;3和AtAMT2;1大量表达于根部表皮细胞,且被认为广泛参与铵根离子依赖的侧根诱导过程[14]。水稻中的AMT1和AMT2亚家族分别包含4个和8个成员,其中OsAMT1.3和OsAMT1.2在根部有表达,有研究认为它们分别承担根部铵感受器和铵转运体的职责,此外过表达OsAMT1.3也会上调OsAMT1.2的表达,同时OsAMT1.2过表达植株也具有根系体量增大和根分支增多这样的优良根表型[15]。有趣的是,与水稻中AMT1亚家族铵转运蛋白受铵态氮诱导上调的情况恰恰相反,拟南芥中AMT1s在铵态氮缺乏时上调,但在铵根充足的条件下表达下调[15-28]。
2. 氮信号调控植物根系构型
2.1 氮信号通过激素途径调控植物根系发育
自然环境下,植物体根系为适应土壤中的复杂氮环境,形成特异的根系构型,拟南芥根系在轻微缺氮环境下,为探寻土壤养分,侧根及主根长增加,侧根数目增多,在重度缺氮环境下,为分配更多营养资源到地上部,根系整体发育凝滞[2, 3]。研究发现,多种植物激素参与氮信号调节侧根起始,主侧根伸长等多个生物学过程[29]。随着这一领域研究的深入,氮信号响应途径与植物激素通路的串扰逐渐明晰,氮信号可以调节多种激素的生物合成与运输,植物激素也参与外源氮信号的感知以及氮素代谢。
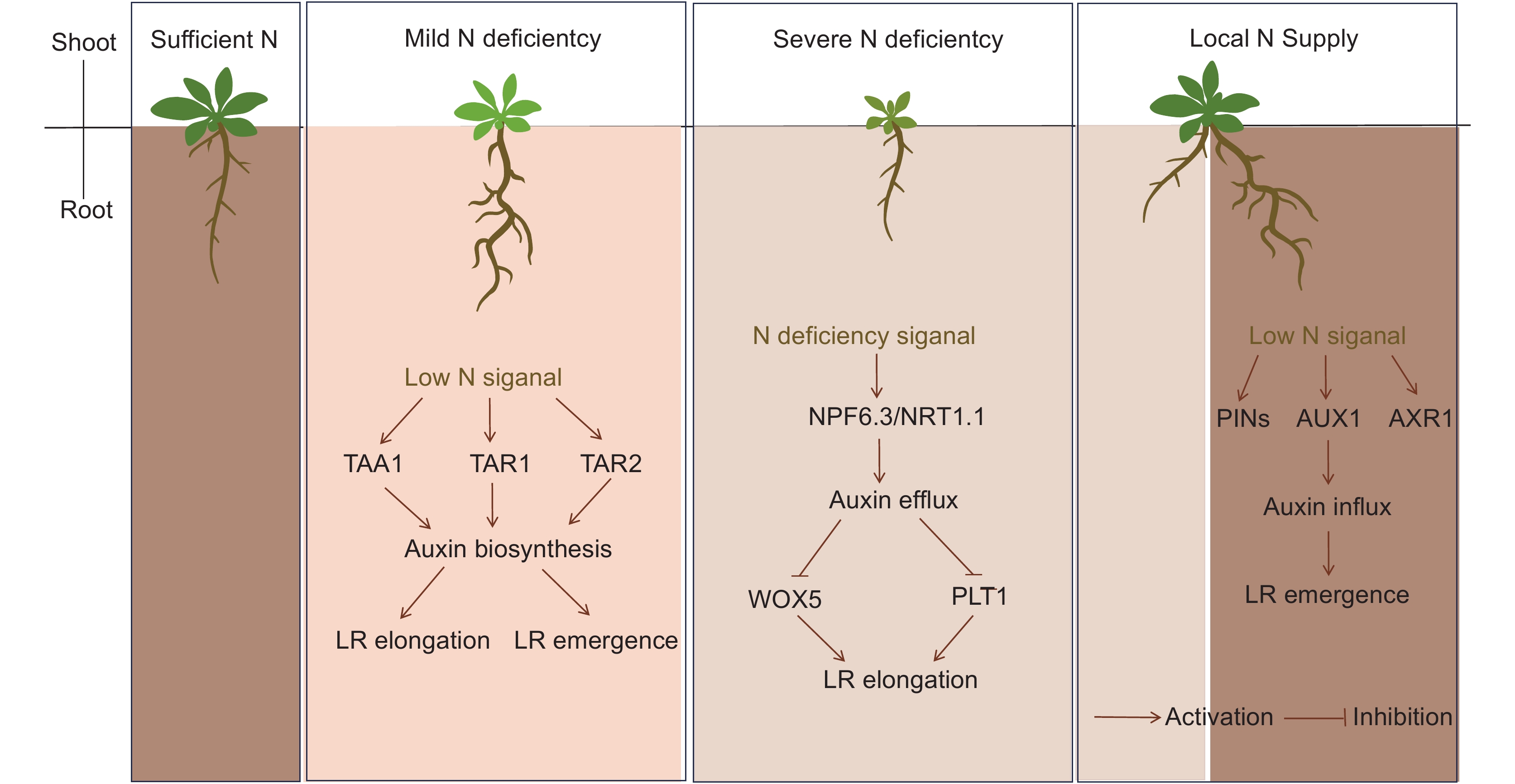
生长素(Auxin)是首个被发现可以受外源氮信号调控的植物激素[30],氮信号通过调控Auxin的合成转运控制局部生长素流量,进而精细调控植物根系构型(图1)。研究证明,拟南芥中Auxin合成基因TAA1、TAR1、TAR2[31]以及YUC3、5、7、8[32]受低氮诱导在根尖表达,调控侧根的伸长和数目。在小麦(Triticum aestivum L.)中,TAR2同样也受低氮诱导调控侧根分支[33]。在植物根部,氮水平影响生长素的极性运输,多种Auxin载体表达受氮水平调节,进而调控下游生长发育过程,Auxin外排蛋白家族PINs在水稻根部局部氮供应的条件下调节侧根伸长[34],在拟南芥中PINs则参与硝酸盐诱导的主根伸长[35]。AUX1和AXR4则作为生长素内流载体,受到局部氮供应诱导表达,致使侧根区域Auxin流量大大增加,从而促进侧根原基的起始[3]。除了直接调节Auxin合成运输相关基因,NPF6.3/NRT1.1作为双亲和性硝态氮转运蛋白,在严重氮缺乏条件下,从侧根根尖转运Auxin,下调下游控制细胞伸长的WOX5、PLT1而抑制侧根伸长[36],类似的,玉米中PIN1、PIN9能够介导局部高硝酸盐条件下的Auxin再分配[37],水稻中OsPIN1b则在低硝酸盐条件下控制Auxin从茎到根的极性运输,促使植株主根伸长[38]。在低氮诱导水稻种子根伸长的过程中也涉及到细胞分裂素(Cytokinin,CK)合成基因OsIPTs的下调,细胞分裂素氧化酶基因OsCKXs的上调,根部CK含量降低,进一步调控下游基因,致使根分生组织细胞增殖和细胞伸长[39]。相反,足量的硝酸盐可以刺激根部CK浓度增高,并在木质部通过载体ABCG14将tZ类CK运往地上部,调控地上部发育[40-42]。
![]() 图 1 不同土壤环境下氮信号通过生长素途径调控拟南芥根系发育氮信号调控生长素生物合成基因(TAA1、TARs)和转运基因(AUXs、AXRs和PINs)调控根系生长素水平,进而调控不同土壤环境(轻度缺氮、重度缺氮和局部氮供给)下拟南芥根系发育。不同颜色代表土壤氮浓度,颜色越深,土壤氮素浓度越高。Figure 1. Nitrogen signaling regulates Arabidopsis thaliana root development through the auxin pathway under different soil environmentsNitrogen signaling regulates expression of auxin biosynthesis genes (TAA1 and TARs) and transport genes (AUXs, AXRs, and PINs) to control auxin levels in roots. This regulation impacts Arabidopsis thaliana root growth and development under various soil conditions (mild N deficiency, severe N deficiency, and local N supply). Colors represent different soil nitrogen concentrations, with darker colors indicating higher levels.
图 1 不同土壤环境下氮信号通过生长素途径调控拟南芥根系发育氮信号调控生长素生物合成基因(TAA1、TARs)和转运基因(AUXs、AXRs和PINs)调控根系生长素水平,进而调控不同土壤环境(轻度缺氮、重度缺氮和局部氮供给)下拟南芥根系发育。不同颜色代表土壤氮浓度,颜色越深,土壤氮素浓度越高。Figure 1. Nitrogen signaling regulates Arabidopsis thaliana root development through the auxin pathway under different soil environmentsNitrogen signaling regulates expression of auxin biosynthesis genes (TAA1 and TARs) and transport genes (AUXs, AXRs, and PINs) to control auxin levels in roots. This regulation impacts Arabidopsis thaliana root growth and development under various soil conditions (mild N deficiency, severe N deficiency, and local N supply). Colors represent different soil nitrogen concentrations, with darker colors indicating higher levels.生长素和细胞分裂素途径与氮信号通路的交汇串扰,使得外源氮信号能利用这些经典植物激素调控植物体外部形态和内在状态以适应复杂自然氮环境,特别是直接感知吸收土壤氮素的根部相关表型。然而,随着相关研究的深入,其他植物激素水平也被发现在一定程度上受到外源氮信号调控。例如在拟南芥中,脱落酸(Abscisic acid,ABA)合成突变体(aba1-1、2-3、2-4和3-2)[43]表现出硝酸盐敏感性缺陷。进一步研究发现,为维持根部分生组织活性,硝酸盐会作为信号分子调节根系内皮层和静止中心中ABA浓度,通过上调β-葡萄糖苷酶1(BG1),硝酸盐可以解除ABA-葡萄糖酯的聚合结构,进而释放具有生物活性的ABA[44, 45]。类似的,小麦中BG1也受硝酸盐诱导上调,促进根部ABA积累[45]。DELLA蛋白是GA信号通路中的核心负调控因子,参与植物应对多种逆境,也是调节植物生长发育所需的重要分子,广泛存在于拟南芥、水稻及其他植物中。在硝酸盐信号刺激下,DELLA蛋白降解,从而在硝酸盐充足条件下促进GA合成。在玉米中,低硝酸盐环境则会下调GA合成基因(ZmKS2、ZmKS4、ZmGA20ox1和ZmGA20ox4),上调其分解基因(ZmGA2ox10、ZmGA2ox5、ZmGA2ox6、ZmGA2ox7和ZmGA2ox12),降低地下部活性GA水平[46]。此外,另一项研究表明,低氮胁迫下,高亲和力转运体基因NRT2.1表达上调,同时乙烯(Ethylene,ETH)信号通路的关键基因CTR1、EIN3和EIL1的上调,快速释放大量乙烯,而乙烯可以下调NRT2.1的表达量,从而形成低氮条件下涉及乙烯释放量和NRT2.1表达量的一个负反馈调节环[47]。拟南芥响应低硝酸盐条件时,类钙调蛋白CML38与PEPR2相互作用,也可以通过下游油菜素甾醇(Brassinosteroids,BRs)途径负调控根的生长[48, 49]。根据前人的研究,低硝酸盐条件下,根系中茉莉酸(Jasmonic acid,JA)水平极大地降低,而水杨酸(Salicylic acid,SA)水平升高[50]。此外,类似于NPF6.3/NRT1.1介导的生长素/氮信号通路串扰,植物体内NPF4.1、4.2、4.5和4.6具有转运ABA的能力,而NPF3和2.10则能够转运GA[29]。然而,ABA和GA通路是否通过这些转运蛋白来完成和氮信号通路的交互尚不清楚。
2.2 氮信号通过非激素途径调控植物根系发育
植物根系周围往往存在多种形式的氮源,其中氨基酸和小肽形式的可溶有机氮,常作为生长在矿物质含量较低的温带地区植物的潜在氮源[51],同时也可以作为调控植物根发育的信号分子发挥作用。植物中探究氨基酸对根生长发育作用的研究相对较少,尽管在多种氨基酸存在的情况下根系生长会受到抑制,但只有谷氨酸能引起生长、根尖形态和根分枝的变化,即使是低浓度的谷氨酸(<50 mmol/L)也会显著抑制主根生长和刺激侧根突破而产生更短、更多的侧根[52, 53]。低氮环境下拟南芥根中会分泌能从木质部易位到茎的小肽CEPs,识别并结合受体CEPR1/2,产生的非分泌性多肽CEPD1及其同源物作为次生信号通过韧皮部运输到根[54, 55]。氮响应CLE- CLV1是一种控制侧根生长的重要机制,在严重氮缺乏环境下,防止侧根向氮缺乏区域扩张。当植物长时间处于低氮环境时,一组CLE小肽(CLE1、3、4和7)表达上调,抑制侧根原基的生长和突破,CLE信号受到其受体CLV1的反馈调节,clv1则表现出侧根原基向侧根的加速转变[56]。
近年来的研究表明,植物体内存在如microRNA、Ca2+等同时参与氮信号和根系生长的因子。植物体内生长发育过程中发挥重要功能的bHLH转录因子和生长素受体TIR1、AFB1、AFB2和AFB3可以被硝酸盐还原和同化代谢产物miR393降解[57]。水稻中与AtNRT1.1同源的5个基因(OsMADS23、25、27、57和61)中4个(OsMADS23、25、27和57)在根中表达,OsMADS25、27和57受到硝酸盐诱导表达[58],OsMADS23、27、57和61受到miR444调控。局部硝酸盐处理将刺激水稻侧根生长,而在miR444a过表达系中,这种反应缺失[59]。氮胁迫条件下,钙信号通路中的重要分子Ca2+传感器CBL7被诱导表达,与野生型相比,cbl7在严重氮缺乏条件下对根系生长的抑制效应更强,同时硝酸盐转运蛋白基因AtNRT2.5和AtNRT2.5表达量降低[60]。
3. 展望
植物根系具有可塑性以适应土壤环境变化。对于经济作物而言,在完成全面推进半矮化育种的第一次绿色革命后,提高产量的一大重点转变到提高氮利用效率,而其中的重要一环就是改良作物根系构型。NUE是一种衡量植物捕获和利用氮素能力的指标,可以简单定义为作物施用单位氮肥的产量。NUE由N吸收效率、N同化效率和N再活化效率组成,涉及N吸收、同化、转运和再活化等几个生物学过程和代谢过程[61]。
随着科学技术的发展,生物技术已经广泛应用于作物根系结构的改良和氮利用效率的提高。基因编辑结合全基因组分析是探索新的NUE靶点并明确其功能的主流手段。此外,基于突变体筛选的图位克隆也是挖掘有助于提高NUE的候选基因的重要策略。反向遗传学手段如T-DNA插入、Ds插入、RNAi和过表达以及新兴的CRISPR/Cas工具为阐明更多NUE基因功能,培育出新的作物品种,降低生产成本,减少环境污染提供了更多可能。基于图位克隆和QTL分析,OsGRF4是一个关键的NUE基因,调控水稻铵盐摄取,靶向OsGRF4的RNAi可以减少铵根摄取,ACTIN1驱动的OsGRF4过表达则会增加铵根摄取。BR合成关键基因AtDWF1通过GWAS被证明参与低氮条件下的根伸长,过表达AtDWF1显著促进植物的生长和氮的积累[29]。
植物中,尽管已经挖掘出一部分参与氮信号调控根系发育的潜在功能基因,但它们参与这一生物学过程的具体分子机制仍需探究,植物激素和氮信号对根系发育的交互调节机制也有待阐明。此外,植物根系外部环境,如土壤pH值[62]、温室效应[63]等对植物无机氮的吸收也具有一定影响,后续应重视这部分研究,更大限度地提高作物NUE,为改善生态环境提高作物品质提供理论支持。在系统生物学背景下协同调控植物生长、光合作用和氮代谢是研究未来农业持续性发展的重要方向。
-
图 1 不同土壤环境下氮信号通过生长素途径调控拟南芥根系发育
氮信号调控生长素生物合成基因(TAA1、TARs)和转运基因(AUXs、AXRs和PINs)调控根系生长素水平,进而调控不同土壤环境(轻度缺氮、重度缺氮和局部氮供给)下拟南芥根系发育。不同颜色代表土壤氮浓度,颜色越深,土壤氮素浓度越高。
Figure 1. Nitrogen signaling regulates Arabidopsis thaliana root development through the auxin pathway under different soil environments
Nitrogen signaling regulates expression of auxin biosynthesis genes (TAA1 and TARs) and transport genes (AUXs, AXRs, and PINs) to control auxin levels in roots. This regulation impacts Arabidopsis thaliana root growth and development under various soil conditions (mild N deficiency, severe N deficiency, and local N supply). Colors represent different soil nitrogen concentrations, with darker colors indicating higher levels.
表 1 拟南芥和水稻根中表达的氮转运蛋白
Table 1 N transport proteins expressed in Arabidopsis thaliana and Oryza sativa roots
转运蛋白
Transport protein表达区域
Expression pattern主要功能
Description参考文献
ReferencesAtNRT1.1 根、保卫细胞 硝酸根吸收,生长素累积 [16] AtNRT1.2 根 根部硝酸盐吸收 [17] AtNRT1.5 根 硝酸根地下到地上部的转运 [18] AtNRT1.8 根、地上部 木质部硝酸盐转运 [18] AtNRT1.9 根部伴胞 硝酸根木质部到韧皮部的转运 [19] AtNRT1.11 根茎韧皮部 木质部硝酸盐转运 [19] AtNRT1.12 根茎韧皮部 木质部硝酸盐转运 [19] AtNRT2.1 根表皮及皮层细胞 硝酸盐吸收 [20] AtNRT2.2 根 硝酸盐的吸收和转运 [21] AtNRT2.3 根、茎 硝酸盐转运 [21] AtNRT2.4 根部表皮质膜 硝酸盐转运 [23] AtNRT2.5 根表皮及皮层细胞、根毛 表达受硝酸盐抑制 [22] AtNRT2.6 根、茎 硝酸盐转运 [21] AtAMT1;1 根表皮及皮层细胞 铵根离子吸收 [23] AtAMT1;2 根皮层根茎内皮层细胞 铵根离子从质外体向维管束转运 [23] AtAMT1;3 根表皮及皮层细胞 铵根离子吸收 [23] AtAMT2;1 根茎木质部 铵根离子地下到地上部的转运 [23] OsNRT1.1b 根表皮、根毛、微管组织、叶鞘、叶片和茎 双亲和力硝酸根转运蛋白 [12] OsNPF2.4 根表皮、木质部薄壁组织、
韧皮部伴细胞、 叶片韧皮部低亲和力硝酸根转运蛋白,
硝酸根地下到地上部的转运[11] OsNPF7.2 根系的厚壁组织、皮层和中柱 低亲和力硝酸根转运蛋白 [24] OsNPF7.9 根系、叶片和根茎结合处 硝酸根地下到地上部的转运 [25] OsNPF8.9 根系的外层、根表皮和根毛 低亲和力硝酸根转运蛋白 [26] OsNRT2.1 根、地上部 高亲和力硝酸根转运蛋白 [13] OsNRT2.2 根 高亲和力转运蛋白 [13] OsNRT2.3a 根部的微管组织和根颈 低亲和力硝酸根转运蛋白,硝酸根地下到地上部的转运 [13] OsNRT2.3b 微管组织、根系和地上部韧皮部 高亲和硝酸根转运蛋白,能够感知细胞溶质内pH的变化 [13] OsNRT2.4 侧根原基和地上部微管组织 假定的高亲和硝酸根转运蛋白 [13] OsAMT1.1 根表皮、微管组织、根颈、维管束、叶肉细胞 铵根离子地下到地上部的转运 [27] OsAMT1.2 微管组织、初生根根尖的内皮层和中柱鞘 铵根离子转运蛋白 [15] OsAMT1.3 根系维管束组织和侧根原基 铵根离子转运蛋白 [15] OsAMT2.1 根系、地上部、叶片、叶鞘 低亲和铵根离子转运蛋白 [28] -
[1] Xuan W,Beeckman T,Xu GH. Plant nitrogen nutrition:sensing and signaling[J]. Curr Opin Plant Biol,2017,39:57−65. doi: 10.1016/j.pbi.2017.05.010
[2] Gruber BD,Giehl RFH,Friedel S,von Wirén N. Plasticity of the Arabidopsis root system under nutrient deficiencies[J]. Plant Physiol,2013,163(1):161−179. doi: 10.1104/pp.113.218453
[3] Zhang HM,Jennings A,Barlow PW,Forde BG. Dual pathways for regulation of root branching by nitrate[J]. Proc Natl Acad Sci USA,1999,96(11):6529−6534. doi: 10.1073/pnas.96.11.6529
[4] Shen TC. The induction of nitrate reductase and the preferential assimilation of ammonium in germinating rice seedlings[J]. Plant Physiol,1969,44(11):1650−1655. doi: 10.1104/pp.44.11.1650
[5] Crawford NM,Glass ADM. Molecular and physiological aspects of nitrate uptake in plants[J]. Trends Plant Sci,1998,3(10):389−395. doi: 10.1016/S1360-1385(98)01311-9
[6] Forde BG. Nitrate transporters in plants:structure,function and regulation[J]. Biochim Biophys Acta,2000,1465(1-2):219−235. doi: 10.1016/S0005-2736(00)00140-1
[7] Miller AJ,Fan X,Orsel M,Smith SJ,Wells DM. Nitrate transport and signalling[J]. J Exp Bot,2007,58(9):2297−2306. doi: 10.1093/jxb/erm066
[8] Krapp A,David LC,Chardin C,Girin T,Marmagne A,et al. Nitrate transport and signalling in Arabidopsis[J]. J Exp Bot,2014,65(3):789−798. doi: 10.1093/jxb/eru001
[9] Ho CH,Lin SH,Hu HC,Tsay YF. CHL1 functions as a nitrate sensor in plants[J]. Cell,2009,138(6):1184−1194. doi: 10.1016/j.cell.2009.07.004
[10] Li YG,Ouyang J,Wang YY,Hu R,Xia KF,et al. Disruption of the rice nitrate transporter OsNPF2.2 hinders root-to-shoot nitrate transport and vascular development[J]. Sci Rep,2015,5:9635. doi: 10.1038/srep09635
[11] Xia XD,Fan XR,Wei J,Feng HM,Qu HY,et al. Rice nitrate transporter OsNPF2.4 functions in low-affinity acquisition and long-distance transport[J]. J Exp Bot,2015,66(1):317−331. doi: 10.1093/jxb/eru425
[12] Hu B,Wang W,Ou SJ,Tang JY,Li H, et al. Variation in NRT1.1B contributes to nitrate-use divergence between rice subspecies[J]. Nat Genet,2015,47(7):834−838.
[13] Feng HM,Yan M,Fan XR,Li BZ,Shen QR,et al. Spatial expression and regulation of rice high-affinity nitrate transporters by nitrogen and carbon status[J]. J Exp Bot,2011,62(7):2319−2332. doi: 10.1093/jxb/erq403
[14] Liu Y,von Wirén N. Ammonium as a signal for physiological and morphological responses in plants[J]. J Exp Bot,2017,68(10):2581−2592. doi: 10.1093/jxb/erx086
[15] Sonoda Y,Ikeda A,Saiki S,Yamaya T,Yamaguchi J. Feedback regulation of the ammonium transporter gene family AMT1 by glutamine in rice[J]. Plant Cell Physiol,2003,44(12):1396−1402. doi: 10.1093/pcp/pcg169
[16] Fan XR,Naz M,Fan XR,Xuan W,Miller AJ,Xu GH. Plant nitrate transporters:from gene function to application[J]. J Exp Bot,2017,68(10):2463−2475. doi: 10.1093/jxb/erx011
[17] Li GW,Tillard P,Gojon A,Christophe M. Dual regulation of root hydraulic conductivity and plasma membrane aquaporins by plant nitrate accumulation and high-affinity nitrate transporter NRT2.1[J]. Plant Cell Physiol,2016,57(4):733−742. doi: 10.1093/pcp/pcw022
[18] Han YL,Song HX,Liao Q,Yu Y,Jian SF,et al. Nitrogen use efficiency is mediated by vacuolar nitrate sequestration capacity in roots of Brassica napus[J]. Plant Physiol,2016,170(3):1684−1698. doi: 10.1104/pp.15.01377
[19] Von Wittgenstein NJ,Le CH,Hawkins BJ,Ehlting J. Evolutionary classification of ammonium,nitrate,and peptide transporters in land plants[J]. BMC Evol Biol,2014,14:11. doi: 10.1186/1471-2148-14-11
[20] Ohkubo Y,Tanaka M,Tabata R,Ogawa-Ohnishi M,Matsubayashi Y. Shoot-to-root mobile polypeptides involved in systemic regulation of nitrogen acquisition[J]. Nat Plants,2017,3:17029. doi: 10.1038/nplants.2017.29
[21] Kotur Z,Mackenzie N,Ramesh S,Tyerman SD,Kaiser BN,Glass ADM. Nitrate transport capacity of the Arabidopsis thaliana NRT2 family members and their interactions with AtNAR2.1[J]. New Phytol,2012,194(3):724−731. doi: 10.1111/j.1469-8137.2012.04094.x
[22] Lezhneva L,Kiba T,Feria-Bourrellier AB,Lafouge F,Boutet-Mercey S,et al. The Arabidopsis nitrate transporter NRT2.5 plays a role in nitrate acquisition and remobilization in nitrogen-starved plants[J]. Plant J,2014,80(2):230−241. doi: 10.1111/tpj.12626
[23] Yuan LX,Loqué D,Kojima S,Rauch S,Ishiyama K,et al. The organization of high-affinity ammonium uptake in Arabidopsis roots depends on the spatial arrangement and biochemical properties of AMT1-type transporters[J]. Plant Cell,2007,19(8):2636−2652. doi: 10.1105/tpc.107.052134
[24] Hu R,Qiu DY,Chen Y,Miller AJ,Fan XR, et al. Knock-down of a tonoplast localized low-affinity nitrate transporter OsNPF7. 2 affects rice growth under high nitrate supply[J]. Front Plant Sci,2016,7:1529.
[25] 冯慧敏,陆宏,王汉卿,李昕玥. 水稻硝酸盐转运蛋白基因OsNPF7. 9在氮素积累和转运中的功能研究[J]. 中国水稻科学,2017,31(5):457−464. Feng HM,Lu H,Wang HQ,Li XY. Function analyses of rice nitrate transporter gene OsNPF7.9 in nitrogen accumulation and transport[J]. Chinese Journal of Rice Science,2017,31(5):457−464.
[26] Léran S,Varala K,Boyer JC,Chiurazzi M,Crawford N,et al. A unified nomenclature of NITRATE TRANSPORTER 1/PEPTIDE TRANSPORTER family members in plants[J]. Trends Plant Sci,2014,19:5−9. doi: 10.1016/j.tplants.2013.08.008
[27] Li C,Tang Z,Wei J,Qu HY,Xie YJ,Xu GH. The OsAMT1.1 gene functions in ammonium uptake and ammonium-potassium homeostasis over low and high ammonium concentration ranges[J]. J Genet Genomics,2016,43(11):639−649.
[28] Suenaga A,Moriya K,Sonoda Y,Ikeda A,von Wirén N,et al. Constitutive expression of a novel-type ammonium transporter OsAMT2 in rice plants[J]. Plant Cell Physiol,2003,44(2):206−211. doi: 10.1093/pcp/pcg017
[29] Xing JP,Cao XC,Zhang MC,Wei X,Zhang J,Wan XY. Plant nitrogen availability and crosstalk with phytohormones signallings and their biotechnology breeding application in crops[J]. Plant Biotechnol J,2023,21(7):1320−1342. doi: 10.1111/pbi.13971
[30] Avery GS,Burkholder PR,Creighton HB. Nutrient deficiencies and growth hormone concentration in helianthus and nicotiana[J]. Am J Bot,1937,24(8):553−557. doi: 10.1002/j.1537-2197.1937.tb09146.x
[31] Ma WY,Li JJ,Qu BY,He X,Zhao XQ,et al. Auxin biosynthetic gene TAR2 is involved in low nitrogen-mediated reprogramming of root architecture in Arabidopsis[J]. Plant J,2014,78(1):70−79. doi: 10.1111/tpj.12448
[32] Jia ZT,Giehl RFH,von Wirén N. Local auxin biosynthesis acts downstream of brassinosteroids to trigger root foraging for nitrogen[J]. Nat Commun,2021,12(1):5437. doi: 10.1038/s41467-021-25250-x
[33] Shao A,Ma WY,Zhao XQ,Hu MY,He X, et al. The auxin biosynthetic TRYPTOPHAN AMINOTRANSFERASE RELATED TaTAR2.1-3A increases grain yield of wheat[J]. Plant Physiol,2017,174(4):2274−2288.
[34] Sun HW,Bi Y,Tao JY,Huang SJ,Hou MM,et al. Strigolactones are required for nitric oxide to induce root elongation in response to nitrogen and phosphate deficiencies in rice[J]. Plant Cell Environ,2016,39(7):1473−1484. doi: 10.1111/pce.12709
[35] Caba JM,Centeno ML,Fernández B,Gresshoff PM,Ligero F. Inoculation and nitrate alter phytohormone levels in soybean roots:differences between a supernodulating mutant and the wild type[J]. Planta,2000,211(1):98−104. doi: 10.1007/s004250000265
[36] Krouk G,Lacombe B,Bielach A,Perrine-Walker F,Malinska K,et al. Nitrate-regulated auxin transport by NRT1.1 defines a mechanism for nutrient sensing in plants[J]. Dev Cell,2010,18(6):927−937. doi: 10.1016/j.devcel.2010.05.008
[37] Yu P,Eggert K,von Wirén N,Li CJ,Hochholdinger F. Cell type-specific gene expression analyses by RNA sequencing reveal local high nitrate-triggered lateral root initiation in shoot-borne roots of maize by modulating auxin-related cell cycle regulation[J]. Plant Physiol,2015,169(1):690−704. doi: 10.1104/pp.15.00888
[38] Sun HW,Tao JY,Bi Y,Hou MM,Lou JJ, et al. OsPIN1b is involved in rice seminal root elongation by regulating root apical meristem activity in response to low nitrogen and phosphate[J]. Sci Rep,2018,8(1):13014.
[39] Wang Q,Zhu YC,Zou X,Li FF,Zhang JL,et al. Nitrogen deficiency-induced decrease in cytokinins content promotes rice seminal root growth by promoting root meristem cell proliferation and cell elongation[J]. Cells,2020,9(4):916. doi: 10.3390/cells9040916
[40] Ko D,Kang J,Kiba T,Park J,Kojima M, et al. Arabidopsis ABCG14 is essential for the root-to-shoot translocation of cytokinin[J]. Proc Natl Acad Sci USA,2014,111(19):7150−7155.
[41] Poitout A,Crabos A,Petřík I,Novák O,Krouk G,et al. Responses to systemic nitrogen signaling in Arabidopsis roots involve trans-zeatin in shoots[J]. Plant Cell,2018,30(6):1243−1257. doi: 10.1105/tpc.18.00011
[42] Zhang KW,Novak O,Wei ZY,Gou MY,Zhang XB, et al. Arabidopsis ABCG14 protein controls the acropetal translocation of root-synthesized cytokinins[J]. Nat Commun,2014,5:3274.
[43] Signora L,de Smet I,Foyer CH,Zhang HM. ABA plays a central role in mediating the regulatory effects of nitrate on root branching in Arabidopsis[J]. Plant J,2001,28(6):655−662. doi: 10.1046/j.1365-313x.2001.01185.x
[44] Ondzighi-Assoume CA,Chakraborty S,Harris JM. Environmental nitrate stimulates abscisic acid accumulation in Arabidopsis root tips by releasing it from inactive stores[J]. Plant Cell,2016,28(3):729−745. doi: 10.1105/tpc.15.00946
[45] Wang M,Zhang PL,Liu Q,Li GJ,Di DW,et al. TaANR1-TaBG1 and TaWabi5-TaNRT2s/NARs link ABA metabolism and nitrate acquisition in wheat roots[J]. Plant Physiol,2020,182(3):1440−1453. doi: 10.1104/pp.19.01482
[46] Wang YB,Yao QQ,Zhang YS,Zhang YX,Xing JP,et al. The role of gibberellins in regulation of nitrogen uptake and physiological traits in maize responding to nitrogen availability[J]. Int J Mol Sci,2020,21(5):1824. doi: 10.3390/ijms21051824
[47] Zheng DC,Han X,An Y,Guo HW,Xia XL,Yin WL. The nitrate transporter NRT2.1 functions in the ethylene response to nitrate deficiency in Arabidopsis[J]. Plant Cell Environ,2013,36(7):1328−1337. doi: 10.1111/pce.12062
[48] Lokdarshi A,Conner WC,McClintock C,Li T,Roberts DM. Arabidopsis CML38,a calcium sensor that localizes to ribonucleoprotein complexes under hypoxia stress[J]. Plant Physiol,2016,170(2):1046−1059. doi: 10.1104/pp.15.01407
[49] Song XY,Li JF,Lyu M,Kong XZ,Hu S,et al. CALMODULIN-LIKE-38 and PEP1 RECEPTOR 2 integrate nitrate and brassinosteroid signals to regulate root growth[J]. Plant Physiol,2021,187(3):1779−1794. doi: 10.1093/plphys/kiab323
[50] Conesa CM,Saez A,Navarro-Neila S,de Lorenzo L,Hunt AG,et al. Alternative polyadenylation and salicylic acid modulate root responses to low nitrogen availability[J]. Plants,2020,9(2):251. doi: 10.3390/plants9020251
[51] Tegeder M,Rentsch D. Uptake and partitioning of amino acids and peptides[J]. Mol Plant,2010,3(6):997−1011. doi: 10.1093/mp/ssq047
[52] Forde BG. Glutamate signalling in roots[J]. J Exp Bot,2014,65(3):779−787. doi: 10.1093/jxb/ert335
[53] Walch-Liu P,Liu LH,Remans T,Tester M,Forde BG. Evidence that L-glutamate can act as an exogenous signal to modulate root growth and branching in Arabidopsis thaliana[J]. Plant Cell Physiol,2006,47(8):1045−1057. doi: 10.1093/pcp/pcj075
[54] Taleski M,Chapman K,Novák O,Schmülling T,Frank M,Djordjevic MA. CEP peptide and cytokinin pathways converge on CEPD glutaredoxins to inhibit root growth[J]. Nat Commun,2023,14(1):1683. doi: 10.1038/s41467-023-37282-6
[55] Taleski M,Imin N,Djordjevic MA. CEP peptide hormones:key players in orchestrating nitrogen-demand signalling,root nodulation,and lateral root development[J]. J Exp Bot,2018,69(8):1829−1836. doi: 10.1093/jxb/ery037
[56] Araya T,Miyamoto M,Wibowo J,Suzuki A,Kojima S,et al. CLE-CLAVATA1 peptide-receptor signaling module regulates the expansion of plant root systems in a nitrogen-dependent manner[J]. Proc Natl Acad Sci USA,2014,111(5):2029−2034. doi: 10.1073/pnas.1319953111
[57] Vidal EA,Araus V,Lu C,Parry G,Green PJ,et al. Nitrate-responsive miR393/AFB3 regulatory module controls root system architecture in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA,2010,107(9):4477−4482. doi: 10.1073/pnas.0909571107
[58] Puig J,Meynard D,Khong GN,Pauluzzi G,Guiderdoni E,Gantet P. Analysis of the expression of the AGL17-like clade of MADS-box transcription factors in rice[J]. Gene Expr Patterns,2013,13(5-6):160−170. doi: 10.1016/j.gep.2013.02.004
[59] Yan YS,Wang HC,Hamera S,Chen XY,Fang RX. miR444a has multiple functions in the rice nitrate-signaling pathway[J]. Plant J,2014,78(1):44−55. doi: 10.1111/tpj.12446
[60] Ma Q,Tang RJ,Zheng XJ,Wang SM,Luan S. The calcium sensor CBL7 modulates plant responses to low nitrate in Arabidopsis[J]. Biochem Biophys Res Commun,2015,468(1-2):59−65. doi: 10.1016/j.bbrc.2015.10.164
[61] Han X,Wu K,Fu XD,Liu Q. Improving coordination of plant growth and nitrogen metabolism for sustainable agriculture[J]. aBIOTECH,2020,1(4):255−275. doi: 10.1007/s42994-020-00027-w
[62] Fan XR,Tang Z,Tan YW,Zhang Y,Luo BB,et al. Overexpression of a pH-sensitive nitrate transporter in rice increases crop yields[J]. Proc Natl Acad Sci USA,2016,113(26):7118−7123. doi: 10.1073/pnas.1525184113
[63] Rennenberg H,Wildhagen H,Ehlting B. Nitrogen nutrition of poplar trees[J]. Plant Biol,2010,12(2):275−291. doi: 10.1111/j.1438-8677.2009.00309.x
-
期刊类型引用(1)
1. 陈鑫悦,姚晓琴,王宝庆,陈虹. 砾石戈壁区核桃园土壤理化性质及其对核桃产质量的影响. 贵州农业科学. 2025(02): 73-82 .  百度学术
百度学术
其他类型引用(1)



 下载:
下载: