A new record of a filamentous cyanobacterial species in China: Oculatella ucrainica Oxana Vinogradova & Tatiana Mikhailyuk
-
摘要:
乌克兰细点丝藻(Oculatella ucrainica Oxana Vinogradova & Tatiana Mikhailyuk)是2017年从土壤结皮与海岸边分离出来并描述为新种的耐旱陆生物种。本研究从我国山西省太原师范学院校园的潮湿土壤和贵州省花溪公园的湿润墙面分离出两株丝状蓝藻,形态上与乌克兰细点丝藻相似,均在丝状体顶端细胞中含有橘色颗粒;16S rRNA基因序列显示,这两株蓝藻与乌克兰细点丝藻的相似性均在99%以上,且在系统发育树中聚为一支,有较高的自展值。此外,这两株丝状蓝藻的ITS二级结构与乌克兰细点丝藻差异较小,进一步表明它们均属于乌克兰细点丝藻,这是我国首次报道乌克兰细点丝藻新记录种。
Abstract:A newly recorded drought-tolerant terrestrial cyanobacterium, Oculatella ucrainica Oxana Vinogradova & Tatiana Mikhailyuk, originally isolated from soil crusts and coastal areas in 2017, is reported in China for the first time. In this study, two filamentous cyanobacteria were isolated from moist soil at Taiyuan Normal University, Shanxi Province, and damp walls in Huaxi Park, Guizhou Province. These strains displayed morphological similarities to O. ucrainica, both featuring orange spots on the apical cells of their trichomes. Phylogenetic analysis based on 16S rRNA gene sequences demonstrated high similarity (above 99%) between the two isolates and O. ucrainica, with both strains clustering on a single branch with high bootstrap support. Furthermore, the secondary structure of these cyanobacteria closely resembled that of O. ucrainica, further corroborating their identification as members of this species. This study represents the first report of O. ucrainica in China.
-
Keywords:
- Oculatella ucrainica /
- Filamentous cyanobacteria /
- New record species /
- Phylogeny /
- Classify
-
由于物种的形态多样性,长期以来描述并区分它们的唯一方法是细胞形态学[1]。作为进化出多细胞结构的原核生物之一,丝状蓝藻在形态特征和遗传发育方面具有多样性[2, 3]。传统的蓝藻分类和鉴定基于形态学方法,但仅依靠形态特征并不能对蓝藻进行准确分类[4, 5]。随着分子生物学和遗传学的发展,遗传、生态和生化等现代方法也应用到蓝藻的分类中[6, 7]。从Geitler在1925年提出的七目系统[8],到Komárek等[9]在2014年提出的八目系统,再到Strunecký等[10]提出的二十目系统,这些分类系统的更新都是为了建立更加合理的分类体系,为进一步对单系的蓝藻类群重修和整合提供了理论依据。因此,运用形态学、生态学和分子数据等多相方法来进行系统发育分析逐渐成为确定蓝藻分类地位的最佳方法。
细点丝藻(Oculatella)属于细点丝藻目细点丝藻科,由Zammit等[11]于2012年建立,目前包括14个种,模式种为O. subterranea Zammit, Billi & Albertano。该属在横壁和无色的鞘处略微收缩,细胞偶尔呈现紫红色,丝状体顶端细胞含有橘色颗粒,可能是一种对光敏感的物质。乌克兰细点丝藻(O. ucrainica O. Vinogradova & T. Mikhailyuk)是由Vinogradova等[12]在2017年从土壤结皮与海岸边分离出来并描述为新种的,该种属于耐旱陆生物种,且其丝状体更宽,藻丝伸长是鞘在丝状体上缩短所致,在系统发育分析中,乌克兰细点丝藻具有单独的进化枝,具有与其他物种不同的二级结构。
据报道,细点丝藻属最早从意大利罗马和马耳他拉巴特的潮湿土壤中分离得到[11],近几年在西班牙也发现了该属其他新种的踪迹[12],但目前该物种在我国还未见报道和描述。本文首次报道了采自我国太原师范学院校园潮湿土壤和贵阳市花溪公园湿润墙面的乌克兰细点丝藻,通过形态学、生态学和系统发育学等多相方法对其进行分类鉴定[13]。新记录种的报道增加了中国蓝藻物种多样性,可为蓝藻的进一步修订和分类提供科学依据。
1. 材料与方法
1.1 藻株的采集、分离与培养
两株丝状蓝藻分别采集于贵州省贵阳市花溪公园的湿润墙面和山西省太原师范学院校园的潮湿土壤表面,使用镊子和小刀进行采集,将样品装入收集瓶中,并记录采集地的经纬度、温度、海拔和pH值等详细信息(表1)。
表 1 采集地的详细信息Table 1. Details of the collection site藻株
Strain采集地
Collection site经纬度
Latitude and longitude海拔
Elevation / m温度
Temperature / ℃pH值
pH value生境
HabitatSXACC0051 山西省太原师范学院校园 26°26ˊ07″N,
106°39ˊ57″E808 16.3 7.59 潮湿土壤 SXACC0050 贵州省贵阳市花溪湿地公园 26°26ˊ07″N,
106°39ˊ57″E1 140 20.5 7.86 湿润墙面 将采集的样品进行富集培养,利用经典的毛细管分离法[14]分离纯化藻株。首先在倒置显微镜下挑取单根藻丝体,使用无菌水多次清洗,然后在含BG-11/CT培养基的24孔细胞培养板中进行培养。
当长出形态一致的藻丝后,转入三角瓶中扩大培养,放置在恒温光照培养箱中,保持光照强度2 000 Lx,温度(25±1)℃,12 h/12 h 的光暗循环[15],纯化后的藻种保存在太原师范学院淡水藻种库中,编号分别为SXACC0050和SXACC0051。
1.2 形态表征
使用Nikon Eclipse NI型光学显微镜[16]观察蓝藻的形态特征,借助NIS-Elements D 5.20软件拍摄藻种的显微图片,并测量细胞的尺寸,尺寸用“min-max”表示,每个样本至少包含30个藻丝体。拍摄的照片使用软件Illustrator CS5和Photoshop CC 2019 SP 20.0.0进行处理。
1.3 DNA 分离、PCR扩增与测序
将藻体研碎后,使用改进的CTAB法[17,18]提取细点丝藻基因组DNA,将提取的DNA放入−20 ℃冰箱中保存备用。选取16S rRNA和16S-23S rRNA基因序列进行系统发育分析,使用引物PA(5ˊ-AGAGTTTGATCCTGGCTCAG-3ˊ)和B23S[19](5ˊ-CTTCGCCTCTGTGTGCCTAGGT-3ˊ)以及F1[20](5ˊ-TTGATCCTGGCTCAGGATGA-3ˊ)和1492[21](5ˊ-GGTTACCTTGTTACGACTT-3ˊ)分别对SXACC0050和SXACC0051进行16S rRNA基因的扩增,引物322(5ˊ-CTCTGTGTGCCTAGGTATCC-3ˊ)和340[22](5ˊ-GGGGAATTTTCCGCAATGGG-3ˊ)用于扩增16S-23S ITS区域。PCR反应体系参考文献[23]报道。PCR循环在95 ℃下预变性3 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸50 s,进行35次循环[24],最后,在72 ℃下延伸5 min。用1%琼脂糖凝胶电泳法纯化PCR产物。
将纯化的PCR产物进行双向测序(北京六合华大基因科技有限公司)。测序后的序列提交到 NCBI(https://www.ncbi.nlm.nih.gov/)GenBank 数据库中。
1.4 基于16S rRNA序列的系统发育分析
将序列与NCBI数据库中的序列进行BLAST比对,选取并下载相似度较高和有代表性的序列。使用Bio Edit 7.0[25] 软件中的Clustal W进行多重序列比对,然后编辑并切除未对齐的两端序列。运用软件MEGA11[26]以邻接法构建NJ系统发育树,使用软件RAxML[27]构建ML系统发育树,最适模型是由MEGA11软件中的Find Best DNA/Protein Models程序对相关序列进行测试,在Model Finder的Akaike信息准则(AIC)下[28],选择最佳拟合模型GTR+I+G进行BI和ML分析,通过使用1 000 次重复的引导分析来估计系统发育树的稳健性[29]。利用Mrbayes 3.1.2[30]软件进行贝叶斯分析,运行1 000 000代,每100代取样,去除前25%样本树,使用软件Fig Tree version 1.4.2 [31]和Adobe Illustrator CS5对系统发育树进行编辑处理。BI、ML和NJ树均以粘菌藻(Gloeobacter violaceus PCC7421)作为外类群。
1.5 16S-23S rRNA ITS二级结构预测及分析
使用软件RNA structure[32]和RNA fold web server预测藻株的二级结构,分析并对比了其D1-D1ˊ、Box-B以及V3螺旋结构,进一步确定藻株类别。所有藻株的结构格式均在Adobe Illustrator CS5软件中进行修改。
2. 结果与分析
2.1 形态学观察
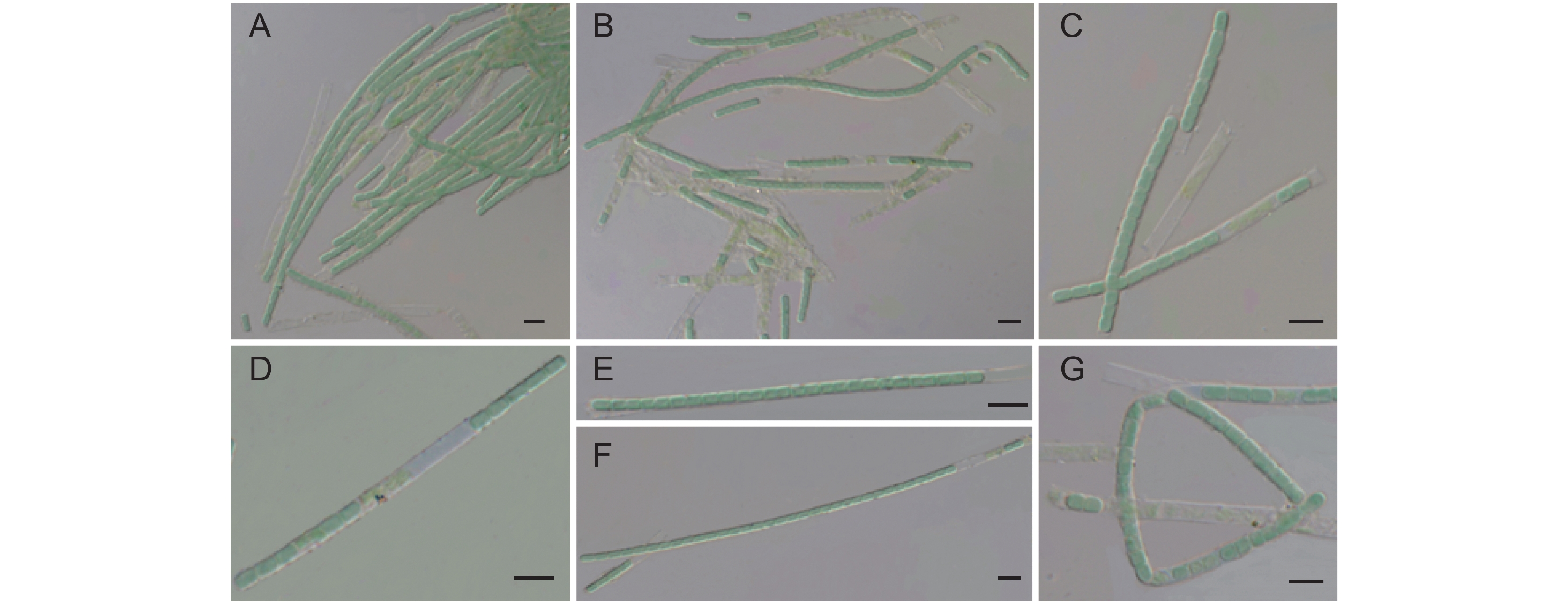
![]() 图 1 光学显微镜下SXACC0050的形态特征A: 群体形态;B:藻丝体;C:有鞘出现;D~F:单根藻丝;G:在横壁和无色鞘处略微收缩。标尺为5 μm。Figure 1. Micrographs of SXACC0050 under light microscopyA: Group morphology; B: Filaments; C: Appearance of sheath; D-F: Single algal filament; G: Slightly constricted at transverse wall and colorless sheath. Scale bars is 5 μm.
图 1 光学显微镜下SXACC0050的形态特征A: 群体形态;B:藻丝体;C:有鞘出现;D~F:单根藻丝;G:在横壁和无色鞘处略微收缩。标尺为5 μm。Figure 1. Micrographs of SXACC0050 under light microscopyA: Group morphology; B: Filaments; C: Appearance of sheath; D-F: Single algal filament; G: Slightly constricted at transverse wall and colorless sheath. Scale bars is 5 μm.Oculatella ucrainica Oxana Vinogradova & Tatiana Mikhailyuk,2017[12]
生境:潮湿的土壤和墙面。
分布:山西省太原师范学院校园和贵州省贵阳市花溪湿地公园。
参考藻株:SXACC0050、SXACC0051。
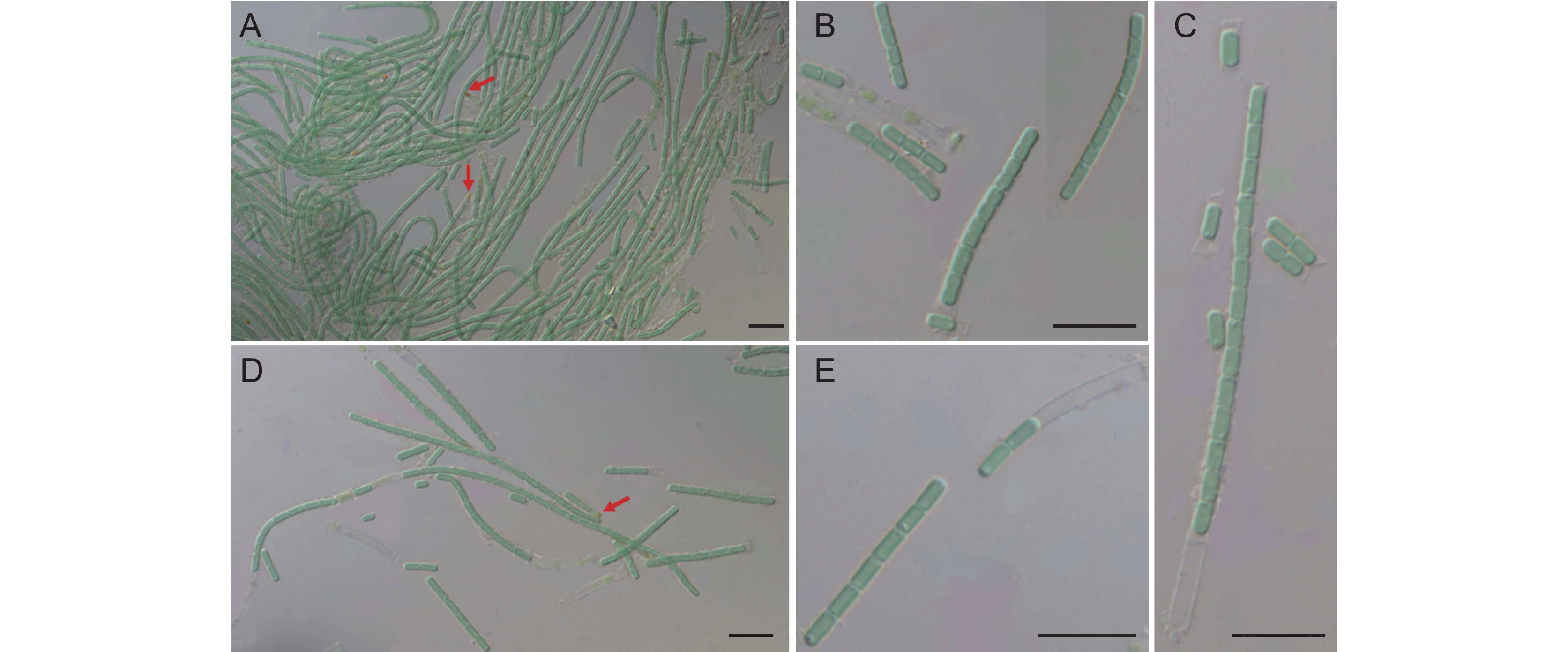
形态描述:藻株蓝绿色,藻丝紧密缠绕在一起,呈典型的簇状(图1:A,图2:A)。丝状体直或弯曲(图1:B~F,图2:B~E),直径约为1~3 μm,由无色鞘包围的细胞组成(图1:C~G,图2 :B~E)。鞘透明且薄,有时为双层,两端偶尔开放(图2:C)。藻丝由细长的细胞组成,细胞圆柱形,宽1~2.5 μm,长1.5~4.5 μm,在横壁和无色鞘处略微收缩(图1:G)。藻丝顶端细胞圆形或圆锥形,含有橘色颗粒(红色箭头表示)。通过藻丝断裂形成2~6个细胞的藻殖段进行繁殖(图1:B,图2:E)。
2.2 系统发育分析
本研究测定了藻株SXACC0050和SXACC0051的16S rRNA 基因序列,长度分别为1 316 bp和1 265 bp,其与细点丝藻属藻株的基因序列相似度分别为98.0%~99.7%和98.3%%~99.9%(表2),与乌克兰细点丝藻的基因序列差异小于0.1%。选择与其相似度较高的序列构建16S rRNA 基因系统发育树,以Gloeobacter violaceus PCC7421为外类群,采用BI、ML和NJ法构建了包含47条相关序列的系统发育树,3种系统发育树具有较为一致的分支位点及拓扑结构,图3为贝叶斯法构建的系统发育树。
表 2 SXACC0050和SXACC0051与细点丝藻属的16S rRNA基因相似性Table 2. Sequence similarity comparison of 16S rRNA gene between SXACC0050, SXACC0051, and Oculatella藻株
Strain1 2 3 4 5 6 7 8 1. SXACC0050 2. SXACC0051 99.7% 3. Oculatella ucrainica strain KZ-5-4-1 99.7% 99.9% 4. Oculatella neakameniensis str. Kovacik 1990/37 98.2% 98.5% 98.5% 5. Oculatella mojaviensis CMT-3BRIN-NPC87 99.5% 99.8% 99.6% 98.4% 6. Oculatella coburnii WJT66-NPBG6A 99.5% 99.8% 99.8% 98.5% 99.8% 7. Oculatella hafneriensis str. HINDAK 1982/12 98.0% 98.3% 98.3% 97.8% 98.3% 98.4% 8. Oculatella dilatativagina UAM 687 99.4% 99.6% 99.5% 98.4% 99.9% 99.5% 98.3% 9. Oculatella kazantipica strain SAG 2567 99.0% 99.2% 99.3% 98.4% 98.9% 99.1% 98.8% 98.9% ![]() 图 3 基于16S rRNA基因序列的BI/ML/NJ系统发育树节点处数字表示在 BI/ML/NJ中大于50%的支持数值,标尺每刻度代表0.07的进化距离。红色字体表示本研究分离的藻株。Figure 3. BI/ML/NJ phylogenetic trees based on 16S rRNA gene sequencesNumbers at each node represent over 50% supporting values in BI/ML/NJ phylogenetic tree, scale bar represents evolution distance of 0.07 per scale. Strains in this study are indicated in red.
图 3 基于16S rRNA基因序列的BI/ML/NJ系统发育树节点处数字表示在 BI/ML/NJ中大于50%的支持数值,标尺每刻度代表0.07的进化距离。红色字体表示本研究分离的藻株。Figure 3. BI/ML/NJ phylogenetic trees based on 16S rRNA gene sequencesNumbers at each node represent over 50% supporting values in BI/ML/NJ phylogenetic tree, scale bar represents evolution distance of 0.07 per scale. Strains in this study are indicated in red.由图3可知,藻株SXACC0050和SXACC0051位于细点丝藻属的进化枝中,且与乌克兰细点丝藻聚为一支(红色字体),BI/ML/NJ的后验概率和自展值分别为0.73/81/59和0.72/82/52,与细点丝藻属中的其他物种分离。结果表明,藻株SXACC0050和SXACC0051大概率属于乌克兰细点丝藻。
2.3 二级结构分析
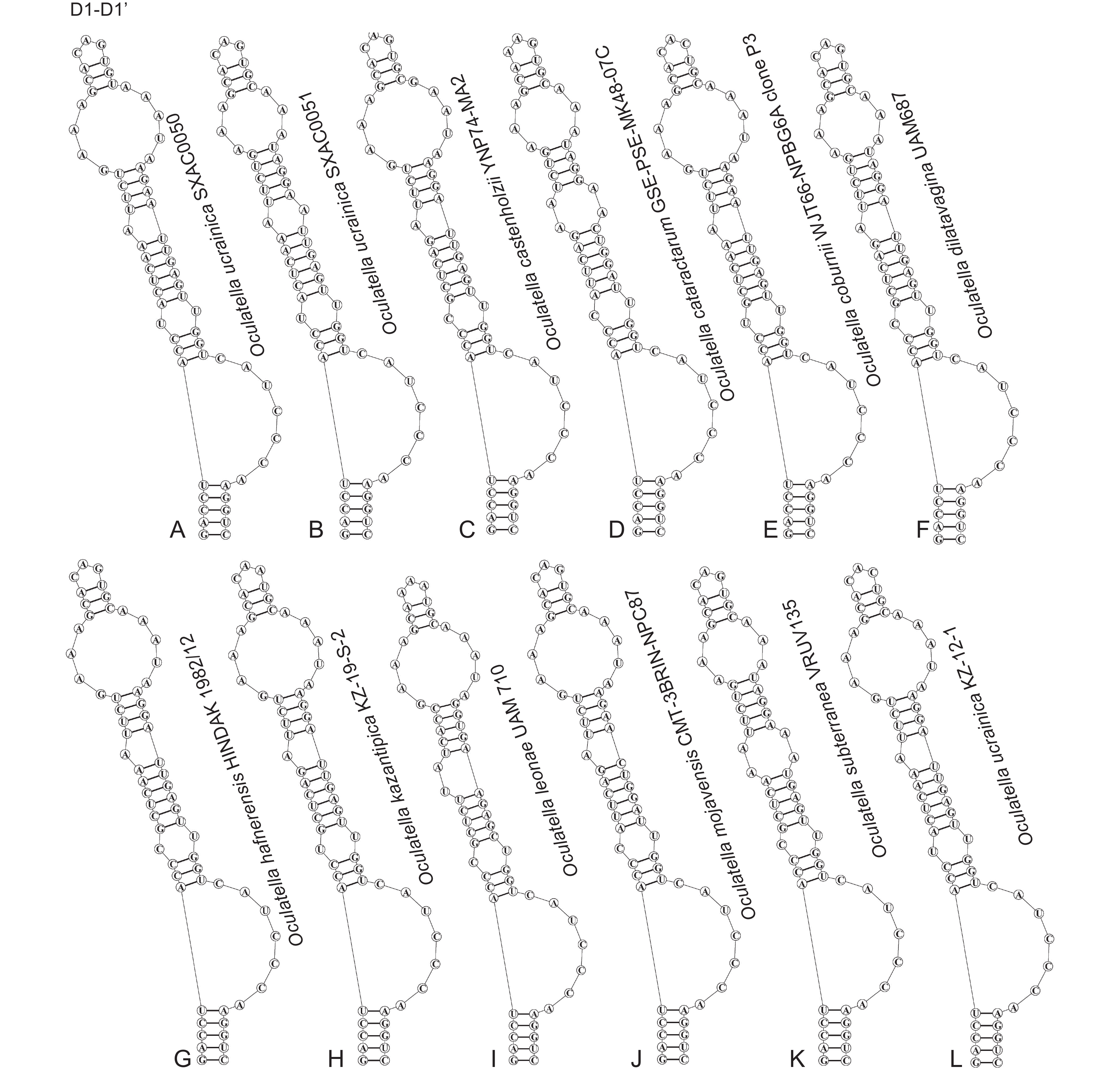
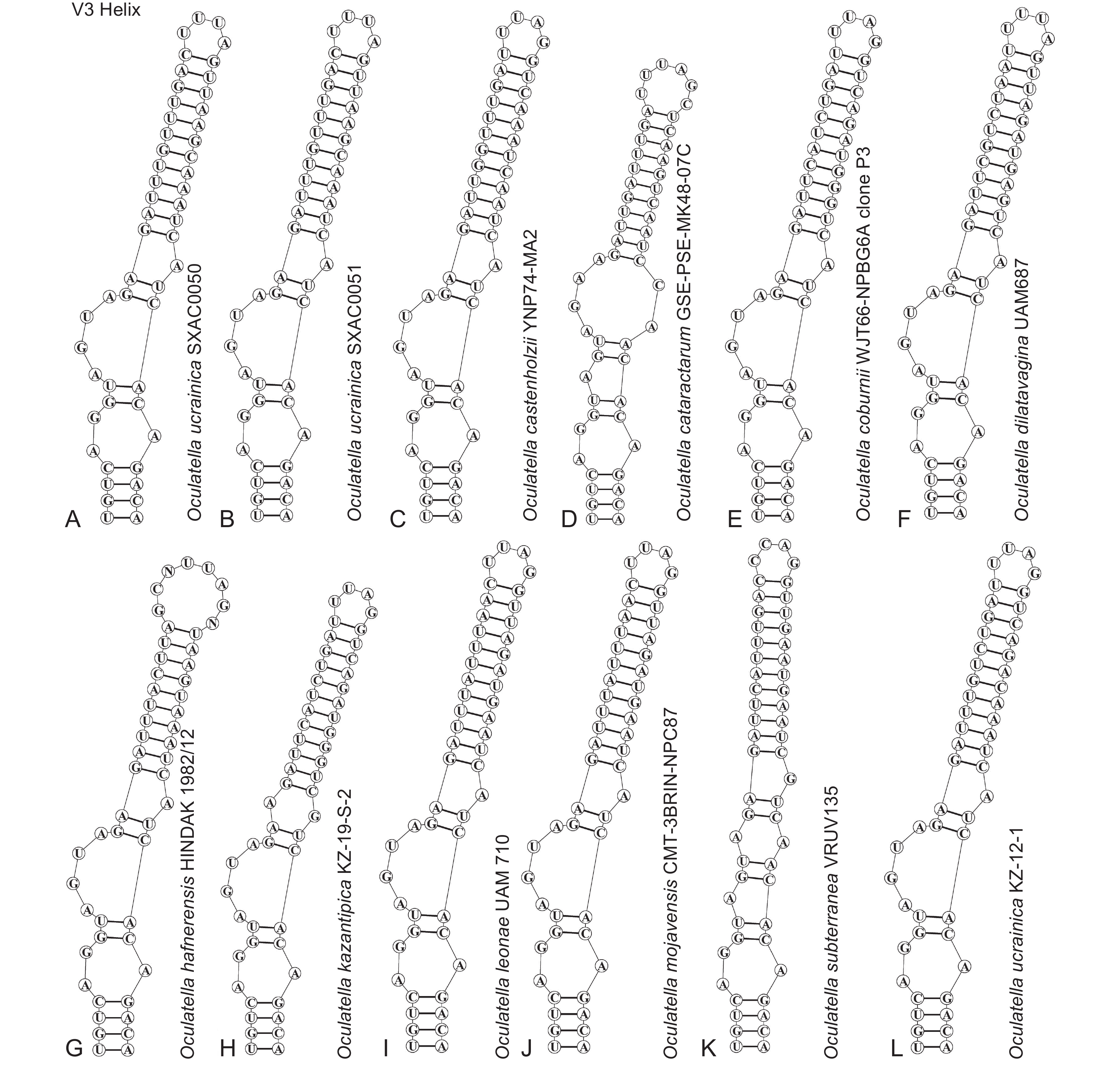
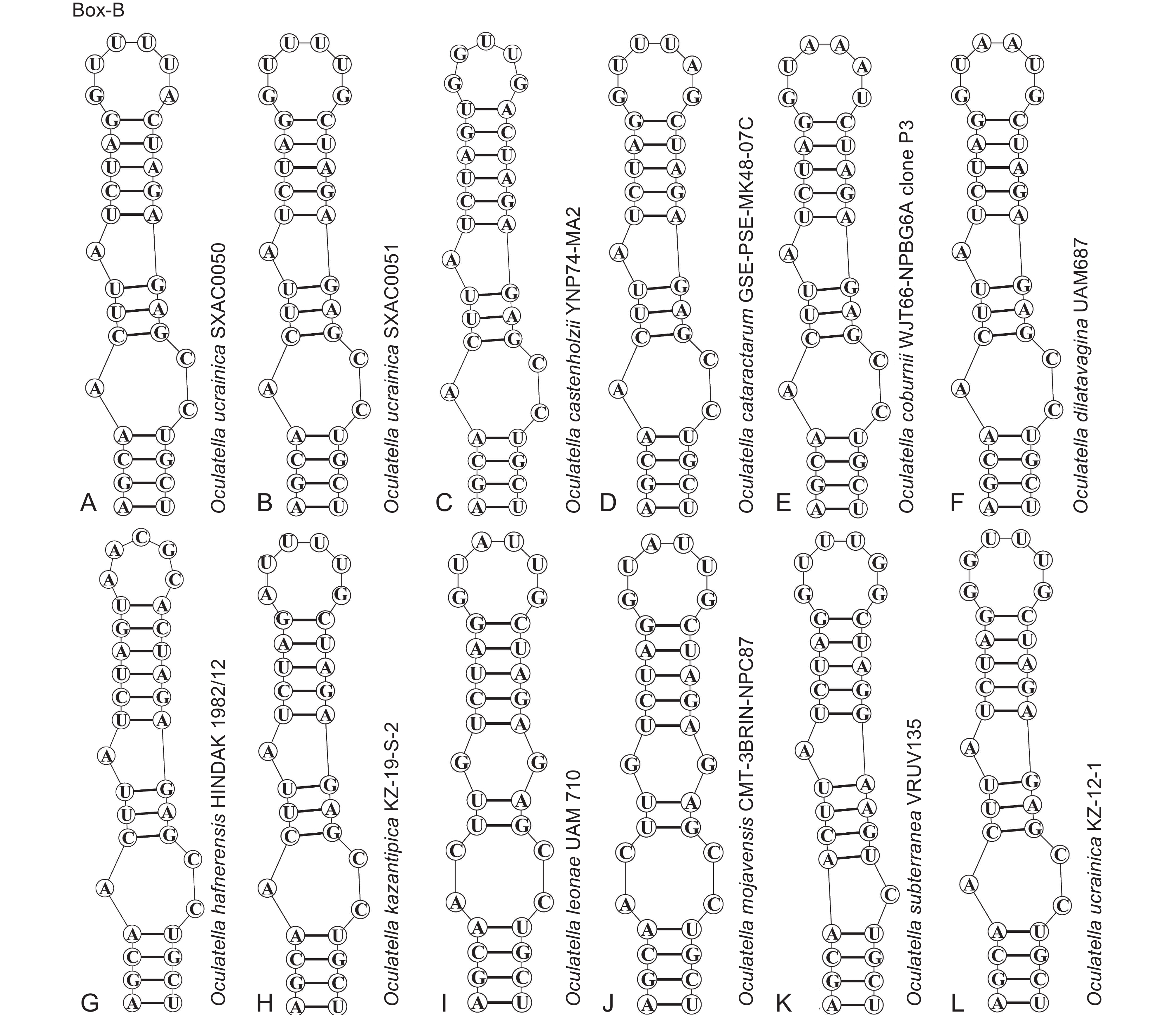
通过测序,得到了SXACC0050(622 bp)和SXACC0051 (622 bp)藻株的ITS序列,选择D1-D1′、Box-B和V3螺旋3个保守区域进行分析。藻株SXACC0050和SXACC0051的二级结构与细点丝藻结构相似,尤其是与乌克兰细点丝藻的二级结构差异很小(图4~图6)。藻株SXACC0050和SXACC0051与乌克兰细点丝藻的D1-D1′螺旋结构相似,其D1-D1′螺旋均为64个核苷酸,共有5个茎环结构,基部茎由1个含5 bp的螺旋构成,基部3′侧环由7个未配对的核苷酸(5′-CAUCCCA-3′)组成,之后是1个1∶1的双侧凸起(U∶U),末端环由3 bp碱基(5′-CAG-3′)构成。但与乌克兰细点丝藻相比,藻株SXACC0050的第33位由胞嘧啶变为尿嘧啶,第41位由鸟嘌呤变为腺嘌呤(图4:A),藻株SXACC0051的茎环结构发生改变(图4:B)。SXACC0050和SXACC0051两个藻株与乌克兰细点丝藻的Box-B结构略有差异。Box-B螺旋由34个核苷酸组成,共有3个茎环结构,基部茎均由1个含4 bp碱基的螺旋构成,末端环由6 bp碱基组成。但与乌克兰细点丝藻相比,两个藻株的末端环碱基有所不同(图5:A、B)。乌克兰细点丝藻的V3螺旋包含52个核苷酸,共有4个茎环结构,基部茎由1个含4 bp碱基的螺旋构成,末端环由4 bp碱基构成。与乌克兰细点丝藻相比,SXACC0050和SXACC0051中该结构的碱基发生变化,第43位由鸟嘌呤变为腺嘌呤,末端环碱基也不同(图6)。分析结果支持两个藻株均属于乌克兰细点丝藻(图4~图6)。
3. 讨论
在现代蓝藻分类学中,理想的蓝藻属和种应该是单系的[9, 33]。近年来,蓝藻的分类依据在不断变化[8, 34, 35],从只通过形态特征描述到加入分子数据,再到结合生态环境等多相方法,蓝藻分类系统得到了大量的修正和重建[36],蓝藻属和种也在向单系方向推进。细点丝藻科在2018年由Mai等[37]从细鞘丝藻科分离出来,该科当时属于聚球藻目,同时他们还将假鱼腥藻目[38]包含的简单丝状蓝藻合并到聚球藻目中,系统发育分析表明,细点丝藻科中混有细鞘丝藻(Leptolyngbya)和假鱼腥藻属(Pseudanabaena)[38],所以该科需要修订和重建。细点丝藻科的模式属为细点丝藻属,根据藻类数据库 Algaebase统计,目前细点丝藻属有14个物种被描述,并被有效接受。该属的大部分物种为陆生种类,其主要形态特征是在丝状体顶端含有特殊的橙色颗粒[11],呈现斑点状,这种颗粒可能是一种对光敏感的物质。细点丝藻属分布的生态环境也非常特殊,其模式种(O. subterranea Zammit, Billi & Albertano)是从马耳他的地下环境中发现的,该地湿度很高,光照强度很低,是较为特殊的极端环境。细点丝藻的16S rRNA基因相似度较高,容易同相邻或相近的属种混淆,需要通过ITS二级结构进行区分。
本研究通过构建系统发育树,发现分别从山西省和贵州省分离到的两株丝状蓝藻SXACC0051和SXACC0050分布在细点丝藻进化分支中,且与乌克兰细点丝藻聚为一支,具有较高的支持值。二者与细点丝藻属几个藻种的序列相似度大于98.7%,高于2014年Kim等[13]提出的区分两个物种的阈值。这两个藻株与细点丝藻属的形态特征相似,均在丝状体顶端出现橘色颗粒,不分枝,均通过丝状体断裂形成2~6个细胞的藻殖段进行繁殖[11]。从细胞大小来看,SXACC0050和SXACC0051的藻丝均比Vinogradova等[12]在2017年描述的乌克兰细点丝藻更宽,这可能是其来源于不同的生态环境造成的。通过ITS二级结构分析,发现两个藻株的D1-D1′、Box-B和V3螺旋这3个保守区域均与乌克兰细点丝藻具有较高的相似性,进一步证实了形态学的鉴定结果。基于此,我们认为,SXACC0050和SXACC0051为中国新记录属细点丝藻属的一个新记录种。乌克兰细点丝藻在我国属于首次报道,也增加了我国蓝藻的物种多样性。目前,有关细点丝藻属的研究报道较少,主要分布在乌克兰[12]、马耳他[11]、意大利、西班牙[39]等国家,大量采集标本和运用多相方法进行鉴定可能有助于解决这个问题。未来我们也应该加强此类研究,以期为我国藻类开发和利用提供科学依据。
-
图 1 光学显微镜下SXACC0050的形态特征
A: 群体形态;B:藻丝体;C:有鞘出现;D~F:单根藻丝;G:在横壁和无色鞘处略微收缩。标尺为5 μm。
Figure 1. Micrographs of SXACC0050 under light microscopy
A: Group morphology; B: Filaments; C: Appearance of sheath; D-F: Single algal filament; G: Slightly constricted at transverse wall and colorless sheath. Scale bars is 5 μm.
图 3 基于16S rRNA基因序列的BI/ML/NJ系统发育树
节点处数字表示在 BI/ML/NJ中大于50%的支持数值,标尺每刻度代表0.07的进化距离。红色字体表示本研究分离的藻株。
Figure 3. BI/ML/NJ phylogenetic trees based on 16S rRNA gene sequences
Numbers at each node represent over 50% supporting values in BI/ML/NJ phylogenetic tree, scale bar represents evolution distance of 0.07 per scale. Strains in this study are indicated in red.
表 1 采集地的详细信息
Table 1 Details of the collection site
藻株
Strain采集地
Collection site经纬度
Latitude and longitude海拔
Elevation / m温度
Temperature / ℃pH值
pH value生境
HabitatSXACC0051 山西省太原师范学院校园 26°26ˊ07″N,
106°39ˊ57″E808 16.3 7.59 潮湿土壤 SXACC0050 贵州省贵阳市花溪湿地公园 26°26ˊ07″N,
106°39ˊ57″E1 140 20.5 7.86 湿润墙面 表 2 SXACC0050和SXACC0051与细点丝藻属的16S rRNA基因相似性
Table 2 Sequence similarity comparison of 16S rRNA gene between SXACC0050, SXACC0051, and Oculatella
藻株
Strain1 2 3 4 5 6 7 8 1. SXACC0050 2. SXACC0051 99.7% 3. Oculatella ucrainica strain KZ-5-4-1 99.7% 99.9% 4. Oculatella neakameniensis str. Kovacik 1990/37 98.2% 98.5% 98.5% 5. Oculatella mojaviensis CMT-3BRIN-NPC87 99.5% 99.8% 99.6% 98.4% 6. Oculatella coburnii WJT66-NPBG6A 99.5% 99.8% 99.8% 98.5% 99.8% 7. Oculatella hafneriensis str. HINDAK 1982/12 98.0% 98.3% 98.3% 97.8% 98.3% 98.4% 8. Oculatella dilatativagina UAM 687 99.4% 99.6% 99.5% 98.4% 99.9% 99.5% 98.3% 9. Oculatella kazantipica strain SAG 2567 99.0% 99.2% 99.3% 98.4% 98.9% 99.1% 98.8% 98.9% -
[1] Fritsch FE. Kryptogamen-flora von deutschland,österreich und der schweiz[J]. Nature,1932,130(3278):292−293. doi: 10.1038/130292a0
[2] Schirrmeister BE,Antonelli A,Bagheri HC. The origin of multicellularity in cyanobacteria[J]. BMC Evol Biol,2011,11(1):45. doi: 10.1186/1471-2148-11-45
[3] Herrero A,Stavans J,Flores E. The multicellular nature of filamentous heterocyst-forming cyanobacteria[J]. FEMS Microbiol Rev,2016,40(6):831−854. doi: 10.1093/femsre/fuw029
[4] Komárek J. Problem of the taxonomic category "species" in cyanobacteria[J]. Algol Stud,2003,109(1):281−297.
[5] 谢艳,张荣臻,张葵东,吕兴菊,张和,等. 中国水华蓝藻新记录属——泽鞘丝藻属(Limnoraphis)[J]. 河南师范大学学报(自然科学版),2023,51(4):125−131. Xie Y,Zhang RZ,Zhang KD,Lü XJ,Zhang H, et al. Limnoraphis (Oscillatoriales,Cyanobacteria),a bloom-forming genus newly recorded from China[J]. Journal of Henan Normal University (Natural Science Edition),2023,51(4):125−131.
[6] Komárek J. A polyphasic approach for the taxonomy of cyanobacteria:principles and applications[J]. Eur J Phycol,2016,51(3):34−353.
[7] 魏爱丽,杨谢,王捷,巩超彦,王清华,李艳晖. 念珠藻类(蓝藻)psbA基因的进化分析[J]. 河南师范大学学报(自然科学版),2023,51(1):114−122. Wei AL,Yang X,Wang J,Gong CY,Wang QH,Li YH. Evolutionary analysis on psbA gene of Nostoc-like (Cyanobacteria)[J]. Journal of Henan Normal University (Natural Science Edition),2023,51(1):114−122.
[8] Geitler L,Ruttner F. Die cyanophyceen der Deutschen limnologischen Sunda-expedition,ihre morphologie,systematik und ökologie. zweiter teil[J]. Arch Hydrobiol Trop Binnengewä sser,1936,14:371−483.
[9] Komárek J,Kaštovský J,Mareš J,Johansen JR. Taxonomic classification of cyanoprokaryotes (cyanobacterial genera) 2014,using a polyphasic approach[J]. Preslia,2014,86(4):295−335.
[10] Strunecký O,Ivanova AP,Mareš J. An updated classification of cyanobacterial orders and families based on phylogenomic and polyphasic analysis[J]. J Phycol,2023,59(1):12−51. doi: 10.1111/jpy.13304
[11] Zammit G,Billi D,Albertano P. The subaerophytic cyanobacterium Oculatella subterranea (Oscillatoriales,Cyanophyceae)gen. et sp. nov.:a cytomorphological and molecular description[J]. Eur J Phycol,2012,47(4):341−354. doi: 10.1080/09670262.2012.717106
[12] Vinogradova O,Mikhailyuk T,Glaser K,Holzinger A,Karsten U. New species of Oculatella (Synechococcales,Cyanobacteria) from terrestrial habitats of Ukraine[J]. Ukr Bot J,2017,74(6):509−520. doi: 10.15407/ukrbotj74.06.509
[13] Kim M,Oh HS,Park SC,Chun J. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. Int J Syst Evol Microbiol,2014,64(Pt 2):346−351.
[14] Rippka R. Recognition and identification of cyanobacteria[J]. Methods Enzymol,1988,167:28−67.
[15] Pachiappan P,Prasath BB,Perumal S,Ananth S,Devi AS, et al. Isolation and culture of microalgae[M]//Perumal S,Thirunavukkarasu AR,Pachiappan P,eds. Advances in Marine and Brackishwater Aquaculture. New Delhi:Springer,2015:1−15.
[16] Van Ranst G,Baert P,Fernandes AC,de Grave J. Technical note:Nikon-TRACK Flow,a new versatile microscope system for fission track analysis[J]. Geochronology,2020,2(1):93−99. doi: 10.5194/gchron-2-93-2020
[17] Fiore MF,Moon DH,Tsai SM,Lee H,Trevors JT. Miniprep DNA isolation from unicellular and filamentous cyanobacteria[J]. J Microbiol Methods,2000,39(2):159−169. doi: 10.1016/S0167-7012(99)00110-4
[18] Neilan BA,Jacobs D,Goodman AE. Genetic diversity and phylogeny of toxic cyanobacteria determined by DNA polymorphisms within the phycocyanin locus[J]. Appl Environ Microbiol,1995,61:3875−3883. doi: 10.1128/aem.61.11.3875-3883.1995
[19] Edwards U,Rogall T,Blöcker H,Emde M,Böttger EC. Isolation and direct complete nucleotide determination of entire genes. Characterization of a gene coding for 16S ribosomal RNA[J]. Nucleic Acids Res,1989,17(19):7843−7853. doi: 10.1093/nar/17.19.7843
[20] Lin S,Wu ZX,Yu GL,Zhu ML,Yu BS,Li RH. Genetic diversity and molecular phylogeny of Planktothrix (Oscillatoriales,cyanobacteria) strains from China[J]. Harmful Algae,2010,9(1):87−97. doi: 10.1016/j.hal.2009.08.004
[21] Turner S,Pryer KM,Miao VPW,Palmer JD. Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis[J]. J Eukaryotic Microbiol,1999,46(4):327−338. doi: 10.1111/j.1550-7408.1999.tb04612.x
[22] Iteman I,Rippka R,de Marsac NT,Herdman M. Comparison of conserved structural and regulatory domains within divergent 16S rRNA-23S rRNA spacer sequences of cyanobacteria[J]. Microbiology,2000,146(6):1275−1286. doi: 10.1099/00221287-146-6-1275
[23] 张军毅,孙蓓丽,朱冰川,石浚哲,周克茹,等. 基于分子标记的藻类鉴定研究进展[J]. 湖泊科学,2021,33(6):1607−1625. doi: 10.18307/2021.0601 Zhang JY,Sun BL,Zhu BC,Shi JZ,Zhou KR,et al. Research progress in the taxonomic identification of algae on the basis of molecular markers[J]. Journal of Lake Sciences,2021,33(6):1607−1625. doi: 10.18307/2021.0601
[24] 肖喆,王捷,石瑛,王清华,李艳晖,谭梅娟. 晋阳湖丝状蓝藻的形态学分析与分子鉴定[J]. 西北植物学报,2022,42(3):427−434. Xiao Z,Wang J,Shi Y,Wang QH,Li YH,Tan MJ. Morphological analysis and molecular identification of filamentous cyanobacteria from Jinyang Lake[J]. Acta Botanica Boreali-Occidentalia Sinica,2022,42(3):427−434.
[25] 彭焕文,王伟. 基于分子数据的系统发生树构建[J]. 植物学报,2023,58(2):261−273. Peng HW,Wang W. Phylogenetic tree reconstruction based on molecular data[J]. Chinese Bulletin of Botany,2023,58(2):261−273.
[26] Tamura K,Stecher G,Kumar S. MEGA11:molecular evolutionary genetics analysis version 11[J]. Mol Biol Evol,2021,38(7):3022−3027. doi: 10.1093/molbev/msab120
[27] Stamatakis A. RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies[J]. Bioinformatics,2014,30(9):1312−1313. doi: 10.1093/bioinformatics/btu033
[28] Kalyaanamoorthy S,Minh BQ,Wong TKF,von Haeseler A,Jermiin LS. ModelFinder:fast model selection for accurate phylogenetic estimates[J]. Nat Methods,2017,14(6):587−589. doi: 10.1038/nmeth.4285
[29] Hoang DT,Chernomor O,von Haeseler A,Minh BQ,Vinh LS. UFBoot2:improving the ultrafast bootstrap approximation[J]. Mol Biol Evol,2018,35(2):518−522. doi: 10.1093/molbev/msx281
[30] Ronquist F,Huelsenbeck JP. MrBayes 3:Bayesian phylogenetic inference under mixed models[J]. Bioinformatics,2003,19(12):1572−1574. doi: 10.1093/bioinformatics/btg180
[31] Pinto AR,Machado Cunha I,Rebelo Gomes E. Fig tree-induced phytophotodermatitis:a case report on the perils of a hobby[J]. Cureus,2023,15(7):e41888.
[32] Reuter JS,Mathews DH. RNAstructure:software for RNA secondary structure prediction and analysis[J]. BMC Bioinformatics,2010,11:129. doi: 10.1186/1471-2105-11-129
[33] Komárek J. Several problems of the polyphasic approach in the modern cyanobacterial system[J]. Hydrobiologia,2018,811(1):7−17. doi: 10.1007/s10750-017-3379-9
[34] 胡鸿钧,魏印心. 中国淡水藻类:系统、分类及生态[M]. 北京:科学出版社,2006:23−27. [35] 黄冰心,丁兰平. 中国海洋蓝藻门新分类系统[J]. 广西科学,2014,21(6):580−586. Huang BX,Ding LP. The catalogue of marine cyanobacteria of China[J]. Guangxi Sciences,2014,21(6):580−586.
[36] Cai FF,Yu GL,Li RH. Description of two new species of Pseudoaliinostoc (Nostocales,Cyanobacteria) from China based on the polyphasic approach[J]. J Oceanol Limnol,2022,40(3):1233−1244. doi: 10.1007/s00343-021-1111-0
[37] Mai T,Johansen JR,Pietrasiak N,Bohunická M,Martin MP. Revision of the Synechococcales(Cyanobacteria) through recognition of four families including Oculatellaceae fam. nov. and Trichocoleaceae fam. nov. and six new genera containing 14 species[J]. Phytotaxa,2018,365(1):1−59. doi: 10.11646/phytotaxa.365.1.1
[38] 李仁辉. 建议将Pseudanabaena译成“假鱼腥藻属”而不是“伪鱼腥藻属”[J]. 湖泊科学,2022,34(4):1392. [39] Osorio-Santos K,Pietrasiak N,Bohunická M,Miscoe LH,Kováčik L,et al. Seven new species of Oculatella (Pseudanabaenales,Cyanobacteria):taxonomically recognizing cryptic diversification[J]. Eur J Phycol,2014,49(4):450−470. doi: 10.1080/09670262.2014.976843



 下载:
下载: